オルトコロナウイルス亜科
RNAウイルスのひとつ
オルトコロナウイルス亜科(オルトコロナウイルスあか、Orthocoronavirinae / コロナウイルス〈Coronavirus〉)は、ゲノムとしてリボ核酸 (RNA) をもつ一本鎖プラス鎖RNAウイルスで、哺乳類や鳥類に病気を引き起こすウイルスのグループの1つ[1]。ニドウイルス目コロナウイルス科に属する[2][3]。
| オルトコロナウイルス亜科 | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
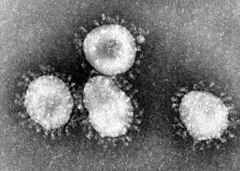 伝染性気管支炎ウイルスの電子顕微鏡写真 | |||||||||||||||||||||
| 分類 | |||||||||||||||||||||
| |||||||||||||||||||||
| 属 | |||||||||||||||||||||
概略
編集
単にコロナウイルスと言った場合、コロナウイルス科、あるいはこのオルトコロナウイルス亜科を指すとされている。コロナウイルス科は、アルファからデルタまでのコロナウイルス以外にも、レトウイルス亜科を含んでいるため、実際の分類範囲としては、コロナウイルス=オルトコロナウイルス亜科となっている。
オルトコロナウイルスは、ウイルス粒子表面のエンベロープ︵膜構造︶に、花弁状の長いスパイク蛋白の突起︵S蛋白、約 20 nm︶を持ち、外観がコロナ︵太陽の光冠︶に似ている[1]。らせん対称性のヌクレオカプシドをもつエンベロープウイルスである。多形性で、大きさは直径80-220nm程度である[1]。ゲノムサイズは約26〜32キロベース (kb) で、既知のRNAウイルスでは最大級である[4]。
症状は生物の種類によって異なり、鶏の場合は上気道疾患を引き起こし、牛や豚の場合は下痢を引き起こす。
ヒトでは、風邪を含む呼吸器感染症を引き起こす。SARSコロナウイルス (SARS-CoV)、MERSコロナウイルス (MERS-CoV) およびSARSコロナウイルス2 (SARS-CoV-2) のようなタイプのウイルスでは、致死性を持つ。
名前
編集
コロナウイルス (Coronavirus) の名称は、ウイルスを電子顕微鏡で観察したときに見られる、ビリオン︵感染性を有するウイルス粒子︶の特徴的な外観に由来する。ビリオンは大きな球状の表面突起の縁をもち、太陽コロナを思わせる像をつくる。
また﹁コロナ: Corona﹂は、ラテン語: corona︵コロナ︶=光冠︵丸い光の輪︶を意味するギリシア語: κορώνη︵korṓnē コロネ︶に由来している[5][6]。
亜科名の﹁オルト: Ortho-﹂は、ギリシア語で﹁歪みのない﹂を意味する ορθός︵オルソス︶が由来である。
歴史
編集
このグループで最初に発見されたのは、1931年に報告されたニワトリの伝染性気管支炎ウイルスである[7]。1940年代にはマウス肝炎ウイルス、豚伝染性胃腸炎ウイルスも報告されている[注1]。
ヒトの病原体としては、1960年に風邪をひいたヒトから、ヒトコロナウイルスB814が発見された。この株は培養が難しかったため後に失われた。1960年代には風邪をひいたヒト患者の鼻腔からヒトコロナウイルス229EおよびヒトコロナウイルスOC43の2つのウイルスが発見された[8]。当初はこれらのウイルスの関係は定かではなかった。ヒトコロナウイルスも一時はヒト呼吸器ウイルス︵Human respiratory virus︶とも呼ばれていた。
1960年代になると、電子顕微鏡写真で構造の類似が指摘され[9]、コロナウイルスと呼ぶ人が多くなった[10]。1971年にはこれらがコロナウイルス属としてまとめられた。
2009年には、分子系統解析の進展により分類の整理が進んだ。コロナウイルス属は解体され、新たにアルファからデルタコロナウイルスが設置された。また、コロナウイルスに近縁なウイルスとしてトロウイルス亜科︵後にトロウイルス科として独立︶が発見され、アルファからデルタまでのコロナウイルスのグループとしてコロナウイルス亜科が設定された。
2018年にはまた新たな動きがあり、トロウイルス亜科がトロウイルス科として独立した一方で、コロナウイルス科の新たなグループとしてレトウイルス亜科が設定された。この時コロナウイルス亜科はオルトコロナウイルス亜科に改名され、現在に至っている。
コロナウイルス及び近縁系統の分類の変遷
編集1971年
| 上位分類 | 下位分類 | |
|---|---|---|
| (設定無し) | コロナウイルス属 | 伝染性気管支炎ウイルス |
| マウス肝炎ウイルス | ||
| ヒト呼吸器ウイルス 等 |
2009年
| 上位分類 | 下位分類 | |
|---|---|---|
| コロナウイルス科 | コロナウイルス亜科 | アルファコロナウイルス属 |
| ベータコロナウイルス属 | ||
| デルタコロナウイルス属 | ||
| ガンマコロナウイルス属 | ||
| トロウイルス亜科 | トロウイルス属 |
2018年
| 上位分類 | 下位分類 | |
|---|---|---|
| コロナウイルス科 | オルトコロナウイルス亜科 | アルファコロナウイルス属 |
| ベータコロナウイルス属 | ||
| デルタコロナウイルス属 | ||
| ガンマコロナウイルス属 | ||
| レトウイルス亜科 | アルファレトウイルス属 |
分類
編集ウイルスの分類においてオルトコロナウイルス亜科は、レトウイルス亜科と共にコロナウイルス科に含まれる[11]。オルトコロナウイルスには4属45種を含む。
オルトコロナウイルス亜科
編集アルファコロナウイルス属
編集- アルファコロナウイルス属(Alphacoronavirus) タイプ種:アルファコロナウイルス1
- 種:Bat coronavirus CDPHE15、Bat coronavirus HKU10、Rhinolophus ferrumequinum alphacoronavirus HuB-2013、ヒトコロナウイルス229E、Lucheng Rn rat coronavirus、Mink coronavirus 1、Miniopterus bat coronavirus 1、Miniopterus bat coronavirus HKU8、Myotis ricketti alphacoronavirus Sax-2011、Nyctalus velutinus alphacoronavirus SC-2013、Pipistrellus kuhlii coronavirus 3398、豚流行性下痢ウイルス(Porcine epidemic diarrhea virus)、Scotophilus bat coronavirus 512、Rhinolophus bat coronavirus HKU2、Human coronavirus NL63、NL63-related bat coronavirus strain BtKYNL63-9b、Sorex araneus coronavirus T14、Suncus murinus coronavirus X74、アルファコロナウイルス1
ベータコロナウイルス属
編集- ベータコロナウイルス属(Betacoronavirus) タイプ種:マウスコロナウイルス
- 種:ベータコロナウイルス1、China Rattus coronavirus HKU24、ヒトコロナウイルスHKU1、マウスコロナウイルス、Myodes coronavirus 2JL14、Bat Hp-betacoronavirus Zhejiang2013、Hedgehog coronavirus 1、MERSコロナウイルス、Pipistrellus bat coronavirus HKU5、Tylonycteris bat coronavirus HKU4、Eidolon bat coronavirus C704、Rousettus bat coronavirus GCCDC1、Rousettus bat coronavirus HKU9、SARS関連コロナウイルス
ガンマコロナウイルス属
編集- ガンマコロナウイルス属(Gammacoronavirus) タイプ種:鶏伝染性気管支炎ウイルス
- 種:Goose coronavirus CB17、Beluga whale coronavirus SW1、鶏伝染性気管支炎ウイルス、Avian coronavirus 9203、Duck coronavirus 2714
デルタコロナウイルス属
編集- デルタコロナウイルス属(Deltacoronavirus) タイプ種:ヒヨドリコロナウイルスHKU11
- 種:Wigeon coronavirus HKU20、ヒヨドリコロナウイルスHKU11、Common moorhen coronavirus HKU21、Coronavirus HKU15、Munia coronavirus HKU13、White-eye coronavirus HKU16、Night heron coronavirus HKU19
構造
編集オルトコロナウイルス亜科に属するウイルスは粒子状であり、その表面は細胞一般と同じ脂質二重膜である。電子顕微鏡で撮影すると表面にスパイクタンパク質が多数生えている様子が王冠のように見える。この脂質二重膜はエンベロープと呼ばれ、スパイクと共に感染先となる宿主細胞を認識する機能を持つヘマグルチニンタンパクが埋め込まれている(特にベータコロナウイルスサブグループAのメンバー)[2]。エンベロープの内部にはウイルスのゲノムがある。ゲノムはタンパク質に包まれた一本のRNAであり、このゲノムRNAがタンパク質に包まれた状態をヌクレオカプシドと呼ぶ。
ゲノム
編集
オルトコロナウイルス亜科︵及び他の殆どのニドウイルス目︶の特徴の一つは、そのゲノムがDNAではなくRNAであることである。そのゲノムRNAは宿主細胞の中でそのまま伝令RNA︵messenger RNA、mRNA、タンパク質に翻訳され得る塩基配列情報と構造を持ったRNA︶として機能する配列構造になっている[5]。mRNAは、ゲノムRNAの ‘3 側から ‘5 側に違う長さで伸張する数本のmRNAから構成され、各mRNAの ‘5 末端はゲノムRNAの 5’ 末端にあるリーダー配列を持つ[5]。
ウイルスタンパクの翻訳は一般に各mRNAの ‘5 末端に存在するオープンリーディングフレーム (ORF) からのみ翻訳される[5]。ゲノムRNA (mRNA-1) の ‘5 末端約 20kb には2つのORF︵1a と 1b で 802kDa をコードする︶からなり、このORF間にはシュードノット (pseudoknot, Pn) と呼ばれる核酸の三次構造を持つ[5]。1a タンパクだけで翻訳が終止する場合と、Pnにより 1a + 1b 融合タンパクが合成されるケースがある。1a + 1b タンパクは16個の調節タンパクに解裂され、プロテアーゼ︵ペプチド結合加水分解酵素︶、RNAポリメラーゼとして働く[5]。
タンパク質
編集増殖過程
編集
オルトコロナウイルスは動物細胞に感染することによって増殖する。その過程は、おおむね、感染 (1、2)、複製 (3 - 5)、放出 (6) の3段階からなる。
(一)ウイルスエンベロープ表面に露出しているスパイクタンパク質Sおよび、種によってはヘマグルチニンタンパク質HEが標的細胞表面の分子を認識し、結合する。
(二)TMPRSS2などの宿主プロテアーゼによって、スパイクタンパク質が切断、活性化を受ける。
(三)ウイルスエンベロープと標的細胞の細胞膜が直接融合、あるいはエンドサイトーシスによってウイルスが細胞内に取り込まれる。直接融合の場合、ウイルスゲノムが細胞内に直接導入されるが、エンドサイトーシスによって取り込まれる場合は、一旦ウイルスが含まれたエンドソームが細胞内に作られ、そこでエンドソーム膜とウイルスが融合することによってウイルスゲノムが導入される。エンドソーム内は通常、プロトンポンプでその内部のpHが下げられるが、これはリソソームへの移送とともにウイルスによって阻害される。
(四)コロナウイルスはプラス鎖の一本鎖RNAをゲノムとして持つため、標的細胞の細胞質でそのままmRNAとして機能する。標的細胞のリボソームに結合して、RNA合成酵素を含むウイルスのタンパク質が作られる。ウイルスのRNA合成酵素はウイルスのゲノム配列以外は複製せず、ウイルスのゲノムRNAだけを鋳型にして、マイナス鎖のRNAとして複製する。これにより、ウイルス自身のRNA塩基配列だけを複製していく準備が整う。
(五)マイナス鎖ウイルスゲノムRNAから遺伝子ごとにプラス鎖RNAが合成され、それらが標的細胞のリボソームに結合し、それぞれからウイルスタンパク質が作られる。またマイナス鎖ゲノムから、ウイルスを構成するプラス鎖ゲノムが複製される。そのゲノムは細胞に侵入してきたRNA配列と同じである。
(六)作られたウイルスタンパク質Nがプラス鎖ゲノムRNAに結合してヌクレオカプシドを作り、標的細胞の小胞体 (ER) に取り込まれる。ウイルス膜タンパク質M、スパイクタンパク質S、ヘマグルチニンHEは標的細胞の小胞体の膜に組み込まれる。この小胞体の膜がそのままウイルスの膜となる。ヌクレオカプシドと小胞体の膜︵エンベロープになる︶からウイルスが作られる。
(七)小胞体からゴルジ体を経由して、エキソサイトーシスによって標的細胞からウイルスが細胞外に放出される。そのとき、ウイルスのスパイクタンパク質はエキソサイトーシスの膜にしがみつくような形で放出される。これにより、ウイルスが複製される。
SARSコロナウイルスの特異的な例では、S上の定義された受容体結合ドメインがウイルスの細胞受容体であるアンジオテンシン変換酵素2 (ACE2) への結合を仲介する[12]。
進化過程
編集
すべてのコロナウイルス︵=オルトコロナウイルス亜科︶の最新の最も近い共通祖先 ︵MRCA︶は、紀元前8000年には存在していたと考えられているが、一部のモデルのMRCAは5500万年以上前に遡ってコウモリとの長期的な共進化を示唆する[13]。 アルファコロナウイルス属のMRCAは紀元前2400年頃、ベータコロナウイルス属紀元前3300年頃、ガンマコロナウイルス属は紀元前2800年頃、デルタコロナウイルス属は紀元前3000年頃と考えられている。飛翔温血脊椎動物であるコウモリと鳥は、コロナウイルスの遺伝子源︵アルファコロナウイルスとベータコロナウイルスはコウモリ 、ガンマコロナウイルスとデルタコロナウイルスは鳥︶にとって、コロナウイルスの進化と普及を促進する宿主として理想的である[14]。
このため多くのヒトコロナウイルスはコウモリに起源をもつ[15]。ヒトコロナウイルスNL63は、紀元1190~1449年の間に、コウモリコロナウイルス (ARCoV 2) と共通の祖先を有していた[16]。ヒトコロナウイルス229Eも、1686~1800年の間に、コウモリコロナウイルス︵GhanaGrp1 Bt CoV︶と共通の祖先を有していた[17]。より最近の例では、1960年以前にアルパカコロナウイルスとヒトコロナウイルス229Eが分岐した[18]。MERSコロナウイルスは、コウモリから中間宿主としてラクダを介してヒトに現れた[19]。MERSコロナウイルスは、数種のコウモリコロナウイルスに関連しており、数世紀前にこれらの種から分岐したようである[20]。
特に、SARSコロナウイルスは、コウモリコロナウイルスとの関係が他のヒトコロナウイルスより深く、ごく最近の1986年ごろ分岐した[21]。キーンコウモリコロナウイルス類とSARSコロナウイルスの進化経路は、SARS関連コロナウイルスが長期間コウモリで共進化していた可能性を示唆する。SARSコロナウイルスの祖先は、カグラコウモリ科のleaf-nose batsに最初に感染した。その後、キクガシラコウモリ科のhorseshoe batsに、更にはジャコウネコ、最後にヒトに感染した[22][23]。
他のベータコロナウイルスとは異なり、ベータコロナウイルス1およびエンベコウイルス亜属のウシコロナウイルスは、コウモリ由来ではなくネズミに起源を持つと考えられている[15][24]。1790年代には、ウマコロナウイルスが種をまたいでウシコロナウイルスから分岐した[25]
1890年代後半には別の種間伝播が起こり、ヒトコロナウイルスOC43がウシコロナウイルスから分岐した[25][26]。1890年のインフルエンザ大流行は、病原体が実際には特定されていないこと、時期やその神経症状から、インフルエンザウイルスではなく、このウイルス︵のスピルオーバー︶によって引き起こされた可能性があると推測されている[27]。ヒトコロナウイルスOC43は、呼吸器疾患を引き起こすほか、神経疾患への関与が疑われている[28]。現在最も一般的な遺伝子型が出現したのは、1950年代である[29]。
マウスの肝臓と中枢神経系に感染するマウス肝炎ウイルスは、系統的にヒトコロナウイルスOC43とウシコロナウイルスに関連する[30]。ヒトコロナウイルスHKU1も、同様にげっ歯類に起源を持つ[15]。
ヒトコロナウイルス
編集詳細は「ヒトコロナウイルス」を参照
ヒトに感染するコロナウイルスは、風邪症候群の4種類と動物から感染する重症肺炎ウイルス2種類 (SARS-CoV, MERS-CoV) が知られていて[31]、更にSARS-CoV-2を加えた計7種類(2020年3月時点)[32]である。アルファコロナウイルス属、ベータコロナウイルス属の下記のものが知られている[32][注 2]。
- アルファコロナウイルス属
- ヒトコロナウイルス229E:風邪の病原体
- ヒトコロナウイルスNL63:風邪の病原体
- ベータコロナウイルス属
- ヒトコロナウイルスHKU1:風邪の病原体
- ヒトコロナウイルスOC43 :風邪の病原体
- SARSコロナウイルス (SARS-CoV):重症急性呼吸器症候群 (SARSサーズ) の病原体
- MERSコロナウイルス (MERS-CoV):中東呼吸器症候群 (MERSマーズ) の病原体
- SARSコロナウイルス2 (SARS-CoV-2) :新型コロナウイルス感染症 (COVID-19コヴィット・ナインティーン) の病原体
動物コロナウイルス
編集
コロナウイルスは家畜、実験動物、ペット、野生動物などあらゆる動物に感染し、様々な疾患を引き起こす[31]。イヌ、ネコ、ウシ、ブタ、ニワトリ、ウマ、ラクダなどの家畜、シロイルカ、キリン、フェレット、スンクス、コウモリ、スズメなどからも固有のコロナウイルスが検出されている[31]。
家畜
編集
●コロナウイルス科 - Coronaviridae
●アルファコロナウイルス属 - Alphacoronavirus
●ペダコウイルス亜属 (Pedacovirus)
●ブタ流行性下痢ウイルス‥豚流行性下痢ウイルス (PEDV)‥致死的[31]。哺乳豚は下痢に伴う脱水によりほぼ100%が死亡するが、日齢が進むにしたがって症状は軽度[1]。
●テガコウイルス亜属 (Tegacovirus)
●ブタ伝染性胃腸炎ウイルス‥豚伝染性胃腸炎ウイルス (TGEV)‥致死的[31]。7日齢以下は致死率ほぼ100%[1]。
●ベータコロナウイルス属 - Betacoronavirus
●エンベコウイルス亜属 - Embecovirus
●ブタ血球凝集性脳脊髄炎ウイルス (Porcine hemagglutinating encephalomyelitis virus; HEV)‥成豚の症状は軽度[1]。
●牛コロナウイルス[1] - Bovine coronavirus
●ガンマコロナウイルス属 - Gammacoronavirus
●イガコウイルス亜属 - Igacovirus
●鶏コロナウイルス - Avian coronavirus
●鶏伝染性気管支炎ウイルス - Avian infectious bronchitis virus (IBV)‥致死的[31]。幼齢なものほど死亡率も高い[1]。
●ブタ呼吸器コロナウイルス (Porcine respiratory coronavirus; PRCoV)‥不顕性であり症状を示さない[1]。
●ウマコロナウイルス‥死亡率約25%[1]
●シチメンチョウコロナウイルス性腸炎‥幼鳥での死亡率は高い[1]。
実験動物
編集ペット
編集脚注
編集注釈
編集- ^ ただしこれらのウイルスは、単に病原菌として分離されたのみで、電子顕微鏡などで特徴づけられたわけではない。
- ^ ヒト腸コロナウイルス4408 - Human enteric coronavirus 4408 (HECV-4408):については除外する。
出典
編集
(一)^ abcdefghijk家畜・家禽のコロナウイルス病 国立研究開発法人 農業・食品産業技術総合研究機構 動物衛生研究部門 2003年4月14日
(二)^ abAMQ King, ed (2011). “Family Coronaviridae”. Ninth Report of the International Committee on Taxonomy of Viruses. Elsevier, Oxford. pp. 806-828. ISBN 978-0-12-384684-6
(三)^ ICTV Master Species List 2009 - v10 (xls) - International Committee on Taxonomy of Viruses (24 August 2010)
(四)^ “Homology-Based Identification of a Mutation in the Coronavirus RNA-Dependent RNA Polymerase That Confers Resistance to Multiple Mutagens”. Journal of Virology 90(16): 7415-28. (August 2016). doi:10.1128/JVI.00080-16. PMC 4984655. PMID 27279608. "CoVs also have the largest known RNA virus genomes, ranging from 27 to 34 kb (31, 32), and increased fidelity in CoVs is likely required for the maintenance of these large genomes (14)."
(五)^ abcdefgh田口 2011.
(六)^ Tyrell DA, Almeida JD, Berry DM. Cunningham CH, Hamre D, Hofstad MS, Mulluci L and McIntosh K. (1968) Coronaviruses. Nature (Lond.) 220: 650.
(七)^ “Coronaviruses, a New Group of Animal RNA Viruses”. Avian Diseases 14(2): 330-336. (1970). doi:10.2307/1588476. ISSN 0005-2086. JSTOR 1588476.
(八)^ “Human coronaviruses: insights into environmental resistance and its influence on the development of new antiseptic strategies”. Viruses 4(11): 3044-3068. (November 2012). doi:10.3390/v4113044. PMC 3509683. PMID 23202515.
(九)^ “Recovery in tracheal organ cultures of novel viruses from patients with respiratory disease. - PMC”. 2022年5月16日閲覧。
(十)^ “Virology: Coronaviruses”. Nature 220 (5168): 650–650. (1968/11/16). doi:10.1038/220650b0 2022年5月16日閲覧。.
(11)^ Virus Taxonomy: 2019 ReleaseICTV
(12)^ “Structure of SARS coronavirus spike receptor-binding domain complexed with receptor”. Science 309 (5742): 1864-1868. (September 2005). Bibcode: 2005Sci...309.1864L. doi:10.1126/science.1116480. PMID 16166518.
(13)^ “A case for the ancient origin of coronaviruses”. Journal of Virology 87(12): 7039-45. (June 2013). doi:10.1128/JVI.03273-1
2. PMC 3676139. PMID 23596293.
(14)^ “Discovery of seven novel Mammalian and avian coronaviruses in the genus deltacoronavirus supports bat coronaviruses as the gene source of alphacoronavirus and betacoronavirus and avian coronaviruses as the gene source of gammacoronavirus and deltacoronavirus”. Journal of Virology 86(7): 3995-4008. (April 2012). doi:10.1128/JVI.06540-11. PMC 3302495. PMID 22278237.
(15)^ abc“Molecular Evolution of Human Coronavirus Genomes”. Trends in Microbiology 25(1): 35-48. (January 2017). doi:10.1016/j.tim.2016.09.001. PMC 7111218. PMID 27743750. "Specifically, all HCoVs are thought to have a bat origin, with the exception of lineage A beta-CoVs, which may have reservoirs in rodents [2]."
(16)^ “Evidence supporting a zoonotic origin of human coronavirus strain NL63”. Journal of Virology 86(23): 12816-25. (December 2012). doi:10.1128/JVI.00906-12. PMC 3497669. PMID 22993147. "If these predictions are correct, this observation suggests that HCoV-NL63 may have originated from bats between 1190 and 1449 CE."
(17)^ “Distant relatives of severe acute respiratory syndrome coronavirus and close relatives of human coronavirus 229E in bats, Ghana”. Emerging Infectious Diseases 15(9): 1377-84. (September 2009). doi:10.3201/eid1509.090224. PMC 2819850. PMID 19788804. "The most recent common ancestor of hCoV-229E and GhanaBt-CoVGrp1 existed in ≈1686-1800 AD."
(18)^ “Identification and characterization of a novel alpaca respiratory coronavirus most closely related to the human coronavirus 229E”. Viruses 4(12): 3689-700. (December 2012). doi:10.3390/v4123689. PMC 3528286. PMID 23235471.
(19)^ “Molecular Evolution of Human Coronavirus Genomes”. Trends in Microbiology 25(1): 35-48. (January 2017). doi:10.1016/j.tim.2016.09.001. PMC 7111218. PMID 27743750.
(20)^ “Genetic characterization of Betacoronavirus lineage C viruses in bats reveals marked sequence divergence in the spike protein of pipistrellus bat coronavirus HKU5 in Japanese pipistrelle: implications for the origin of the novel Middle East respiratory syndrome coronavirus”. Journal of Virology 87(15): 8638-50. (August 2013). doi:10.1128/JVI.01055-13. PMC 3719811. PMID 23720729.
(21)^ “Evolutionary insights into the ecology of coronaviruses”. Journal of Virology 81(8): 4012-20. (April 2007). doi:10.1128/jvi.02605-06. PMC 1866124. PMID 17267506.
(22)^ “SARS-Coronavirus ancestor's foot-prints in South-East Asian bat colonies and the refuge theory”. Infection, Genetics and Evolution 11(7): 1690-702. (October 2011). doi:10.1016/j.meegid.2011.06.021. PMC 7106191. PMID 21763784.
(23)^ “Evolutionary relationships between bat coronaviruses and their hosts”. Emerging Infectious Diseases 13(10): 1526-32. (October 2007). doi:10.3201/eid1310.070448. PMC 2851503. PMID 18258002.
(24)^ “Discovery of a novel coronavirus, China Rattus coronavirus HKU24, from Norway rats supports the murine origin of Betacoronavirus 1 and has implications for the ancestor of Betacoronavirus lineage A”. Journal of Virology 89(6): 3076-92. (March 2015). doi:10.1128/JVI.02420-14. PMC 4337523. PMID 25552712.
(25)^ ab“Evolutionary dynamics of bovine coronaviruses: natural selection pattern of the spike gene implies adaptive evolution of the strains”. The Journal of General Virology 94(Pt 9): 2036-2049. (September 2013). doi:10.1099/vir.0.054940-0. PMID 23804565. "See Table 1"
(26)^ “Complete genomic sequence of human coronavirus OC43: molecular clock analysis suggests a relatively recent zoonotic coronavirus transmission event”. Journal of Virology 79(3): 1595-604. (February 2005). doi:10.1128/jvi.79.3.1595-1604.2005. PMC 544107. PMID 15650185.
(27)^ “Complete genomic sequence of human coronavirus OC43: molecular clock analysis suggests a relatively recent zoonotic coronavirus transmission event”. Journal of Virology 79(3): 1595-604. (February 2005). doi:10.1128/JVI.79.3.1595-1604.2005. PMC 544107. PMID 15650185. "However, it is tempting to speculate about an alternative hypothesis, that the 1889-1890 pandemic may have been the result of interspecies transmission of bovine coronaviruses to humans, resulting in the subsequent emergence of HCoV-OC43."
(28)^ “Hosts and Sources of Endemic Human Coronaviruses”. Advances in Virus Research 100: 163-188. (2018). doi:10.1016/bs.aivir.2018.01.001. ISBN 9780128152010. PMC 7112090. PMID 29551135.
(29)^ “Molecular epidemiology of human coronavirus OC43 reveals evolution of different genotypes over time and recent emergence of a novel genotype due to natural recombination”. Journal of Virology 85(21): 11325-37. (November 2011). doi:10.1128/JVI.05512-11. PMC 3194943. PMID 21849456.
(30)^ “Mouse hepatitis virus infection of the CNS: a model for defense, disease, and repair”. Frontiers in Bioscience 13(13): 4393-406. (May 2008). doi:10.2741/3012. PMC 5025298. PMID 18508518.
(31)^ abcdefghコロナウイルスとは 国立感染症研究所, Jan 10, 2020 Accessed: 2020-01-20
(32)^ ab“中国・新型コロナ﹁遺伝子情報﹂封じ込めの衝撃”. 東洋経済オンライン (2020年3月5日). 2020年3月6日閲覧。
参考文献
編集
●田口文広︵日本獣医生命科学大学獣医感染症学教室︶﹁特集 Positive Strand RNA Virus のウイルス学 - 3.コロナウイルス﹂︵PDF︶﹃ウイルス﹄第61巻2号、2011年、205-210頁。
●原澤亮 ﹁動物ウイルスの新しい分類 (2005)﹂ ﹃獣医畜産新報﹄58号 921-931頁 2005年 ISSN 0447-0192
●見上彪監修 ﹃獣医感染症カラーアトラス﹄ 文永堂出版 2006年 ISBN 4830032030
●中込治・神谷茂︵編集︶ 編﹃標準微生物学﹄︵第12版︶医学書院、2015年2月15日。ISBN 978-4-260-02046-6。 NCID BB18056640。OCLC 904535631。全国書誌番号:22540957。
●Tyrell DA, Almeida JD, Berry DM. Cunningham CH, Hamre D, Hofstad MS, Mulluci L and McIntosh K. (1968) Coronaviruses. Nature (Lond.) 220: 650.
●Cryo-EM structure of the 2019-nCoV spike in the prefusion conformation, Daniel Wrapp, Nianshuang Wang, Kizzmekia S. Corbett, Jory A. Goldsmith, Ching-Lin Hsieh, Olubukola Abiona, Barney S. Graham, Jason S. McLellan1, Science, 19 Feb 2020: eabb2507, DOI: 10.1126/science.abb2507