核酸

構造
編集一次構造
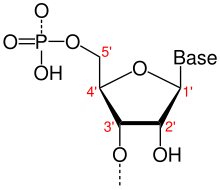
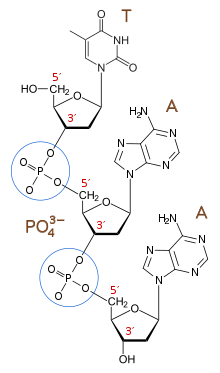
編集核酸の一次構造とは、(デオキシ)ヌクレオシド成分がホスホジエステル結合によって、連続的に連結され、枝分かれのない、ポリヌクレオチド(ヌクレオチドの重合体。核酸と区別して、20程度の短いものを指すことがある)鎖を形成させるような(デオキシ)ヌクレオシド配列である。
二次構造
編集核酸の二次構造とは、一本鎖の主にホモポリヌクレオチド(塩基成分が同一のヌクレオチド重合体)の場合には、塩基間の相互作用によって規定されるヌクレオシド成分の空間的配置をさす。2本の相補鎖の場合には、同一の鎖の隣接塩基間の相互作用と、互いに平行している鎖の対向塩基間の水素結合により安定化された規則的な二重螺旋(DNAには三重、四重螺旋も存在する)を意味する。
三次構造
編集核酸の三次構造は、固定化された二重螺旋とそれ以外のタイプの配列で形成される。
四次構造
編集核酸の四次構造は、リボソームやヌクレオソームのような核蛋白質と相互作用している高分子の空間的配置を意味する。特に、ポリヌクレオチドとポリペプチドの相互依存による高分子構造を指す。
核酸塩基
編集核酸塩基 (nucleobase) は核酸 (DNA, RNA) を構成する塩基成分で、主なものにアデニン、グアニン、シトシン、チミン、ウラシルがあり、それぞれ A, G, C, T, U と略す。構造の骨格からプリン塩基 (A, G) とピリミジン塩基 (C, T, U) とに分けられる。
| 塩基 | 略号 | 分類 | 構造式 | DNA or RNA |
ヌクレオシド | リボヌクレオチド | デオキシリボヌクレオチド |
|---|---|---|---|---|---|---|---|
| アデニン | A | プリン塩基 | DNA and RNA |
アデノシン | アデノシン一リン酸 (AMP) アデノシン二リン酸 (ADP) アデノシン三リン酸 (ATP) |
デオキシアデノシン一リン酸 (dAMP) デオキシアデノシン二リン酸 (dADP) デオキシアデノシン三リン酸 (dATP) | |
| グアニン | G | グアノシン | グアノシン一リン酸 (GMP) グアノシン二リン酸 (GDP) グアノシン三リン酸 (GTP) |
デオキシグアノシン一リン酸 (dGMP) デオキシグアノシン二リン酸 (dGDP) デオキシグアノシン三リン酸 (dGTP) | |||
| チミン | T | ピリミジン塩基 | DNA | チミジン または 5-メチルウリジン |
5-メチルウリジン一リン酸 (TMP) 5-メチルウリジン二リン酸 (TDP) 5-メチルウリジン三リン酸 (TTP) |
チミジン一リン酸 (dTMP) チミジン二リン酸 (dTDP) チミジン三リン酸 (dTTP) | |
| シトシン | C | DNA and RNA |
シチジン | シチジン一リン酸 (CMP) シチジン二リン酸 (CDP) シチジン三リン酸 (CTP) |
デオキシシチジン一リン酸 (dCMP) デオキシシチジン二リン酸 (dCDP) デオキシシチジン三リン酸 (dCTP) | ||
| ウラシル | U | RNA | ウリジン | ウリジン一リン酸 (UMP) ウリジン二リン酸 (UDP) ウリジン三リン酸 (UTP) |
デオキシウリジン一リン酸 (dUMP) デオキシウリジン二リン酸 (dUDP) デオキシウリジン三リン酸 (dUTP) |
核酸やヌクレオチドの構成単位(の繰り返し数)として、たとえば、10塩基(1本鎖の場合)または10塩基対(2重鎖の場合)などと便宜的に用いる。
塩基対における水素結合
編集| 略号 | 塩基(略称の由来) |
|---|---|
| A | アデニン (Adenine) |
| T | チミン (Thymine) |
| G | グアニン (Guanine) |
| C | シトシン (Cytosine) |
| U | ウラシル (Uracil) |
| R | プリン (puRine) |
| Y | ピリミジン (pYrimidine) |
| M | A あるいは C (aMino) |
| K | G あるいは T (Keto) |
| S | G あるいは C (G と C の結合は強い (Strong)) |
| W | A あるいは T (A と T の結合は弱い (Weak)) |
| B | G あるいは T あるいは C (A の次は B) |
| H | A あるいは T あるいは C (G の次は H) |
| V | A あるいは G あるいは C (TU の次は V) |
| D | A あるいは G あるいは T (C の次は D) |
| N | AGTCのどれか (aNy) |
化学的性質
編集変性
編集 の項で詳述する。
また、溶液のイオン強度にも影響を受ける。螺旋分子の主鎖には負電荷を持つリン酸基があり、2本の鎖上のこれらの負電荷は互いに近くにあるので、遮蔽されていなければ鎖同士を反発させようとし、分離を促す。イオン濃度が高いと、陽イオンによって負電荷を遮断し、螺旋は安定化される。
G/C含量が増えるたびに、また溶液のイオン強度が強くなるたびに、変性にかかる時間は増加する。溶液のイオン濃度(他に温度、pHなども)を一定に保てばこの時間は塩基組成に依存するので、測定により、その螺旋構造の安定性を定量化することができる。安定性の指標として主に、温度、pH、塩基組成からの計算などがあり、それぞれ、Tm、pHm、
の項で詳述する。
また、溶液のイオン強度にも影響を受ける。螺旋分子の主鎖には負電荷を持つリン酸基があり、2本の鎖上のこれらの負電荷は互いに近くにあるので、遮蔽されていなければ鎖同士を反発させようとし、分離を促す。イオン濃度が高いと、陽イオンによって負電荷を遮断し、螺旋は安定化される。
G/C含量が増えるたびに、また溶液のイオン強度が強くなるたびに、変性にかかる時間は増加する。溶液のイオン濃度(他に温度、pHなども)を一定に保てばこの時間は塩基組成に依存するので、測定により、その螺旋構造の安定性を定量化することができる。安定性の指標として主に、温度、pH、塩基組成からの計算などがあり、それぞれ、Tm、pHm、 と表す。以下にそれぞれの詳細を記述する。
と表す。以下にそれぞれの詳細を記述する。
 ここでD∞, Dt, D0は、完全に変性した分子の吸光度、ある中間温度でのポリヌクレオチド溶液の吸光度、低温でのポリヌクレオチドの吸光度である。
上で﹁変性完了の瞬間の温度の、中間の温度が融解温度﹂と述べたが、この式から表現すると、融解温度とは﹁螺旋部分の割合と非螺旋部分の割合が等しくなる(θ = 1 - θ = 0.5)温度﹂である。
Tmの値は、一定の外部条件化では一定であり、ためにその構造のみで規定される螺旋分子の安定性を指標することができる。
ここでD∞, Dt, D0は、完全に変性した分子の吸光度、ある中間温度でのポリヌクレオチド溶液の吸光度、低温でのポリヌクレオチドの吸光度である。
上で﹁変性完了の瞬間の温度の、中間の温度が融解温度﹂と述べたが、この式から表現すると、融解温度とは﹁螺旋部分の割合と非螺旋部分の割合が等しくなる(θ = 1 - θ = 0.5)温度﹂である。
Tmの値は、一定の外部条件化では一定であり、ためにその構造のみで規定される螺旋分子の安定性を指標することができる。
pHm
編集- Δ G37°
編集構造安定エネルギーという。上で示した通り、螺旋構造の安定性はG/C含量に依存することを述べたが、実はそれだけでなく、スタッキング相互作用も関与している。水素結合は螺旋の軸に垂直に、スタッキング相互作用はほぼ平行に形成されるため、両者の、安定性への寄与を分けて考えることが可能である。 は37度における構造形成の自由エネルギーを意味し、 は水素結合とスタッキング相互作用の両者の寄与から予想された、安定性の指標の一つである。
この指標はI. Tinocoら[注 3]が1971年に最塩基対モデルとして提案され、このモデルは「核酸の塩基対形成に関して最も影響を与えるのは既に生成している隣の塩基対である」という考えを基本にしている。なぜなら、水素結合の強度は1塩基対における二つの塩基の組み合わせに決定され、スタッキング相互作用は距離の6乗に反比例するので、ある塩基対と隣接塩基対のさらに隣の塩基対との間に働く力は無視できると考えられるためである。すなわち、螺旋構造の安定性は、隣接する塩基対の足し合わせによって求められると考えられた。
螺旋構造において可能な最近接塩基対の組は、DNA/DNAおよびRNA/RNAで10種類、DNA/RNAで16種類である。[2]
- DNA/DNA二重螺旋
DNA DNA DNA DNA
- RNA/RNA二重螺旋
RNA RNA RNA RNA
- RNA/DNA二重螺旋
RNA DNA RNA DNA
もし螺旋構造の安定性がこのモデルに従えば、異なる塩基配列を持つ螺旋分子同士でも、同じ最近接塩基対の組成を持つのなら安定性は等しい。最近接塩基対モデルから、上図に示した最近接塩基対の組の構造安定エネルギーの実験的測定の網羅から、構造安定性は解読されている。
存在
編集ほぼすべての生物に含まれていると考えられる。重量比では、酵母(Baker's yeast/Saccharomyces cerevisiae)、海苔(Purple laver)、白子(鮭)、牡蠣、大豆、肝臓(豚)などでの検出値が高い。[3]
注釈
編集- ^ 蛋白質の変性については変性#変性(生体高分子)参照
- ^ КООПБРАТИВНОСТЬの暫定的和訳。英語ではcooperativeness
- ^ I. Tinoco, Jr., O. C. Uhlenbeck, M. D. Levine
出典
編集- ^ N. K. カチェトコフ/E. I. ブドフスキー 編、橋爪たけし 監訳「核酸の有機化学 上」 1974年 講談社出版
- ^ 下の図のアイディアは杉本直己「遺伝子化学」2002年 p36 に書かれている図3.9から流用
- ^ “Nucleic Acid Contents of Japanese Foods”. NIPPON SHOKUHIN KOGYO GAKKAISHI 36 (11): Table 2. (1989). doi:10.3136/nskkk1962.36.11_934.