酵素反応速度論
表示
| なぜ拡散より速く動態を示す酵素があるのか?[1] |

酵素反応速度論 (こうそはんのうそくどろん) とは酵素によって触媒される化学反応を反応速度の面から研究する学問。酵素の反応速度論を研究することで、酵素反応の機構、代謝における役割、活性調節の仕組み、薬物や毒が酵素をどう阻害するかといったことを明らかにできる。
酵素は通常蛋白質分子であり、他の分子 (酵素の基質という) に作用する。基質は、酵素の活性部位に結合し、段階的に生成物へと変化を遂げる。この過程は、反応機構と呼ばれる。反応機構は、単一基質機構と、複数基質機構に分類できる。
一つの基質としか結合しない酵素、例えばトリオースリン酸イソメラーゼの研究では酵素が基質と結合する際の解離定数や回転率の測定を目指す。
酵素が複数の基質と結合する場合、例えばジヒドロ葉酸還元酵素 (右図) では、基質が結合し、生成物が解離する順序を明らかにすることもできる。1つの基質と結合し、複数の生成物を放出する酵素の例としてプロテアーゼ が挙げられる。この酵素は1つの基質蛋白質を切断して、2つのポリペプチドにする。2つの基質を1つに結合する酵素もある。DNAポリメラーゼはヌクレオチドをDNAに結合する。これらの酵素の反応機構は複雑で何段階にも及ぶことが多いが、通常律速段階があって、これが全体の反応速度を決定する。律速段階は化学反応であったり、(生成物が酵素から離れる際など) 酵素や基質のコンフォメーションの変化であったりする。
酵素の立体構造が分かると、速度論的な情報の解釈に有利である。例えば構造から触媒過程で基質や生成物がどのように結合しているか、反応中にどう変化するかが分かる。また、特定のアミノ酸残基が反応機構で果たす役割が分かることもある。酵素によっては反応中に構造が大きく変化するものがあるが、このような場合は酵素の構造を触媒を受けない基質類似体が結合した場合と、結合していない場合それぞれで決定しておくとよい。
生体で触媒として働くのは蛋白質でできた酵素だけではない。RNAによる触媒、つまりリボソームのようなリボザイムは、RNAスプライシングや翻訳といった多くの過程で不可欠である。リボザイムと酵素の主要な違いは、RNAのほうが触媒できる反応が限られているということだ。もっとも、RNAによる触媒の機構も、蛋白質酵素の場合と同じ方法で解析し、分類することができる。
総論
[編集]
酵素によって触媒される反応でも、その反応物と生成物は触媒を受けていない場合と変わらない。他の触媒と同じく、酵素も反応物と生成物の間の化学平衡を動かすことはない[2]。ただし、触媒されていない反応と違って、酵素触媒下の反応は反応速度に飽和点を持つ。ある酵素濃度のもとで基質濃度が相対的に低いと、反応速度は基質濃度に比例して上昇する。酵素分子は自由に反応を触媒でき、基質濃度が上がれば基質と酵素が出会う頻度も上昇するからだ。ところが基質濃度がある程度高くなると、反応速度は理論的上限値に漸近する。酵素の活性部位は、ほとんど全て基質と結合してしまい、反応速度は酵素固有の回転率によって決まるようになる。この両極端のちょうど中間にあたる基質濃度は、KMで表される。
酵素の速度論的特徴の中で特に重要なのは特定の基質に対して酵素が飽和するのはいつかということと、その時の最大反応速度がどれだけかということである。これらの特徴から、酵素が細胞の中でどんな働きをしており条件が変化したときに酵素がどう反応するかを推察できる。
酵素の試験
[編集]
酵素の試験 (アッセイ) とは、実験室で酵素反応の速度を測ることである。酵素は反応を触媒する過程で消費されることはないため、実際に測るのは基質か生成物の濃度変化である。測定方法にはいろいろある。紫外・可視・近赤外分光法では、生成物と反応物の吸光度の違いを測る。放射能分析では生成物が徐々にできてくるのを放射性元素の取り込みや放出によって測る。分光法は、反応速度を連続的に測定できるので便利である。放射能分析では、サンプルを取り出して測定する必要がある (非連続的分析である) が、多くの場合非常に鋭敏で、ごくわずかな酵素活性でも測定できる[3]。よく似た方法として質量分析器を使う手もあり、基質が生成物へ変化する過程で安定同位体が取り込まれたり放出されるのを観測する。
最も敏感な酵素測定法はレーザを使う方法である。顕微鏡下で酵素1分子にレーザの焦点を合わせ、反応を触媒する過程での変化を観察する。反応中に補因子の蛍光が変化するのを測定したり、蛋白質の一部分を蛍光色素でラベルして触媒中の動きを調べる[4]。こうした実験から酵素単一分子の速度論やダイナミクス (反応中の動き) について新しい知見が得られつつある。従来の方法は数百万にも及ぶ多数の酵素分子の挙動を平均的したものを観察していた[5][6]。
酵素測定における典型的なグラフが左に示してある。反応開始時点では初期速度にしたがって線形に (時間に比例して) 生成物ができる。時間が経つ (グラフの右) と反応速度は低下する。基質が消費され生成物が蓄積するからだ。初期速度で反応が進む時間の長さは、測定条件によって変わり、数ミリ秒のこともあれば、数時間に及ぶこともある。通常の測定ではこの期間が約1分になるようにしておくと実験が容易である。ただし、液体を急速に混ぜ合わせる装置を使えば初期速度の段階が1秒に満たない反応でも測定できる。これをストップフロー法という[7]。これらの高速な測定技術は、後述するように定常状態に達する前の速度を調べるのに不可欠である。
酵素反応速度論の実験では反応のこの初期部分、つまり生成物が時間に比例してできてくる線形部分に注目することが多い。しかし、反応全体を測定してそのデータを非線形の速度方程式に当てはめることもできる[8]。このような測定をprogress-curve 解析という。この方法は初期速度が速すぎて正確に測定できないときに有効である。
単一基質反応
[編集]単一基質機構を持つ酵素としてはトリオースリン酸イソメラーゼや二ホスホグリセリン酸ムターゼのようなイソメラーゼや、アデニル酸シクラーゼやRNAリアーゼであるハンマーヘッドリボザイムのような分子内リアーゼがある。ただし基質が一つしかない酵素でも反応機構は単一基質機構でないことがある。カタラーゼがその例で、まず基質である過酸化水素の1つ目の分子と反応して酵素自身が酸化され、続いて2つ目の分子によって還元される。確かに基質は1種類だが修飾された酵素が中間体として存在している以上、カタラーゼの反応機構はピンポン機構というべきである。これについては複数基質反応として後述する。
ミカエリス-メンテン速度論
[編集]

詳細は「ミカエリス・メンテン式」を参照
酵素によって触媒される反応は飽和を示すので、反応速度は基質を増やしてもそれに比例して線形に増えるわけではない。反応の初期速度を基質濃度 ([S]で表す) を変えながら測定すると、反応速度 (v) は右に示すように、[S] にあわせて上昇する。しかし[S] がさらに増えると酵素は基質で飽和し、反応速度は酵素の最大反応速度 Vmaxに達する。
単一基質反応におけるミカエリス-メンテンのモデルを右に示した。まず酵素Eと基質Sが反応して、酵素基質複合体ESを作る二分子反応が起こる。単分子反応  で表される触媒反応の機構は実際には複雑かもしれないが、通常1つの律速段階があって、触媒作用を速度定数k2を持つ1つの触媒反応で表現できる。
で表される触媒反応の機構は実際には複雑かもしれないが、通常1つの律速段階があって、触媒作用を速度定数k2を持つ1つの触媒反応で表現できる。
![{\displaystyle {\begin{matrix}v=k_{2}[{\mbox{ES}}]\end{matrix}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/befe56463761168b47df5be5bfa796c6d3f6a7dd) (式1).
k2は、kcatや回転数とも呼ばれ、酵素が1秒に行える反応回数の上限を示す。
基質濃度 [S] が小さい場合、酵素は遊離型Eと基質酵素複合体ESの平衡状態にある。[S] を増やすと[E] が減って [ES] が増え、平衡が右に傾く。反応速度は [ES] によって決まるため、[S] のわずかな変化でも反応速度が大きく変わる。しかし、[S] が非常に大きくなると、酵素は完全に基質で飽和し、全て基質酵素複合体ESとなる。こうなると、反応速度 (v≈k2[E]tot=Vmax) は、[S]が少し変化したくらいでは変わらない。ここで、[E]totは、酵素の全濃度であり、
(式1).
k2は、kcatや回転数とも呼ばれ、酵素が1秒に行える反応回数の上限を示す。
基質濃度 [S] が小さい場合、酵素は遊離型Eと基質酵素複合体ESの平衡状態にある。[S] を増やすと[E] が減って [ES] が増え、平衡が右に傾く。反応速度は [ES] によって決まるため、[S] のわずかな変化でも反応速度が大きく変わる。しかし、[S] が非常に大きくなると、酵素は完全に基質で飽和し、全て基質酵素複合体ESとなる。こうなると、反応速度 (v≈k2[E]tot=Vmax) は、[S]が少し変化したくらいでは変わらない。ここで、[E]totは、酵素の全濃度であり、
![{\displaystyle [{\mbox{E}}]_{\mathrm {tot} }\ {\stackrel {\mathrm {def} }{=}}\ [{\mbox{E}}]+[{\mbox{ES}}]}](https://wikimedia.org/api/rest_v1/media/math/render/svg/d6be4bf3259d0b0e0cb5c7ca927bc7d35f6831ad) 飽和条件での [ES] 濃度にほぼ等しい。
ミカエリス-メンテンの式[9]は、反応速度 vが、酵素基質結合の平衡や速度定数 k2とどう関係するかを示す式である。レオノール・ミカエリスとモード・レオノーラ・メンテン は、 k2が k-1 よりもずっと小さいとき (平衡の仮定)、次の式を導いた[10]。
飽和条件での [ES] 濃度にほぼ等しい。
ミカエリス-メンテンの式[9]は、反応速度 vが、酵素基質結合の平衡や速度定数 k2とどう関係するかを示す式である。レオノール・ミカエリスとモード・レオノーラ・メンテン は、 k2が k-1 よりもずっと小さいとき (平衡の仮定)、次の式を導いた[10]。
![{\displaystyle v={\frac {V_{\max[}{\mbox{S}}]}{K_{m}+[{\mbox{S}}]}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/b9fc1a556482d5787a07f8418562cf95ac8ffb37) (式2)
この式は、単一基質機構をもつ酵素ほとんどの速度論の基礎となる。
ミカエリス定数 Kmは、酵素の反応速度が Vmax の半分になるときの基質濃度として定義されている。このことはミカエリス-メンテン式で[S] = Kmを代入してみれば確認できる。反応の律速段階が基質の解離よりもずっと遅い場合 (k2 << k-1)、ミカエリス定数 Kmは複合体ESの解離係数に概ね等しい。ただし、このような状況は比較的まれである。
多くの状況では、k2 > k-1 であり、Briggs-Haldane 状態と呼ばれる[11]。定常状態近似から分かるように、ミカエリス-メンテン式はこれらの状況でも成立する。初期速度の段階では反応速度はほとんど一定であり、[ES] も変化しないことが分かる (式1)。
(式2)
この式は、単一基質機構をもつ酵素ほとんどの速度論の基礎となる。
ミカエリス定数 Kmは、酵素の反応速度が Vmax の半分になるときの基質濃度として定義されている。このことはミカエリス-メンテン式で[S] = Kmを代入してみれば確認できる。反応の律速段階が基質の解離よりもずっと遅い場合 (k2 << k-1)、ミカエリス定数 Kmは複合体ESの解離係数に概ね等しい。ただし、このような状況は比較的まれである。
多くの状況では、k2 > k-1 であり、Briggs-Haldane 状態と呼ばれる[11]。定常状態近似から分かるように、ミカエリス-メンテン式はこれらの状況でも成立する。初期速度の段階では反応速度はほとんど一定であり、[ES] も変化しないことが分かる (式1)。
![{\displaystyle {\frac {d}{dt}}[{\mbox{ES}}]=k_{1}[{\mbox{E}}][{\mbox{S}}]-k_{2}[{\mbox{ES}}]-k_{-1}[{\mbox{ES}}]\approx 0.}](https://wikimedia.org/api/rest_v1/media/math/render/svg/3daed02cecf2893959a37ddb0ef331e2abd2deca) よって、[ES] は下の式であたえられる。
よって、[ES] は下の式であたえられる。
![{\displaystyle [{\mbox{ES}}]\approx {\frac {[{\mbox{E}}]_{\mathrm {tot} }[{\mbox{S}}]}{[{\mbox{S}}]+K_{m}}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/6af63680a6b832123f8fb44e0d4bb3b0e10adb9d) ここでミカエリス定数 Kmの定義は、
ここでミカエリス定数 Kmの定義は、
![{\displaystyle K_{m}\ {\stackrel {\mathrm {def} }{=}}\ {\frac {k_{2}+k_{-1}}{k_{1}}}\approx {\frac {[{\mbox{E}}][{\mbox{S}}]}{[{\mbox{ES}}]}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/428e7c1efa5baf314c725f64da8b0ef44c0034c3) ([E] は遊離の酵素濃度)。まとめると、反応速度 vの一般式は再びミカエリス-メンテン式に戻る。
([E] は遊離の酵素濃度)。まとめると、反応速度 vの一般式は再びミカエリス-メンテン式に戻る。
![{\displaystyle v=k_{2}[\mathrm {ES} ]={\frac {k_{2}[{\mbox{E}}]_{\mathrm {tot} }[{\mbox{S}}]}{[{\mbox{S}}]+K_{m}}}={\frac {V_{\max[}{\mbox{S}}]}{[{\mbox{S}}]+K_{m}}}.}](https://wikimedia.org/api/rest_v1/media/math/render/svg/daeeef493437fcf0ceedb52bf21f19064d63cecf) 特異度定数
特異度定数  は、酵素が基質を生成物に変換する際の効率を示す。ミカエリス定数の定義から、ミカエリス-メンテンの式は次のような形にも書ける。
は、酵素が基質を生成物に変換する際の効率を示す。ミカエリス定数の定義から、ミカエリス-メンテンの式は次のような形にも書ける。
![{\displaystyle v=k_{2}[\mathrm {ES} ]={\frac {k_{2}}{K_{m}}}[{\mbox{E}}][{\mbox{S}}]}](https://wikimedia.org/api/rest_v1/media/math/render/svg/ce445b17dc8f27b81e07db99d3532af0be5c53ee) ここで[E] は遊離の酵素濃度である。つまり、特異度定数とは遊離の酵素が遊離の基質と結合して生成物をつくる際の実質的な2次の速度式である。特異度定数は溶液中で酵素と基質が出会う頻度によって制限されるが、その値は1010 M−1 s−1にも及ぶ[12]。驚くべきことだが、この最大速度は基質や酵素の大きさとはほとんど関係ない[13]。2つの基質があったとき、その特異度係数の違いは酵素がそれぞれの基質を変換する際の効率の違いを定量的に表す。基質濃度 [S] が小さいとき ([S] << Km)、ミカエリス-メンテン式のグラフの傾きから、特異度定数を知ることもできる。
ここで[E] は遊離の酵素濃度である。つまり、特異度定数とは遊離の酵素が遊離の基質と結合して生成物をつくる際の実質的な2次の速度式である。特異度定数は溶液中で酵素と基質が出会う頻度によって制限されるが、その値は1010 M−1 s−1にも及ぶ[12]。驚くべきことだが、この最大速度は基質や酵素の大きさとはほとんど関係ない[13]。2つの基質があったとき、その特異度係数の違いは酵素がそれぞれの基質を変換する際の効率の違いを定量的に表す。基質濃度 [S] が小さいとき ([S] << Km)、ミカエリス-メンテン式のグラフの傾きから、特異度定数を知ることもできる。
 で表される触媒反応の機構は実際には複雑かもしれないが、通常1つの律速段階があって、触媒作用を速度定数k2を持つ1つの触媒反応で表現できる。
で表される触媒反応の機構は実際には複雑かもしれないが、通常1つの律速段階があって、触媒作用を速度定数k2を持つ1つの触媒反応で表現できる。
![{\displaystyle {\begin{matrix}v=k_{2}[{\mbox{ES}}]\end{matrix}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/befe56463761168b47df5be5bfa796c6d3f6a7dd) (式1).
k2は、kcatや回転数とも呼ばれ、酵素が1秒に行える反応回数の上限を示す。
基質濃度 [S] が小さい場合、酵素は遊離型Eと基質酵素複合体ESの平衡状態にある。[S] を増やすと[E] が減って [ES] が増え、平衡が右に傾く。反応速度は [ES] によって決まるため、[S] のわずかな変化でも反応速度が大きく変わる。しかし、[S] が非常に大きくなると、酵素は完全に基質で飽和し、全て基質酵素複合体ESとなる。こうなると、反応速度 (v≈k2[E]tot=Vmax) は、[S]が少し変化したくらいでは変わらない。ここで、[E]totは、酵素の全濃度であり、
(式1).
k2は、kcatや回転数とも呼ばれ、酵素が1秒に行える反応回数の上限を示す。
基質濃度 [S] が小さい場合、酵素は遊離型Eと基質酵素複合体ESの平衡状態にある。[S] を増やすと[E] が減って [ES] が増え、平衡が右に傾く。反応速度は [ES] によって決まるため、[S] のわずかな変化でも反応速度が大きく変わる。しかし、[S] が非常に大きくなると、酵素は完全に基質で飽和し、全て基質酵素複合体ESとなる。こうなると、反応速度 (v≈k2[E]tot=Vmax) は、[S]が少し変化したくらいでは変わらない。ここで、[E]totは、酵素の全濃度であり、
![{\displaystyle [{\mbox{E}}]_{\mathrm {tot} }\ {\stackrel {\mathrm {def} }{=}}\ [{\mbox{E}}]+[{\mbox{ES}}]}](https://wikimedia.org/api/rest_v1/media/math/render/svg/d6be4bf3259d0b0e0cb5c7ca927bc7d35f6831ad) 飽和条件での [ES] 濃度にほぼ等しい。
ミカエリス-メンテンの式[9]は、反応速度 vが、酵素基質結合の平衡や速度定数 k2とどう関係するかを示す式である。レオノール・ミカエリスとモード・レオノーラ・メンテン は、 k2が k-1 よりもずっと小さいとき (平衡の仮定)、次の式を導いた[10]。
飽和条件での [ES] 濃度にほぼ等しい。
ミカエリス-メンテンの式[9]は、反応速度 vが、酵素基質結合の平衡や速度定数 k2とどう関係するかを示す式である。レオノール・ミカエリスとモード・レオノーラ・メンテン は、 k2が k-1 よりもずっと小さいとき (平衡の仮定)、次の式を導いた[10]。
![{\displaystyle v={\frac {V_{\max[}{\mbox{S}}]}{K_{m}+[{\mbox{S}}]}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/b9fc1a556482d5787a07f8418562cf95ac8ffb37) (式2)
この式は、単一基質機構をもつ酵素ほとんどの速度論の基礎となる。
ミカエリス定数 Kmは、酵素の反応速度が Vmax の半分になるときの基質濃度として定義されている。このことはミカエリス-メンテン式で[S] = Kmを代入してみれば確認できる。反応の律速段階が基質の解離よりもずっと遅い場合 (k2 << k-1)、ミカエリス定数 Kmは複合体ESの解離係数に概ね等しい。ただし、このような状況は比較的まれである。
多くの状況では、k2 > k-1 であり、Briggs-Haldane 状態と呼ばれる[11]。定常状態近似から分かるように、ミカエリス-メンテン式はこれらの状況でも成立する。初期速度の段階では反応速度はほとんど一定であり、[ES] も変化しないことが分かる (式1)。
(式2)
この式は、単一基質機構をもつ酵素ほとんどの速度論の基礎となる。
ミカエリス定数 Kmは、酵素の反応速度が Vmax の半分になるときの基質濃度として定義されている。このことはミカエリス-メンテン式で[S] = Kmを代入してみれば確認できる。反応の律速段階が基質の解離よりもずっと遅い場合 (k2 << k-1)、ミカエリス定数 Kmは複合体ESの解離係数に概ね等しい。ただし、このような状況は比較的まれである。
多くの状況では、k2 > k-1 であり、Briggs-Haldane 状態と呼ばれる[11]。定常状態近似から分かるように、ミカエリス-メンテン式はこれらの状況でも成立する。初期速度の段階では反応速度はほとんど一定であり、[ES] も変化しないことが分かる (式1)。
![{\displaystyle {\frac {d}{dt}}[{\mbox{ES}}]=k_{1}[{\mbox{E}}][{\mbox{S}}]-k_{2}[{\mbox{ES}}]-k_{-1}[{\mbox{ES}}]\approx 0.}](https://wikimedia.org/api/rest_v1/media/math/render/svg/3daed02cecf2893959a37ddb0ef331e2abd2deca) よって、[ES] は下の式であたえられる。
よって、[ES] は下の式であたえられる。
![{\displaystyle [{\mbox{ES}}]\approx {\frac {[{\mbox{E}}]_{\mathrm {tot} }[{\mbox{S}}]}{[{\mbox{S}}]+K_{m}}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/6af63680a6b832123f8fb44e0d4bb3b0e10adb9d) ここでミカエリス定数 Kmの定義は、
ここでミカエリス定数 Kmの定義は、
![{\displaystyle K_{m}\ {\stackrel {\mathrm {def} }{=}}\ {\frac {k_{2}+k_{-1}}{k_{1}}}\approx {\frac {[{\mbox{E}}][{\mbox{S}}]}{[{\mbox{ES}}]}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/428e7c1efa5baf314c725f64da8b0ef44c0034c3) ([E] は遊離の酵素濃度)。まとめると、反応速度 vの一般式は再びミカエリス-メンテン式に戻る。
([E] は遊離の酵素濃度)。まとめると、反応速度 vの一般式は再びミカエリス-メンテン式に戻る。
![{\displaystyle v=k_{2}[\mathrm {ES} ]={\frac {k_{2}[{\mbox{E}}]_{\mathrm {tot} }[{\mbox{S}}]}{[{\mbox{S}}]+K_{m}}}={\frac {V_{\max[}{\mbox{S}}]}{[{\mbox{S}}]+K_{m}}}.}](https://wikimedia.org/api/rest_v1/media/math/render/svg/daeeef493437fcf0ceedb52bf21f19064d63cecf) 特異度定数
特異度定数  は、酵素が基質を生成物に変換する際の効率を示す。ミカエリス定数の定義から、ミカエリス-メンテンの式は次のような形にも書ける。
は、酵素が基質を生成物に変換する際の効率を示す。ミカエリス定数の定義から、ミカエリス-メンテンの式は次のような形にも書ける。
![{\displaystyle v=k_{2}[\mathrm {ES} ]={\frac {k_{2}}{K_{m}}}[{\mbox{E}}][{\mbox{S}}]}](https://wikimedia.org/api/rest_v1/media/math/render/svg/ce445b17dc8f27b81e07db99d3532af0be5c53ee) ここで[E] は遊離の酵素濃度である。つまり、特異度定数とは遊離の酵素が遊離の基質と結合して生成物をつくる際の実質的な2次の速度式である。特異度定数は溶液中で酵素と基質が出会う頻度によって制限されるが、その値は1010 M−1 s−1にも及ぶ[12]。驚くべきことだが、この最大速度は基質や酵素の大きさとはほとんど関係ない[13]。2つの基質があったとき、その特異度係数の違いは酵素がそれぞれの基質を変換する際の効率の違いを定量的に表す。基質濃度 [S] が小さいとき ([S] << Km)、ミカエリス-メンテン式のグラフの傾きから、特異度定数を知ることもできる。
ここで[E] は遊離の酵素濃度である。つまり、特異度定数とは遊離の酵素が遊離の基質と結合して生成物をつくる際の実質的な2次の速度式である。特異度定数は溶液中で酵素と基質が出会う頻度によって制限されるが、その値は1010 M−1 s−1にも及ぶ[12]。驚くべきことだが、この最大速度は基質や酵素の大きさとはほとんど関係ない[13]。2つの基質があったとき、その特異度係数の違いは酵素がそれぞれの基質を変換する際の効率の違いを定量的に表す。基質濃度 [S] が小さいとき ([S] << Km)、ミカエリス-メンテン式のグラフの傾きから、特異度定数を知ることもできる。
ミカエリス-メンテン式の線形プロット
[編集]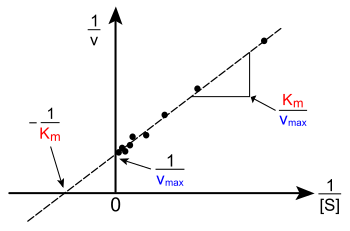
バージニア大学の、ミカエリス-メンテン速度論のチュートリアル (英語) で[注 1]、速度論的パラメータが変わったときの酵素の挙動を試してみることができる。
v と [S] を縦横に取ってグラフを描くと、直線にはならない。[S] が小さいうちは線形だが[S] が大きい部分では飽和し、グラフが曲がってくるのだ。コンピュータで非線形回帰分析ができるようになる前はこの非線形性のせいでKm と Vmax を正確に読み取ることは難しかった。そのため、ラインウィーバー=バークプロット、イーディー=ホフステー図、ヘインズ=ウルフプロット といった線形化手法が編みだされた。これらの表現法は、データを表示するには有用だが、速度係数を求めるためにはもはや使われていない。非線形回帰によってより正確に係数を求めるソフトウェアがあるためだ[14]。
ラインウィーバー=バークプロット︵別名:二重逆数プロット︶は速度論的データを示す際によく使われる。これはミカエリス-メンテン式の両辺の逆数を取ると得られる。右に示したようにミカエリス-メンテン式の線形化であり、y = mx + c 型の直線となる。y 切片は 1/Vmax に等しく、x切片は -1/Km を表す。
![{\displaystyle {\frac {1}{v}}={\frac {K_{m}}{V_{\max[}{\mbox{S}}]}}+{\frac {1}{V_{\max }}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/3ace7013310ad596d2ba08978b8011292459dccc) もちろん、 1/[S] が負となるような部分の数値を測定することはできない。1/[S] = 0 である下限 (y切片) は基質濃度無限大にあたり、右に示すように 1/v=1/Vmax である。また、x切片は、正の濃度における実験データから外挿したものである。しかもラインウィーバー=バークプロットは、基質濃度が低い条件で測定したデータを過大評価しており、Vmax とKmを不正確にする恐れがある[15]。より正確な線形プロットは、イーディー=ホフステー図である。この図ではv を、v/[S] に対してプロットする。3つ目の線形プロットは、ヘインズ・ウルフプロットであり、[S]/v を [S] に対してプロットする。どの場合でも、データを正規化することで、実験の量を減らし、結果の信頼性を改善することができるし、視覚的または数値的な解析にも適している[16]。
もちろん、 1/[S] が負となるような部分の数値を測定することはできない。1/[S] = 0 である下限 (y切片) は基質濃度無限大にあたり、右に示すように 1/v=1/Vmax である。また、x切片は、正の濃度における実験データから外挿したものである。しかもラインウィーバー=バークプロットは、基質濃度が低い条件で測定したデータを過大評価しており、Vmax とKmを不正確にする恐れがある[15]。より正確な線形プロットは、イーディー=ホフステー図である。この図ではv を、v/[S] に対してプロットする。3つ目の線形プロットは、ヘインズ・ウルフプロットであり、[S]/v を [S] に対してプロットする。どの場合でも、データを正規化することで、実験の量を減らし、結果の信頼性を改善することができるし、視覚的または数値的な解析にも適している[16]。
![{\displaystyle {\frac {1}{v}}={\frac {K_{m}}{V_{\max[}{\mbox{S}}]}}+{\frac {1}{V_{\max }}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/3ace7013310ad596d2ba08978b8011292459dccc) もちろん、 1/[S] が負となるような部分の数値を測定することはできない。1/[S] = 0 である下限 (y切片) は基質濃度無限大にあたり、右に示すように 1/v=1/Vmax である。また、x切片は、正の濃度における実験データから外挿したものである。しかもラインウィーバー=バークプロットは、基質濃度が低い条件で測定したデータを過大評価しており、Vmax とKmを不正確にする恐れがある[15]。より正確な線形プロットは、イーディー=ホフステー図である。この図ではv を、v/[S] に対してプロットする。3つ目の線形プロットは、ヘインズ・ウルフプロットであり、[S]/v を [S] に対してプロットする。どの場合でも、データを正規化することで、実験の量を減らし、結果の信頼性を改善することができるし、視覚的または数値的な解析にも適している[16]。
もちろん、 1/[S] が負となるような部分の数値を測定することはできない。1/[S] = 0 である下限 (y切片) は基質濃度無限大にあたり、右に示すように 1/v=1/Vmax である。また、x切片は、正の濃度における実験データから外挿したものである。しかもラインウィーバー=バークプロットは、基質濃度が低い条件で測定したデータを過大評価しており、Vmax とKmを不正確にする恐れがある[15]。より正確な線形プロットは、イーディー=ホフステー図である。この図ではv を、v/[S] に対してプロットする。3つ目の線形プロットは、ヘインズ・ウルフプロットであり、[S]/v を [S] に対してプロットする。どの場合でも、データを正規化することで、実験の量を減らし、結果の信頼性を改善することができるし、視覚的または数値的な解析にも適している[16]。
速度定数の実際的な意義
[編集]
酵素の速度論を研究することは二つの基本的な理由から重要である。まず、酵素がどう働くかを考えるのに役立つ。また、生体内で酵素が示す挙動を予測するのにも役立つ。上のように定義された速度定数 Kmと Vmax は、複数の酵素が共同して代謝を制御する仕組みを理解する上で不可欠である。
このような制御を理解することは、単純な系においてさえ容易ではない。例えば、オキサロ酢酸は、ミトコンドリアの中でリンゴ酸デヒドロゲナーゼによって生成される。その後、リンゴ酸シンターゼ、ホスホエノールピルビン酸カルボキシラーゼ、アスパラギン酸アミノトランスフェラーゼなどによって消費され、それぞれクエン酸回路、糖新生、アスパラギン酸生合成へと進む。それぞれの経路にどれだけのオキサロ酢酸が進むかを予測するには、オキサロ酢酸の濃度だけでなく各酵素の濃度と速度論的係数の情報が不可欠である。代謝経路の動態を予想することの究極の目的は酵素の速度論と遺伝子発現の莫大なデータを1個体全体の数学的モデルとしてまとめあげることにある。この目標は真核生物に関しては、まだまだ先が長いが、バクテリアに関しては既にEscherichia coliのモデルが作られて試験中である[17][18]。
複数基質反応
[編集]複数基質反応の速度方程式は、基質結合の仕方や順序を記述する複雑なものとなる。このような反応を解析する際、1つ目の基質 A の濃度を一定にして2つ目の基質 B の濃度だけを変えると簡単である。この場合、単一基質酵素と同じ挙動になり、v を [S] に対してプロットすると、基質 B に対する見かけ上の定数 Km と Vmax が得られる。いくつかの A の濃度についてこの測定をすれば、反応機構を考察するのに役立つデータが得られる。2つの基質 A と B を生成物 P と Q に変化させる酵素の場合は、反応機構として三重複合体機構とピンポン機構の二つがある。
三重複合体機構
[編集]
このタイプの酵素では基質が2つとも同時に酵素に結合し、E-A-B という三重の複合体を作る。基質結合の順序は、決まっていない場合 (random-order) と決まっている場合 (ordered) がある。三重複合体機構を持つ酵素について、Aの濃度を固定してBの濃度 [S] に対する vのグラフをラインウィーバー=バークプロットで描く。Aの濃度を変えてグラフを何本か描くと、これらの直線は一点で交わる。
三重複合体機構を持つ酵素の例としては、グルタチオン-S-トランスフェラーゼ[19]、ジヒドロ葉酸還元酵素[20]、DNAポリメラーゼ[21]が挙げられる。次のリンク先 (英語) は、ジヒドロ葉酸還元酵素やDNAポリメラーゼの反応機構を短いアニメーションで紹介している[注 2][注 3]。
ピンポン機構
[編集]
右に示したように、ピンポン機構を示す酵素は、Eと修飾型 E* という2つの状態を持つ。修飾型 E* は 反応中間体 と呼ばれる。この機構では、基質Aが結合すると酵素は E* になる。例えば、反応中心に基質の一部が転移し、基質の残りの部分が解離する。1つ目の基質が離れてはじめて2つ目の基質が修飾型酵素 E* に結合し、反応できる。反応した酵素は非修飾型Eに戻る。Aの濃度を固定して、Bの濃度を変えながら [S] に対する vのグラフをラインウィーバー=バークプロットで描く。いくつかのA濃度についてこれを行うと、平行線となる。
ピンポン機構を示す酵素としては、チオレドキシンペルオキシダーゼのような酸化還元酵素[22]、アシルノイラミン酸シチジル転移酵素のような転移酵素[23]、トリプシンやキモトリプシン のようなセリンプロテアーゼ[24]がある。セリンプロテアーゼは非常に普遍的かつ多様な酵素群であり、消化酵素 (トリプシン、キモトリプシン、エラスターゼ) や血液凝固カスケードの酵素の一部も含まれる。セリンプロテアーゼの場合、E* 中間体はアシル-酵素複合体であり、基質蛋白質のペプチド結合に活性部位のセリン残基が求核付加してできる[注 4]。
非ミカエリス-メンテン速度論
[編集]
v を [S] に対してプロットした時 シグモイド曲線 となる酵素がある。これは、活性部位への基質結合が協調的であることを示す場合が多い。つまり、1つの基質結合が、続く基質結合に影響するのである。この現象は、複数の活性中心が相互作用しているような多量体酵素でよく見られる。[25] 協調の機構はヘモグロビンの場合と似ており、基質が1つの活性部位に結合すると、他の活性部位の基質への親和性が変化するのである。正の協調は、1つ目の基質結合が他の活性部位の基質親和性を高める場合に起こる。負の協調は、1つ目の基質結合が他の活性部位の親和性を下げる場合に起きる。
アロステリック酵素の例としては、負の協調を示す哺乳類のチロシンt-RNA合成酵素[26]、正の協調を示すバクテリアの アスパラギン酸トランスカルバモイラーゼ[27]やホスホフルクトキナーゼ[28]が挙げられる。
協調現象は驚くほど普遍的であり、基質濃度の変化にあわせて酵素の反応を変えるのに役立っている。正の協調を示す酵素は [S] に敏感に反応し、基質濃度のわずかな変化で大きな活性変化を見せる。負の協調では、逆に、[S] が少し変化したぐらいでは活性があまり変化しない。
Hill 方程式 は[29]非ミカエリス-メンテン型の速度論において、協調の程度を定量的に表すのによく使われる。この式から得られる Hill 定数 nは、基質の1つの活性部位への結合が他の活性部位での基質結合にどう影響するかを表す。1未満の値は負の協調を表し、1を越える値は正の協調を示す。
前定常状態速度論
[編集]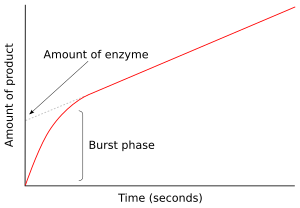
酵素を基質と混合した直後には、生成物はまだできていないし、反応中間体も存在しない。この後数ミリ秒間の反応を扱う学問を、前定常状態速度論という。つまり、前定常状態速度論では、定常状態以前の段階での酵素基質中間体 (ESや E*) の生成と消費を扱う。
この手法が最初に用いられたのはキモトリプシンによる加水分解反応の研究であった。反応機構の調査では、中間体を検出することが動かぬ証拠となることが多い。例えば、前述のピンポン機構では、高速な速度論的測定によって生成物Pの放出や修飾された酵素中間体 E* の生成を検出できる。キモトリプシンの場合、活性部位にある求核性セリンが基質を攻撃することで、アシル酵素中間体ができる。
右図では、酵素から E* が反応開始後数秒のうちに急速にできる。定常状態に達すると速度は低下する。反応が爆発的に進むこの期間 (burst 期) は、酵素の単一回転に相当する。よって、この間に放出される生成物は (グラフのy切片で表されているが)、その測定において活性を持っていた酵素の量をも表す[30]。
化学的機構
[編集]
酵素の速度論を研究する重要な目的の1つが、酵素反応の機構を化学的に説明することである。基質が生成物に至る化学反応の各段階を明らかにするのである。上に述べたような速度論的手法によって、反応中間体がどんな速度で生成し、相互変換するかを明らかにできるが、各中間体が何なのかを確実に決定することはできない。
反応液の条件を変えたり、酵素に変異を加えたり、少し異なる基質を使って速度論的解析を行うと、機構の化学的側面が分かってくることが多い。反応の律速段階や中間体が明らかになるからである。例えば、水素原子との共有結合の切断はよく律速段階となっている。考えられる水素転移のうちどれが律速段階であるかは、水素原子を1つずつ安定同位体 (重水素) に置換することで調べられる。律速段階にかかわる水素原子が置換されると、一次同位体効果によって反応速度が変化する。重水素への結合は通常の水素への結合よりも切れにくいからである[31]。13C/12Cや 18O/16Oなど他の原子についても同じような効果を観測することができるが、水素の場合ほど顕著ではない[32]。
同位体を使えば、基質分子の各部分が生成物分子のどこに対応するかを調べることもできる。例えば、最終産物中の酸素原子の由来を明らかにするのは難しいことが多い。水から来たのかもしれないし、基質から来たかもしれないからだ。これをはっきりさせるには、反応に関与する分子に含まれる酸素原子を、1つ1つ安定同位体 18Oで置換して、生成物中の同位体を調べればよい[33]。酵素の化学的機構を調べるには、溶液のpHを変えながら反応速度や同位体効果を調べるのもよい[34]。金属イオンなどの補因子を変えたり[35]、保存されているアミノ酸残基を選択的に変異させたり、基質アナログの存在下で酵素の挙動を調べるのも有用である[36]。
酵素の阻害
[編集]
可逆的阻害剤
[編集]
可逆的阻害剤は、Km と Vmax に及ぼす影響の違いによって、競合的、不競合的、非競合的、混合型に分類される。これは、阻害剤が結合する相手が、酵素Eなのか、酵素基質複合体ESなのか、両方なのかによって決まる (右図および、下表参照)。阻害剤がどのタイプにあたるのかは、阻害剤濃度を変えながら酵素の反応速度を測れば分かる。阻害剤濃度ごとに ラインウィーバー=バークプロット や イーディー=ホフステー図 を描くと[15]、阻害剤の種類によってグラフの変化のしかたが異なる。記述を簡単にするために、
![{\displaystyle \alpha =1+{\frac {[{\mbox{I}}]}{K_{i}}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/7d05bffbc19ef9047581c87887ae0eea5c11e7fe) and
and ![{\displaystyle \alpha ^{\prime }=1+{\frac {[{\mbox{I}}]}{K_{i}^{\prime }}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/e8b2033b5725a8b85d7425d409192cf11885c199) とおく。ここでKi と K'i はそれぞれ、酵素または基質酵素複合体に対する阻害剤の解離定数である。可逆的阻害剤があると、酵素の見かけ上の KmとVmax は、それぞれ (α/α')Km と (1/α')Vmax に低下する。
とおく。ここでKi と K'i はそれぞれ、酵素または基質酵素複合体に対する阻害剤の解離定数である。可逆的阻害剤があると、酵素の見かけ上の KmとVmax は、それぞれ (α/α')Km と (1/α')Vmax に低下する。
![{\displaystyle \alpha =1+{\frac {[{\mbox{I}}]}{K_{i}}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/7d05bffbc19ef9047581c87887ae0eea5c11e7fe) and
and ![{\displaystyle \alpha ^{\prime }=1+{\frac {[{\mbox{I}}]}{K_{i}^{\prime }}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/e8b2033b5725a8b85d7425d409192cf11885c199) とおく。ここでKi と K'i はそれぞれ、酵素または基質酵素複合体に対する阻害剤の解離定数である。可逆的阻害剤があると、酵素の見かけ上の KmとVmax は、それぞれ (α/α')Km と (1/α')Vmax に低下する。
とおく。ここでKi と K'i はそれぞれ、酵素または基質酵素複合体に対する阻害剤の解離定数である。可逆的阻害剤があると、酵素の見かけ上の KmとVmax は、それぞれ (α/α')Km と (1/α')Vmax に低下する。
| 阻害剤の種類 | 見かけ上の Km | 見かけ上の Vmax | ||
| Ki only | () | 競合的 | ||
| Ki' only | () | 不競合的 | ||
| Ki = Ki' | () | 非競合的 | ||
| Ki ≠ Ki' | () | 混合型 |
非線形回帰によって酵素の速度論的データを上記の速度方程式にフィットすれば[37]、解離定数 Ki と K'i を正確に算出できる。
不可逆的阻害剤
[編集]
阻害剤には、酵素を不可逆的に阻害するものもある。不可逆的阻害剤の多くは、活性部位の残基と共有結合を作って酵素を修飾してしまい、自殺基質と呼ばれる。このような反応は指数的減衰を示し、通常飽和がある。飽和未満では阻害剤との反応は一次反応の速度論を示す。
触媒の機構
[編集]
酵素と基質の相互作用を表すモデルとしてよく使われるのが induced-fit モデルである[38]。このモデルでは、酵素-基質相互作用は始めのうちこそ弱いが、この相互作用によって酵素の立体配座 (コンフォメーション) がまもなく変化し、基質との結合が強くなると考える。また、立体配座の変化のため、活性部位の触媒に関わる残基が基質中の変化を受ける結合に近づく[39]。結合が成立すると、何らかの機構で反応の遷移状態のエネルギーが低下する。つまり、反応の別経路ができる。触媒の機構としては、結合のひずみによる触媒、反応部位同士が近接して適切な位置関係にあることによる効果、活性部位からのプロトン授受による触媒、共有結合を伴う触媒、量子トンネル効果などがある[40]。
酵素の速度論的考察だけでは、触媒の種類を特定することはできない。しかし、速度論的データから可能性を絞りこんだのち、別の手段で確認することができる。例えば、burst 期前定常状態を示すピンポン機構型酵素では共有結合が触媒過程で重要だろうと推測できる。また、pHを変えたときにVmax が大きく変わるが Kmはあまり変化しない酵素の場合、活性部位が特定のイオン化状態にあることが触媒に重要なのだろうと言える。
注釈
[編集]リンク先は英語のサイトである。
- ^ Link: Interactive Michaelis–Menten kinetics tutorial (Java required)
- ^ Link: dihydrofolate reductase mechanism (Gif)
- ^ Link: DNA polymerase mechanism (Gif)
- ^ Link: Chymotrypsin mechanism (Flash required)
参考文献
[編集]- ^ Hsieh M, Brenowitz M (August 1997). “Comparison of the DNA association kinetics of the Lac repressor tetramer, its dimeric mutant LacIadi, and the native dimeric Gal repressor”. J. Biol. Chem. 272 (35): 22092–6. doi:10.1074/jbc.272.35.22092. PMID 9268351.
- ^ Ebbing, D.D. General chemistry (4th edition) Houghton Mifflin Co. 1993, ISBN 0-395-63696-5
- ^ Eisenthal R. Danson M.J. (Eds), Enzyme Assays: A Practical Approach. Oxford University Press (2002) ISBN 0-19-963820-9
- ^ Xie XS, Lu HP. Single-molecule enzymology. J Biol Chem. 1999 Jun 4;274(23):15967-70. PMID 10347141
- ^ Lu H (2004). “Single-molecule spectroscopy studies of conformational change dynamics in enzymatic reactions”. Current pharmaceutical biotechnology 5 (3): 261–9. doi:10.2174/1389201043376887. PMID 15180547.
- ^ Schnell J, Dyson H, Wright P (2004). “Structure, dynamics, and catalytic function of dihydrofolate reductase”. Annual review of biophysics and biomolecular structure 33: 119–40. doi:10.1146/annurev.biophys.33.110502.133613. PMID 15139807.
- ^ Gibson Q.H. Rapid mixing: Stopped flow Methods in Enzymology, (1969) 16:187–228
- ^ Duggleby, R.G. Analysis of enzyme progress curves by non-linear regression. Methods in Enzymology, (1995) 249:61–90.
- ^ Michaelis L. and Menten M.L. Kinetik der Invertinwirkung Biochem. Z. 1913; 49:333–369 English translation Accessed 6 April 2007
- ^ Cha S (1968). “A simple method for derivation of rate equations for enzyme-catalyzed reactions under the rapid equilibrium assumption or combined assumptions of equilibrium and steady state”. J. Biol. Chem. 243 (4): 820–5. PMID 5638598.
- ^ Briggs GE, Haldane JB. A Note on the Kinetics of Enzyme Action. Biochem J. 1925;19(2):338-9. PMID 16743508
- ^ Stroppolo ME, Falconi M, Caccuri AM, Desideri A (2001). “Superefficient enzymes”. Cell. Mol. Life Sci. 58 (10): 1451–60. doi:10.1007/PL00000788. PMID 11693526.
- ^ Davis ME, Madura JD, Sines J, Luty BA, Allison SA, McCammon JA (1991). “Diffusion-controlled enzymatic reactions”. Meth. Enzymol. 202: 473–97. PMID 1784185.
- ^ Jones ME (1992). “Analysis of algebraic weighted least-squares estimators for enzyme parameters”. Biochem. J. 288 ( Pt 2): 533–8. PMID 1463456.
- ^ a b Tseng SJ, Hsu JP. A comparison of the parameter estimating procedures for the Michaelis–Menten model. J Theor Biol. 1990 Aug 23;145(4):457–64. PMID 2246896
- ^ Bravo, I.G., Busto, F., De Arriaga, D., Ferrero, M. A., Rodríguez-Aparicio, L. B., Martínez-Blanco H., Reglero, A. A normalised plot as a novel and time-saving tool in complex enzyme kinetic analysis Biochem. J. (2001). 358, 573–583. PMID 11577687
- ^ Almaas E, Kovacs B, Vicsek T, Oltvai ZN, Barabasi AL. Global organization of metabolic fluxes in the bacterium Escherichia coli. Nature. 2004 Feb 26;427(6977):839-43. PMID 14985762
- ^ Reed JL, Vo TD, Schilling CH, Palsson BO. An expanded genome-scale model of Escherichia coli K-12 (iJR904 GSM/GPR). Genome Biol. 2003;4(9):R54. PMID 12952533
- ^ Dirr H, Reinemer P, Huber R. X-ray crystal structures of cytosolic glutathione S-transferases. Implications for protein architecture, substrate recognition and catalytic function. Eur J Biochem. 1994 Mar 15;220(3):645-61. PMID 8143720
- ^ Stone SR, Morrison JF. Dihydrofolate reductase from Escherichia coli: the kinetic mechanism with NADPH and reduced acetylpyridine adenine dinucleotide phosphate as substrates. Biochemistry. 1988 Jul 26;27(15):5493–9. PMID 3052577
- ^ Fisher PA. Enzymologic mechanism of replicative DNA polymerases in higher eukaryotes. Prog Nucleic Acid Res Mol Biol. 1994;47:371-97. PMID 8016325
- ^ Akerman SE, Muller S. 2-Cys peroxiredoxin PfTrx-Px1 is involved in the antioxidant defence of Plasmodium falciparum. Mol Biochem Parasitol. 2003 Aug 31;130(2):75-81. PMID 12946843
- ^ Bravo, I.G., Barrallo, S., Ferrero, M. A., Rodríguez-Aparicio, L. B., Martínez-Blanco H., Reglero, A. “Kinetic properties of the Acylneuraminate Cytidylytransferase from Pasteurella haemolytica A2”. Biochem. J. (2001) 358, 585-598. [1]
- ^ Kraut J. Serine proteases: structure and mechanism of catalysis. Annu Rev Biochem. 1977;46:331-58. PMID 332063
- ^ Ricard J, Cornish-Bowden A. Co-operative and allosteric enzymes: 20 years on. Eur J Biochem. 1987 Jul 15;166(2):255-72. PMID 3301336
- ^ Ward WH, Fersht AR., Tyrosyl-tRNA synthetase acts as an asymmetric dimer in charging tRNA. A rationale for half-of-the-sites activity. Biochemistry. 1988 Jul 26;27(15):5525–30. PMID 3179266
- ^ Helmstaedt K, Krappmann S, Braus GH., Allosteric regulation of catalytic activity: Escherichia coli aspartate transcarbamoylase versus yeast chorismate mutase. Microbiol. Mol. Biol. Rev. 2001 Sep;65(3):404-21 PMID 11528003
- ^ Schirmer T, Evans PR., Structural basis of the allosteric behaviour of phosphofructokinase. Nature. 1990 Jan 11;343(6254):140-5. PMID 2136935
- ^ Hill, A. V. The possible effects of the aggregation of the molecules of haemoglobin on its dissociation curves. J. Physiol. (Lond.), 1910 40, iv-vii.
- ^ Bender ML, Begue-Canton ML, Blakeley RL, Brubacher LJ, Feder J, Gunter CR, Kezdy FJ, Killheffer JV Jr, Marshall TH, Miller CG, Roeske RW, Stoops JK. The Determination of the Concentration of Hydrolytic Enzyme Solutions : a-Chymotrypsin, Trypsin, Papain, Elastase, Subtilisin, and Acetylcholinesterase. J Am Chem Soc. 1966 Dec 20;88(24):5890-913. PMID 5980876
- ^ Cleland WW. The use of isotope effects to determine enzyme mechanisms. Arch Biochem Biophys. 2005 Jan 1;433(1):2–12. PMID 15581561
- ^ Northrop D (1981). “The expression of isotope effects on enzyme-catalyzed reactions”. Annu. Rev. Biochem. 50: 103–31. doi:10.1146/annurev.bi.50.070181.000535. PMID 7023356.
- ^ Baillie T, Rettenmeier A (1986). “Drug biotransformation: mechanistic studies with stable isotopes”. Journal of clinical pharmacology 26 (6): 448–51. PMID 3734135.
- ^ Cleland WW. Use of isotope effects to elucidate enzyme mechanisms. CRC Crit Rev Biochem. 1982;13(4):385–428. PMID 6759038
- ^ Christianson DW, Cox JD. Catalysis by metal-activated hydroxide in zinc and manganese metalloenzymes. Annu Rev Biochem. 1999;68:33–57. PMID 10872443
- ^ Kraut D, Carroll K, Herschlag D (2003). “Challenges in enzyme mechanism and energetics”. Annu. Rev. Biochem. 72: 517–71. doi:10.1146/annurev.biochem.72.121801.161617. PMID 12704087.
- ^ Leatherbarrow RJ. Using linear and non-linear regression to fit biochemical data. Trends Biochem Sci. 1990 Dec;15(12):455–8. PMID 2077683
- ^ Koshland DE, Application of a Theory of Enzyme Specificity to Protein Synthesis. Proc. Natl. Acad. Sci. U.S.A. 1958 Feb;44(2):98–104. PMID 16590179
- ^ Hammes G (2002). “Multiple conformational changes in enzyme catalysis”. Biochemistry 41 (26): 8221–8. doi:10.1021/bi0260839. PMID 12081470.
- ^ Sutcliffe M, Scrutton N (2002). “A new conceptual framework for enzyme catalysis. Hydrogen tunnelling coupled to enzyme dynamics in flavoprotein and quinoprotein enzymes”. Eur. J. Biochem. 269 (13): 3096–102. doi:10.1046/j.1432-1033.2002.03020.x. PMID 12084049.
参考となる書籍
[編集]以下のリストは英語文献ばかりであるが、邦訳があるかもしれない。
入門書
- Athel Cornish-Bowden, Fundamentals of Enzyme Kinetics. (3rd edition), Portland Press 2004, ISBN 1-85578-158-1.
- Nicholas Price, Lewis Stevens, Fundamentals of Enzymology, Oxford University Press, 1999. ISBN 0-19-850229-X
- Tim Bugg, An Introduction to Enzyme and Coenzyme Chemistry Blackwell Publishing, 2004 ISBN 1-4051-1452-5
発展的な本
- Irwin H. Segel, Enzyme Kinetics : Behavior and Analysis of Rapid Equilibrium and Steady-State Enzyme Systems. Wiley-Interscience; New Ed edition 1993, ISBN 0-471-30309-7.
- Alan Fersht, Structure and Mechanism in Protein Science : A Guide to Enzyme Catalysis and Protein Folding. W. H. Freeman, 1998. ISBN 0-7167-3268-8
- Santiago Schnell, Philip K. Maini, A century of enzyme kinetics: Reliability of the KM and vmax estimates, Comments on Theoretical Biology 8, 169–187, 2004 DOI: 10.1080/08948550390206768
- Chris Walsh, Enzymatic Reaction Mechanisms. W. H. Freeman and Company. 1979. ISBN 0-7167-0070-0
- Paul F. Cook and W.W. Cleland Enzyme kinetics and mechanism Garland Science, 2007 ISBN 0-8153-4140-7
外部リンク
[編集]全て英語である。
- Animation of an enzyme assay — 反応条件を変えたときの変化について
- MACiE — 酵素の反応機構データベース
- ENZYME — Expasy 酵素命名法データベース
- EzCatDB —触媒機構データベース
- BRENDA — 酵素データベースで、基質、阻害剤、反応式を掲載している。
- SABIO-RK - 反応速度論のデータベース
- Joseph Kraut's Research Group, University of California San Diego —いくつかの反応機構のアニメーション
- Symbolism and Terminology in Enzyme Kinetics —酵素速度論における概念と用語の解説
- An introduction to enzyme kinetics — 酵素速度論についてのオンラインチュートリアル
- Enzyme kinetics animated tutorial —音声つきのアニメーションによる酵素の解説










