酵素︵こうそ︶とは、生体でおこる化学反応を触媒する物質である。
 核酸塩基代謝に関与するプリンヌクレオシドフォスフォリラーゼの構造︵リボン図︶
核酸塩基代謝に関与するプリンヌクレオシドフォスフォリラーゼの構造︵リボン図︶
研究者は酵素構造を抽象化したリボン図を基質特異性を考察するときにしばしば利用する。
酵素は生物が物質を消化する段階から吸収・輸送・代謝・排泄に至るまでのあらゆる過程に関与しており、生体が物質を変化させて利用するのに欠かすことができない。
多くの酵素は生体内で作り出されるタンパク質をもとにして構成されている。そのため、生体内での生成や分布の特性、あるいは熱やpHにより変性し活性を失う︵失活︶といった特性などは、他のタンパク質と同様である。
そして生体を機関に例えるならば、核酸塩基配列が表すゲノムが設計図に相当するのに対して、酵素は組み立て工具に相当する。酵素は生化学研究において当初から研究されている一大分野であり、その成果は応用されバイオテクノロジーが生み出したツールとして研究の場面から日常生活まで広く利用されている。
酵素反応︿生化学反応﹀の場である生体内にはさまざまな物質が混在している。それゆえ酵素の特徴である作用する物質︵基質︶をえり好みする性質︵基質特異性︶と目的の反応だけを進行させる性質︵反応選択性︶は、さまざまな化学変化が秩序立って進むことに役立っている。
人類は、古来より発酵という形で酵素を利用してきた。そして今日では、酵素の利用は食品製造だけにとどまらず、化学工業製品の製造や日用品の機能向上など広い分野に応用されている。医療においても、酵素量を検査して診断したり、酵素作用を調節する治療薬を用いるなど、酵素が深く関っている。
役割
細胞内の主要代謝経路
細胞呼吸における酵素の調節機構
(上の経路図の緑・紫矢印部分のみ)
赤点が酵素、黒線が調節機構を表す。丸く配置された赤点がTCAサイクルである。
生体内での酵素の役割は、生命を構成する有機、無機化合物を取り込み、必要な化学反応を引き起こすことにある。生命現象は多くの代謝経路を含み、それぞれの代謝経路は多段階の化学反応からなっている。
小さな細胞内では、その中で起こるさまざまな化学反応を担当する形で多くの種類の酵素がはたらいている。それぞれの酵素は自分の形に合った特定の原料化合物︵基質︶を外から取り込み、担当する化学反応を触媒し、生成物を外へと放出する。そして再び次の反応のために別の基質を取り込む。
ここで放出された生成物は、別の化学反応を担当する酵素の作用を受けて、さらに別の生体物質へと代謝されてゆく。その繰り返しで酵素の触媒反応は進行し、ひいては生命活動が維持されていく。
生体内では化学工業のプラントのように基質と生成物の容器が隔てられているわけではなく、さまざまな物質が渾然一体となって存在している。しかし生命現象をつくる代謝経路でいろいろな化合物が無秩序に反応してしまっては生命活動は維持できない。
したがって酵素は、生体内の物質の中から作用するべき物を選び出さなければならない。また、反応で余分な物を作り出してしまうと周囲に悪影響を及ぼしかねないことから、ある基質に対して起こす反応は1通りでなければならない。酵素は生体内の化学反応を秩序立てて進めるために、このように高度な基質選択性と反応選択性を持つ。
さらにアロステリズム、阻害などにより化学反応の進行を周りから制御する機構を備えた酵素もある。それらの選択性や制御性を持つことで、酵素は渾然とした細胞内で必要なときに必要な原料を選択し、目的の生成物のみを産生するのである。
このように、細胞よりも小さいスケールで組織的な作用をするのが酵素の役割である。人間が有史以前より利用していた発酵も細胞内外で起こる酵素反応の一種である。
発見
ヒトの唾液に含まれるアミラーゼ(リボン図)。薄黄はカルシウムイオン、黄緑は塩化物イオン
エドゥアルト・ブフナー
ノーベル化学賞
エミール・フィッシャー
最初に発見された酵素はジアスターゼ︵アミラーゼ︶で、1832年にA・パヤン (Anselme Payen) とJ・F・ペルソ (Jean Francois Persoz) によるものである。命名も彼らが行った[1]。彼らは翌1833年には麦芽の無細胞抽出液によるでんぷんの糖化を発見し、生命︵細胞︶が存在しなくても、発酵のプロセスの一部が進行することを初めて発見した。
また、1836年にはT・シュワンにより、胃液中にタンパク質分解酵素のペプシンが発見・命名されている[2]。この頃の酵素は生体から抽出されたまま、実体不明の因子として分離・発見されている。
﹁酵素 (enzyme)﹂という語は酵母の中 (in yeast) という意味のギリシア語の"εν ζυμη"に由来するもので、1878年にドイツのウィルヘルム・キューネによって命名された[3]。
19世紀当時、ルイ・パスツールによって、生命は自然発生せず、生命がないところでは発酵︵腐敗︶現象が起こらないこと示されていた。このため﹁有機物は生命の助けを借りなければ作ることができない﹂とする生気説が広く信じられており、酵素作用が生命から切り離すことができる化学反応︵生化学反応︶のひとつにすぎないということは画期的な発見であった。
しかし生物から抽出するしか方法がなく、微生物と同様に加熱すると失活する性質を酵素が持っていたため、その現象は酵素が引き起こしているのか、それとも目に見えない生命︵細胞︶が混入して引き起こしているのかを区別することは困難であった。
このため、酵素が生化学反応を起こすという考え方はすぐには受け入れられず、当時のヨーロッパの学会では酵素の存在を否定するパスツールらの生気説派と酵素の存在を認めるユストゥス・フォン・リービッヒらの発酵素説派とに分かれて論争は続いた。
最終的には、1896年にエドゥアルト・ブフナーが酵母の無細胞抽出物を用いてアルコール発酵を達成したことにより、生気説は完全に否定され、酵素の存在が認知された[4]。
鍵と鍵穴説
上述したように、19世紀後半にはまだ酵素は生物から抽出される実体不明の因子と考えられていたが、酵素の性質に関する研究は進んだ。その研究の早い段階で、酵素の特徴として基質特異性と反応特異性が認識されていた。
これを概念モデルとして集大成したのが、1894年にドイツのエミール・フィッシャーが発表した鍵と鍵穴説である[5]。これは、基質の形状と酵素のある部分の形状が鍵と鍵穴の関係にあり、形の似ていない物質は触媒されない、と酵素の特徴を概念的に表した説である。
現在でも酵素の反応素過程のモデルとして十分に通用する。ただし、フィッシャーはこのモデルの実体がなんであるかについては科学的な実証を行っていない。
酵素の実体の発見
1926年にジェームズ・サムナーがナタマメウレアーゼの結晶化に成功し、初めて酵素の実体を発見した[6]。サムナーは自らが発見した酵素ウレアーゼはタンパク質であると提唱したが、当時サムナーが研究後進国の米国で研究していたこともあり、酵素の実体がタンパク質であるという事実はなかなか認められなかった。
その後、タンパク質からなる酵素が次々と発見され、酵素の実体がタンパク質であるということが広く認められるようになった。
酵素と分子細胞生物学
20世紀後半になると、X線回折を初めとした生体分子の分離・分析技術が向上し、生命現象を分子の構造が引き起す機能として理解する分子生物学と、細胞内の現象を細胞小器官の機能とそれに関係する生体分子の挙動として理解する細胞生物学が成立した。これらの学問によりさらに酵素研究が進展する。すなわち酵素の機能や性質が 、酵素や酵素を形成するタンパク質の構造やそのコンホメーション変化によって説明づけられるようになった。
酵素の機能がタンパク質の構造に起因するものであれば、何らかの酵素に適した構造を持つものは酵素としての機能を発現しうると考えることができる。実際に、1986年にはトーマス・チェックらが、タンパク質以外で初めて酵素作用を示す物質︵リボザイム︶を発見している[7]。
今日においては、この酵素の構造論と機能論に基づいて人工的な触媒作用を持つ超分子︵人工酵素︶を設計、開発する研究も進められている[8][9][10]。
特性
酵素は生体内での代謝経路のそれぞれの生化学反応を担当するために、有機化学で使用されるいわゆる触媒とは異なる基質特異性や反応特異性などの機能上の特性を持つ。
また、酵素はタンパク質を基に構成されているため、他のタンパク質と同様に失活の特性、すなわち熱やpHにより変性し活性を失う特性を持つ。次に酵素に共通の特性である基質特異性と反応特異性、および失活について説明する。
基質特異性
基質に結合する酵素
クリックで拡大・説明
酵素は作用する物質を選択する能力をもち、その特性を基質特異性︵きしつとくいせい、substrate specificity︶と呼ぶ。
たとえば、酵素︵ペプチターゼ;ペプチド分解酵素︶を作用させてタンパク質を分解する場合は、特定の場所のペプチドを加水分解したり、場合によっては基質として認識せずにまったく作用しない場合もある。
一方、タンパク質を︵酵素ではなく︶酸触媒で加水分解する場合は、ペプチド結合の任意の箇所に作用するため、いろいろな長さのアミノ酸配列のペプチドやアミノ酸が生成されることになる。また、ペプチド分解酵素はペプチド結合のみに反応し、他の結合︵エステルやグリコシド結合︶には作用しないが、酸触媒ならばペプチド結合も他の結合も区別することなく同じように分解する。
この特性は酵素研究のごく初期から認識されており、鍵と鍵穴に喩えたモデルで説明されていた。20世紀中旬以降、X線結晶解析で酵素分子の立体構造が特定できるようになり、鍵穴のしくみの手がかりが入手できるようになった。
すなわち、酵素であるタンパク質の立体構造にはさまざまな大きさや形状のくぼみが存在し、それはタンパク質の一次配列︵アミノ酸の配列順序︶に応じて決定付けられている。前述の鍵穴はまさにタンパク質立体構造のくぼみ︵クラフト︶である。酵素は、くぼみに合った基質だけをくぼみの奥に存在する酵素の活性中心へ導くことで、酵素作用を発現する。
今日では、X線結晶解析によって立体構造を決定しなくとも、過去の知見や計算機化学に基づき、タンパク質の一次配列情報やその設計図となる遺伝子の塩基配列情報から立体構造を予測することが可能になりつつある。さらに、生物界に存在しないタンパク質酵素を設計することも可能であるし、タンパク質以外の物質で同様な手法により人工酵素を設計することも可能である。
逆に生物界に存在する酵素に適合する基質を設計すると、各種酵素の阻害剤をつくることも可能となった。酵素や阻害剤が設計できるようになったことは、医薬品や分子生物学研究の発展に役立っている。
誘導適合
基質に結合することで誘導適合する酵素
クリックで拡大・説明
基質の結合した酵素は、それが結合していない酵素よりもエントロピーが減少していると考えられており、事実、基質を結合させた酵素はあらゆるストレス︵熱やpHの変化など︶に対して安定である。これは酵素の立体構造変化が起きているためであると考えられている。
すなわち、基質が結合すると酵素が触媒反応に適した形状に変化すると考えられている。そして酵素の立体構造変化に従い、基質の立体構造も変化し遷移状態へと向かう。そうすると遷移状態に向かう反応の過程がエントロピーの減少とともに促進されることによって反応の活性化エネルギーを低下させていると考えられている。これらの誘導的な化学反応を生じる考え方を誘導適合という。
誘導適合は基質特性を発現する上でも重要であるが、酵素活性発現とも関連し、アロステリック効果などを通じて酵素活性の制御とも関連している。
反応特異性
生体内ではある1つの基質に着目しても、作用する酵素が違えば生成物も変わってくる。通常、酵素は1つの化学反応しか触媒しない性質を持ち、これを酵素の反応特異性と呼ぶ。
酵素が反応特異性を持つために、消化酵素などいくつかの例外を除けば、通常1つの酵素は生体内の複雑な代謝経路の1か所のみを担当している。このことは、生体を恒常的に維持するための重要な性質である。
まず、ある代謝経路が存在するかどうかは、その代謝経路を担当する固有の酵素が存在するかどうかに左右されるので、その酵素タンパク質を産生する遺伝子の発現によって制御できる。また、代謝生産物の1つが過剰になった場合、その代謝経路を担当する固有の酵素の活性にフィードバック阻害がおこるため、過剰な生産が動的に制御される。
酵素はそれぞれに固有の基質と生化学反応を担当するが、同じ生体内でも組織や細胞の種類が異なると、別種の酵素が同じ基質の同じ生化学反応を担当する場合がある。このような関係の酵素を互いにアイソザイム (isozyme) と呼ぶ。
酵素作用の失活
酵素が役割を果たすときあるいはその活性を失う原因には、酵素を構成するタンパク質の立体構造︵コンホメーション︶が深く関与している。失活の原因となる要因としては、熱、pH、塩濃度、溶媒、他の酵素による作用などが知られている。
タンパク質は熱、pH、塩濃度、溶媒など置かれた条件の違いにより容易に立体構造を替えるが、条件が大きく変わると立体構造が不可逆的に大きく変わり、酵素の場合は失活することもある。場合によっては、微生物の汚染によって発生するペプチダーゼなどの消化酵素によりタンバク質の構造が失われ失活することもある。
したがって、酵素反応は至適温度・至適pHや水溶媒など条件が限定される。言い方を変えると、酵素は水中や常温、常圧、中性付近のpHなど温和な条件で化学反応を進行させるともいえる。
ただし、生物の多様性は非常に広いので、好熱菌、好酸性菌、好アルカリ菌などの持つ酵素︵イクストリーモザイム︶のように、極端な温度やpHに耐えうるとされるものもあり、こうした極限環境微生物の応用から酵素の工業利用が現実的になりはじめている。
分類
酵素の分類方法はいくつかあるが、ここでは酵素の所在による分類と、基質と酵素反応の種類(基質特異性と反応特異性の違い)による系統的分類を取り上げる。後者による分類は酵素の命名法と関連している。
所在による分類
酵素は生物体内における反応の全てを起こしているといって過言ではない。そのため、代謝反応の関与する生物体内であれば普遍的に存在し、生体膜︵細胞膜や細胞小器官の膜︶に結合している膜酵素と、細胞質や細胞外に存在する可溶型酵素とに分類される。可溶型酵素のうち、細胞外に分泌される酵素を特に分泌型酵素と呼ぶ。
このような酵素の種類の違いは、酵素以外のタンパク質の種類の違い︵膜タンパク質、分泌型タンパク質︶と同様に、立体構造における疎水性側鎖と親水性側鎖の一次構造上の分布︵タンパク質配列のモチーフ︶の違いによる。他のタンパク質と同様に酵素も細胞内のリボゾームで生合成されるが、モチーフは遺伝子に依存するので、その構造には酵素の進化を反映している。遺伝的に近隣の酵素は類似のモチーフを持ち、酵素群のグループを形成する。
膜酵素
膜酵素の模式図
左から埋没型、貫通型、付着型
可溶型酵素
細胞質に存在している酵素は、水に良く溶け可溶性酵素と呼ばれるものも多い。細胞質での代謝にはこの可溶性酵素が多く関わっている。可溶性酵素は、外部には親水性アミノ酸、内部には疎水性アミノ酸が集まることで、球形の立体構造を取っている場合が多い。
分泌型酵素
酵素は細胞内で産生されるが、産生後に細胞外に分泌されるものもあり、分泌型酵素と呼ばれる。消化酵素が代表例であり、細胞外に存在する物質を取り込みやすいように消化するために分泌される。その形状は可溶性酵素と同じく球形をしている場合が多い。
生物に対して何らかの刺激︵熱、pH、圧力などの変化︶を与えると、その刺激に対してエキソサイトーシスと呼ばれる分泌形態で分泌型酵素を放出する現象が見られる場合がある。構造生物学の進歩において、最初に結晶化され立体構造が決定されていった酵素の多くは分泌型酵素であった。
系統的分類
酵素を反応特異性と基質特異性の違いによって分類すると、系統的な分類が可能となる。このような系統的分類を表す記号として、EC番号がある。
EC番号は"EC"[11] に続けた4個の番号"EC X.X.X.X"︵Xは数字︶による表記がなされるが、左から右にかけて分類が細かくなっていく。EC番号はまず反応特異性を酸化還元反応、転移反応、加水分解反応、解離反応、異性化反応、ATPの補助を伴う合成の6つのグループに分類する。
●EC 1.X.X.X—酸化還元酵素
●EC 2.X.X.X—転移酵素
●EC 3.X.X.X—加水分解酵素
●EC 4.X.X.X—リアーゼ
●EC 5.X.X.X—異性化酵素
●EC 6.X.X.X—リガーゼ
さらに各グループで分類基準は異なるが、反応特異性と基質特異性との違いとで細分化されてゆく。EC番号は全ての酵素についてこの番号が割り振られており、現在約 3,000 種類ほどの反応が見つかっている[12]。
またある活性を担う酵素が他の活性を有することも多く、ATPアーゼなどはATP加水分解反応のほかにタンパク質の加水分解反応への活性も持っている︵EC番号、酸化還元酵素、転移酵素、加水分解酵素、リアーゼ、異性化酵素、リガーゼなどを参照︶。
命名法
酵素の名前は国際生化学連合の酵素委員会によって命名され、同時にEC番号が与えられる。酵素の名称には﹁常用名﹂と﹁系統名﹂が付される。常用名と系統名の違いについて例をあげながら説明する‥
︵例︶次の酵素は全く同じ酵素︵EC番号=EC 1.1.1.1︶
●系統名—アルコール‥NAD+ オキシドレダクターゼ︵酸化還元酵素︶
基質分子の名称︵複数の場合は併記︶と反応の名称を連結して命名される。系統名における反応の名称には規制がある。
●常用名—アルコールデヒドロゲナーゼ︵脱水素酵素︶
系統名と同じ規則で命名されるが、基質の一部を省略して短縮されたりしている。また、命名規則に従わない酵素も多く、DNAポリメラーゼなどはそのひとつである。
古くに発見され命名された酵素については、上述の規則ではなく当時の名称がそのまま使用されている。
ペプシン、トリプシン、キモトリプシン、カタラーゼ
などがこれにあたる。
構成
酵素と補因子の関係
クリックで拡大と説明
酵素はタンパク質から構成されるが、タンパク質のみで構成される場合もあれば、非タンパク質性の構成要素を含むタンパク質︵複合タンパク質︶の場合もある。酵素が複合タンパク質の場合、補因子︵補欠分子族など︶と結合しないと活性が発現しない。言い換えると補因子以外のタンパク質部分であるアポ酵素は活性を持たないが、そのタンパク質部分と補因子とが結合したホロ酵素は活性を持つ。
補因子の例としては、タンパク質と結合した金属イオンやヘム鉄など補欠分子族が組み込まれる場合もあるし、あるいは常時酵素の構造に組み込まれていないが、酵素反応が生じる際に基質と共存することが必要とされる低分子の有機化合物である補酵素の場合もある[13]。たとえば、いくつかのビタミンは補酵素であることが知られている[14]。補因子は酵素との結合の強弱で分類されるが、その境界は曖昧である。
また、酵素を構成するタンパク質鎖︵ペプチド鎖︶は複数本であったり、複数種類であったりする場合がある。複数本のペプチド鎖から構成される場合、立体構造を持つそれぞれのペプチド鎖をサブユニットと呼ぶ。
補欠分子族
酵素はタンパク質から構成されるが、タンパク質だけではなく非タンパク質性の分子である補欠分子族︵ほけつぶんしぞく、prosthetic group︶が活性中心に組み込まれることが必要な酵素も存在する[18]。補欠分子族は無機化合物の場合もあれば有機化合物の場合もある補因子の一種である。そして、補欠分子族が酵素から遊離しうる場合は補酵素と呼ばれる。
カタラーゼ、P450などの活性中心に存在するヘム鉄などが代表的な補欠分子族である。あるいは 金属プロテアーゼの亜鉛イオンなど直接タンパク質と結合している場合もある。生体が要求する微量金属元素は多くの場合で補欠分子族として酵素に組み込まれている。
補酵素
補欠分子族の一種で、酵素の活性中心において酵素反応を補助する非たんぱく質の有機化合物を補酵素︵ほこうそ、coenzyme︶と呼ぶ[19]。酵素の活性中の要素であるため補欠分子族に属するが、酵素から遊離しやすかったり、NADPHのように酵素反応の進行により基質とともに消費される点で典型的な補欠分子族とは性質が異なる。
とはいえ、たとえば、P450酵素のヘム鉄は酵素と共有結合しており遊離しないので補酵素と呼ばないが、酵素と共有結合していても遊離しうるリポ酸は補酵素である。したがって、補酵素であるか補欠分子族であるかの基準は厳密ではない。
酵素タンパク質が熱により変性し失活するのに対して、補酵素は耐熱性を示し、かつ透析により酵素タンパク質より分離することが可能なため、補因子として早い時期からその存在が知られていた。1931年にはオットー・ワールブルクにより初めて補酵素が発見されている。ビタミンあるいはビタミンの代謝物に補酵素となるものが多い。
NAD、NADP、FMN、FAD、チアミン二リン酸、ピリドキサールリン酸、コエンザイムA、リポ酸、葉酸などが代表的な補酵素であり、サプリメントとして健康食品に利用されるものも多い。
サブユニットとアイソザイム
ホウレンソウRubisCOは大サブユニットと小サブユニットのヘテロダイマーの8量体で構成される。(サブユニット毎に色分け)
酵素が複数のペプチド鎖(タンパク質鎖)から構成される場合がある。その場合、各ペプチド鎖はそれぞれ固有の三次構造(立体構造)をとり、サブユニットと呼ばれる。サブユニット構成を酵素の四次構造と呼ぶ場合もある。
ヒト乳酸デヒドロゲナーゼと
アイソザイムタイプ
アイソザイム
タイプ
|
サブユニット
構成
|
組織分布
|
| LD1 |
H4 |
心臓
|
| LD2 |
H3M |
骨格筋
・横隔膜
・腎臓など
|
| LD3 |
H2M2
|
| LD4 |
HM3
|
| LD5 |
M4 |
肝臓
|
例えばヒトにおける乳酸デヒドロゲナーゼ︵LDH; E.C. 1.1.1.27︶は4つのサブユニットから構成される四量体だが、体内組織の位置によりサブユニット構成が異なることが知られている。この場合、サブユニットは心筋型︵H[17]︶と骨格筋型︵M[17]︶の2種類で、そのいずれか4つが組み合わされて乳酸デヒドロゲナーゼが構成される︵例えばH2個とM2個から構成されるH2M2など︶。したがって5タイプの乳酸デヒドロゲナーゼが存在するが、これらは同じ基質で同じ生化学反応を担当するアイソザイムの関係にある。これを応用すると、例えば臨床検査で乳酸デヒドロゲナーゼのアイソザイムタイプを同定︵電気泳動で同定できる︶して、疾患が肝炎であるか心筋疾患であるかを識別することができる。
なお、ここに示した以外の要因︵遺伝子変異による一次構造の変化など︶によってアイソザイムとなる場合もある。
複合酵素
複合酵素の模式図
脂肪酸生成系
クリックで拡大・解説
一連の代謝過程を担当する複数の酵素がクラスターを形成して複合酵素となる場合も多い。
代表例として脂肪酸合成系の複合酵素を示す。これらは [ACP]S-アセチルトランスフェラーゼ (AT; E.C. 2.3.1.38)、マロニルトランスフェラーゼ (MT; E,C.2.3.1.39)、3-オキソアシル-ACPシンターゼI (KS)、3-オキソアシル-ACPレダクターゼ (KR; E.C. 1.1.1.100)、クロトニル-ACPヒドラターゼ (DH; E.C. 4.2.1.58)、エノイル-ACPレダクターゼ (ER; E.C. 1.3.1.10) の6種類の酵素がアシルキャリアタンパク質 (ACP) と共にクラスターとなって複合酵素を形成している。脂肪酸合成系はほとんどが複合酵素で、単独の酵素はアセチルCoAカルボギラーゼ (TE; E.C. 6.4.1.2) のみである[15]。
生化学
酵素反応速度
日本工業規格に﹁酵素は選択的な触媒作用を持つタンパク質を主成分とする生体高分子物質﹂ (JIS K 3600-1310) と定義されているように触媒として利用されるが、化学工業などで用いられる典型的な金属触媒とは反応の特性が異なる。
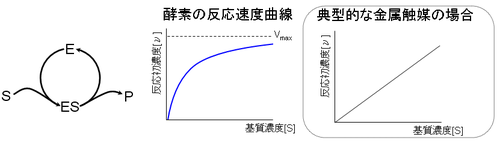
第一に酵素反応の場合、基質濃度[S]が高くなると反応速度が飽和する現象が見られる。酵素の場合、基質濃度を高く変えると、反応速度は飽和最大速度 Vmaxへと至る双曲線を描く。一方、金属触媒の場合、反応初速度[ν]は触媒濃度に依存せず基質濃度[S]の一次式で決定される。
このことは、酵素と金属触媒との粒子状態の違いによって説明できる。金属触媒の場合、触媒粒子の表面は金属原子で覆われており、無数の触媒部位が存在する。それに対して酵素の場合、酵素分子が基質に比べて巨大な場合が多く、活性中心を高々1か所程度しか持たない。そのため金属触媒に比べて、基質と触媒︵酵素︶とが衝突頻度しても︵活性中心に適合し︶反応を起こす頻度が小さい。そして基質濃度が高まると、少ない酵素の活性中心を基質が取り合うようになるので、飽和現象が生じる。このように酵素反応では、酵素と基質が組み合った基質複合体を作る過程が反応速度を決める律速過程になっていると考えられる。
酵素反応の定式化
1913年、L・ミカエリスとM・メンテンは酵素によるショ糖の加水分解反応を測定し、﹁鍵と鍵穴﹂モデルと実験結果から酵素基質複合体モデルを導き出し、酵素反応を定式化した。このモデルによると、酵素は次のように示される。
酵素 (E) + 基質 (S)  酵素基質複合体 (ES) → 酵素 (E) + 生産物 (P)
酵素基質複合体 (ES) → 酵素 (E) + 生産物 (P)
すなわち、酵素反応は、酵素と基質が一時的に結びついて酵素基質複合体を形成する第1の過程と、酵素基質複合体が酵素と生産物とに分離する第2の過程とに分けられる。
極めて分子活性の高い酵素に炭酸脱水酵素があるが、この酵素は1秒当たり百万個の二酸化炭素を炭酸イオンに変化させる ( )。
)。
阻害様式と酵素反応速度
酵素の反応速度曲線を阻害剤のない原系の場合を青線、阻害剤の存在する系を赤線で示す
酵素の反応速度は、基質と構造の似た分子の存在や、後述のアロステリック効果により影響を受ける(阻害される)。阻害作用の種類によって、酵素の反応速度の応答の様式(阻害様式)が変わる。そこで、反応速度や反応速度パラメーターを解析して阻害様式を調べることで、逆にどのような阻害作用を受けているかを識別することができる。どのような阻害様式であるかを調べることによって、酵素がどのような調節作用を受けているか類推することができる。調節作用を研究することは医薬品開発においては酵素作用を制御することにより症状を改善する新たな治療薬の開発に応用されている。
阻害様式は大きく分けると次のように分類される:
- 拮抗阻害(競争阻害)
- 拮抗的ではない阻害
- 混合型阻害
酵素反応の活性化エネルギー
一般に化学反応の進行する方向は化学ポテンシャルが小さくなる方向(エネルギーを消費する方向)に進行し、反応速度は反応の活性化エネルギーが高いか否かに大きく左右される(化学平衡や反応速度論を参照)。
酵素反応は触媒反応で、化学反応の一種なので、その性質は同様である。ただし、一般に触媒反応は化学反応の中でも活性化エネルギーが低いのが通常であるが、酵素反応の活性化エネルギーは特に低いものが多い。
一般に活性エネルギーが15,000cal/molから10,000cal/molに低下すると、反応速度定数はおよそ4.5×107倍になる。
反応機構モデル
酵素の基質特異性はなぜ発揮されるのか、活性化エネルギーをいかにして下げるのかなど、無機触媒や酸塩基触媒などと違う基本的特性を生み出す酵素反応の機構については、いまだ統一的な解答が得られたとはいえない。しかし今日では、構造生物学の発展や組み換えタンパク質作成による変異導入などのテクニックを用いることにより、その片鱗が明らかにされつつある。
タンパク質分解酵素セリンプロテアーゼを例にあげると、基質が酵素に結合することで反応系のエントロピーが減少する働き︵エントロピー・トラップ︶により酵素複合体を形成する。
 キモトリプシンの酸塩基触媒部位
結合した基質は誘導適合により活性中心に反応に適した状態で固定され生成物へと反応が進行する。ここではセリンプロテアーゼりの一種であるキモトリプシンの例を示す。
キモトリプシンの酸塩基触媒部位
結合した基質は誘導適合により活性中心に反応に適した状態で固定され生成物へと反応が進行する。ここではセリンプロテアーゼりの一種であるキモトリプシンの例を示す。
- His57 がプロトンを負に荷電した Asp102 に譲渡する
- His57 が塩基となり、活性中心の Ser195 からプロトンを奪う
- Ser195 が活性化されて(負に荷電して)基質を攻撃する
- His57 がプロトンを基質に譲渡する
- Asp102 から His57 がプロトンを奪い 1. の状態に戻る
遷移状態と抗体酵素
酵素反応において、酵素基質複合体から生成物へと変化する過程では、原子間の結合距離や角度などが変形した分子構造となる遷移状態や反応中間体を経由する。
言い換えると、化学反応がしやすい分子の形状が遷移状態であり、酵素は酵素基質複合体が誘導適合することでその状態を作り出している。遷移状態は活性ポテンシャルの高い状態に相当するので、少ないエネルギーで反応中間体の状態を乗り越えて生成物へと変化する。
遷移状態を作ることが酵素タンパクの主たる役割だとすれば、結合により遷移状態を作り出すことができれば酵素になるとも考えられる。
実際に酵素と同じように分子構造を識別し、その分子と結合する生体物質に抗体がある。1986年、アメリカのトラモンタノらは、酵素と同じ働きをするように意図して製造した抗体が意図通りの酵素作用を示すことを発見し、抗体酵素 (abzyme) と名づけた。
あるいは超分子化合物により、人工酵素を作り出す研究も成果を上げている。
酵素反応の調節機構
生体が酵素活性の大小を制御するには、酵素の量を制御する場合と、酵素の性質を変化させる場合とがある。それらは次のように分類される‥[22]
(一)酵素タンパク質の合成量制御による酵素量の増大
(二)酵素タンパク質が他の生体分子と可逆的に作用することによる酵素活性の変化
(三)酵素タンパク質が修飾されることによる酵素活性の変化
1の調整は遺伝子の発現量の転写調節により実現し、2や3については酵素の質的な変化であり、1の転写制御より素早い応答を示す。
2や3の調節の例として﹁フィードバック阻害﹂が挙げられる。フィードバック阻害により、生産物が過剰になると酵素活性が低減し、生産物が減ると酵素活性は復元する。
利用
酵素は実生活の色々な場面で応用されている。1つは酵素自体を利用するもので、代表的な分野として食品加工業が挙げられる。もう1つは生体が持つ酵素を観測・制御するもので、代表的な分野として医療・製薬業が挙げられる。
食品
チーズの製造にはレンネットが利用される
人間は有史以前から、保存食などを作り出すために、生物の持つ発酵作用を利用してきた。たとえば、味噌や醤油、酒などの発酵食品の製造には、伝統的に麹や麦芽などの生物を利用してきた。これは見方を変えると、生物を利用することで、生物が含む酵素を間接的に利用してきたといえる。今日では、酵素の実体や機能の詳細が判明したので、発酵食品であっても生物を使わずに酵素自体を作用させて製造する場合もあり、酵素を使って食品の性質を意図したように変化させることが可能になっている。
酵素反応は、一般に流通している加工食品の多くにおいて製造工程中に利用されているほか、でん粉を原料とした各種糖類の製造にも用いられている。また、果汁の清澄化や苦味除去、肉の軟化といった品質改良や、リゾチームによる日持ち向上などにも用いられている。最初に発見された酵素であるジアスターゼはアミラーゼの一種であり、消化剤として用いられる。
日用品
今日では、洗剤や化粧品などの日用品に高い付加価値を付けるために酵素が利用される場合が多い。
たとえば洗濯の場合汗しみや食べ物しみは石鹸単独では落としにくい。それは単純な油しみと違って固形物であるタンパク質を含んでおり、しみ成分が固形分と絡まって衣類の繊維に強く接着しているため、界面活性剤だけで洗濯しても汚れを落としきれない。そこで、タンパク質を分解する酵素であるプロテアーゼを含んだ酵素入り洗剤が広く利用されている。
ただし、通常のプロテアーゼは石鹸が溶けたアルカリ性領域では作用しないため、アルカリ性領域で良好に作用する︵至適pHを持つ︶アルカリプロテアーゼが利用されている。
アルカリプロテアーゼは、1947年にオッテセン (M. Ottesen) らが好アルカリ菌から発見した。今日ではアルカリプロテアーゼは酵素入り洗剤用に大量生産されており、工業製品として生産されるプロテアーゼの60%以上を占めるようになっている[15]。
 パパイヤから得るパパイン︵リボン図︶
プロテアーゼ以外には、衣類のセルロース繊維を部分的に分解して汚れが拡散しやすいようにするために、セルラーゼを添加している洗剤もある。
パパイヤから得るパパイン︵リボン図︶
プロテアーゼ以外には、衣類のセルロース繊維を部分的に分解して汚れが拡散しやすいようにするために、セルラーゼを添加している洗剤もある。
同じような例として、食器の洗剤に酵素であるプロテアーゼ︵タンパク質汚れ︶やリパーゼ︵油汚れ︶を添加することで汚れ落ちを増強したり、アミラーゼ︵澱粉質の糊︶を添加することで流水だけで洗浄する自動食器洗浄機でも汚れが落ちるように工夫している例が挙げられる。
化粧品への酵素の応用例としては、脱毛剤にケラチンを分解する酵素パパイン︵プロテアーゼの一種︶を添加することで、皮膚から突出したむだ毛を分解切断する例などがある。
医療
20世紀に入り増大した酵素の知見は、医療や治療薬に劇的な改革をもたらした。ヒトの体内で生じている代謝には酵素が関与しているため、酵素の存在量を測定する臨床検査により疾病を診断することが可能になっている(サブユニットとアイソザイム節の乳酸デヒドロゲナーゼの例を参照)。
また酵素による調節︿ホメオスタシス﹀の失調が病気の原因である場合は、酵素活性を抑制する治療薬によって症状を治療することができる。
あるいは、酵素が欠損する先天性の代謝異常疾患が知られているが、発病前に酵素の量を検査することで、発症を抑える治療を行うことができる︿記事 遺伝子疾患に詳しい﹀。
工業利用の技術(固定化酵素)
また、製品に含まれなくとも食品工業から香料・医薬品原料などファインケミカルの分野まで多方面の食品原料や化成品の製造に利用されている。
 バイオリアクター装置(小型)
たとえば、生体より抽出された酵素を工業化学で利用する際の技術として、酵素の固定化が一般化している。固定化とは、工業用酵素を土台となる物質︵担体︶に固定して用いる方法である。経済的に生産するためには、逆反応がおこらないように反応系から生成物を効率よく除去する必要がある。しかし、このとき同時に酵素も除去してしまうと、本来は再生・再利用可能な触媒である酵素も使い捨てになってしまう。固定化は、この問題を解決する方法である。
バイオリアクター装置(小型)
たとえば、生体より抽出された酵素を工業化学で利用する際の技術として、酵素の固定化が一般化している。固定化とは、工業用酵素を土台となる物質︵担体︶に固定して用いる方法である。経済的に生産するためには、逆反応がおこらないように反応系から生成物を効率よく除去する必要がある。しかし、このとき同時に酵素も除去してしまうと、本来は再生・再利用可能な触媒である酵素も使い捨てになってしまう。固定化は、この問題を解決する方法である。
今日では、固定化酵素は、バイオリアクター技術として食品工業から香料・医薬品原料などファインケミカルの分野まで多方面の化成品の製造に利用されている。バイオリアクターは、ポンプにより基質︵原料︶を注入すると同時に生成物を流出させる生産装置で、酵素を担体とともに柱状の反応装置内に固定することで、酵素のリサイクルの問題や連続生産による経済性の向上などの問題点を解決している。バイオリアクター用の酵素あるいは酵素を含む微生物の固定化には、紅藻類から単離される多糖類のκ-カラギーナン︵食品・化粧品のゲル化剤にも利用される︶が汎用される。
世界で初めて固定化酵素を使った工業化に成功したのは千畑一郎、土佐哲也らであり、1967年にDEAE-Sepadex担体に固定化したアミノアシラーゼ (E.C. 3.5.1.14) を使って、ラセミ体である N-アシル-DL-アミノ酸の混合物から目的の L-アミノ酸のみを不斉加水分解して光学活性なアミノ酸を得る方法を開発した[15]。
バイオセンサー
酵素の基質特異性と反応性を利用して化学物質を検出するセンサーが実用化されている。これらは生体由来の機能を利用することからバイオセンサーと呼ばれ、1960年代に研究が始まり1976年にアメリカでグルコースセンサーが市販されて以来、医療診断や環境測定などの場面で用いられてきた[23]。酵素を用いるバイオセンサーは特に酵素センサーと呼ばれる。
電気化学と酵素の化学が組み合わせられたグルコースセンサーでは、電極の上にグルコースオキシダーゼが固定化されている。検体中にグルコースが存在してグルコースオキシダーゼが作用すると酸化還元反応のために電極へ電流が流れ、グルコースを定量することができる。糖尿病患者が自身の血糖値を調べるために用いる市販の血糖値測定器では、このグルコースセンサーが利用されている。
ほか、蛍光発光、水晶振動子、表面プラズモン共鳴などの原理と酵素とを組み合わせたバイオセンサーが研究されている。
生命の起源と酵素
現存する全ての生物種において、酵素を含む全てのタンパク質の設計図はDNA上の遺伝情報であるゲノムに基づいている。一方、DNA自身の複製や合成にも酵素を必要としている。つまり酵素の存在はDNAの存在が前提であり、一方でDNAの存在は酵素の存在が前提であるから、ゲノムの起源についてはパラドックスが存在していた。最近の研究では、このパラドックスについて、いまだ確証はないものの以下のように説明している。
 リボザイムの作用機序
リボザイムの作用機序
リボザイムは配列を認識してmRNAを特定部位で切断する
1986年にアメリカのトーマス・チェックらによって発見されたリボザイムは、触媒作用を有するRNAであり、次の3種類の反応を触媒することが知られている‥[24]
(一)自分自身に作用してRNAを切断する。︵グループ I, II, III イントロンの自己スプライシング︶
(二)他のRNAに作用してRNAを切断する。︵リボヌクレアーゼP︶
(三)ペプチド結合の形成。︵リボゾーム23S rRNA︶
特性1および2からは、RNAは自己複製していた段階の存在があるとも考えられる。また、特性3からは、RNAが酵素の役割も担う場合があることがわかる。このことから、仮説ではあるが、現在のゲノムの発現機構︵セントラルドグマと言い表される︶が確立する前段階において、遺伝子と酵素との役割を同じRNAが担っているRNAワールドという段階が存在したと考えられている。
なお、特性3の例として挙げた23S rRNAは、大腸菌のタンパク質を合成するリボゾーム内に存在する。大腸菌のリボゾームにおいては、アミノアシルtRNAから合成されるペプチドへアミノ酸を転位・結合させる酵素の活性中心の主役が、タンパク質ではなく23S rRNAとなっている[25]。さらに、この場合の酵素作用︵ペプチジルトランスフェラーゼ活性︶は、23S rRNAのドメインVに依存することも判明している[26]。
また、リボザイムが自己切断する際には鉛イオンが関与する例が判明している。このことから、RNAもタンパク質酵素の補因子と共通の仕組みを持てるという可能性が示唆されている[27]。
RNAワールド説によると、ゲノムを保持する役割はDNAへ、酵素機能はタンパク質へと淘汰が進んで、RNAワールドが今日のセントラルドグマへと進化したと考えられている。その段階では、次のようなRNAの特性が進化の要因として寄与したと推定されている[28]。
遺伝子の保管庫がDNAではなくRNAと考えた場合、RNAには不利な特性がある。それはリボース2'位の水酸基が存在することでエステル交換により環状ヌクレオシド︵環状AMPなど︶を形成してヌクレオチドが切断されやすいという性質を持っている点である。これに対してDNAは、リボース2'位の水酸基を欠くので環状リン酸エステルを形成せず、RNAの場合より安定なヌクレオチドを形成する。
また、立体構造の多様性について考察すると、RNAの立体構造はタンパク質に比べて高次構造が単純になることが判明している。そのため、RNAから構成される酵素に比べ、タンパク質から構成される酵素のほうが立体構造の多様性が大きく、基質特異性の面や遷移状態モデルを形成する上でより性能の良い酵素になると考えられる[29]。
人工酵素
分子構造が分子認識と遷移状態の形成に関与していることが判明して以来、酵素の構造を変化させることで人工的な酵素︵人工酵素︶を作り出す試みがなされている。
そのアプローチ方法としては
(一)酵素たんぱく質の設計を変える方法
(二)超分子化合物を設計する方法
が挙げられる。
前者は1980年代頃から試みられており、アミノ酸配列を変異させて酵素の特性がどのように変化するのか、試行錯誤的に研究がなされた。異種の生物間でゲノムを比較できるようになり、異なる生物に由来する同一酵素について共通性の高い部分とそうでない部分とが明確なったので、それを踏まえて配列を変化させるのである︵いわゆるバイオテクノロジー技術の一環︶。1990年代以降にはコンピュータの大幅な速度向上とデータの大容量化が進行し、実際のタンパク質を測定することなく、コンピュータシミュレーションにより一次配列からタンパク質の立体構造を設計し、物性を予測することができつつある。また、2000年代に入るとゲノムの完全解読が色々な生物種で完了し、遺伝子情報から分子生物学上の問題を解決しようとする試み︵バイオインフォマティクス技術︶がなされている。そして現在、バイオインフォマティクス情報からタンパク質機能を解明するプロテオミックス技術へと応用が展開されつつある。2008年には、計算科学的な手法によって設計された、実際にケンプ脱離の触媒として機能する酵素が報告されている[30]。
次に、超分子化合物を設計する方法については、1980年代頃より、分子認識を行う超分子化合物︵すなわち基質特異性をモデル化した化合物︶の研究が開始された。当初は基質構造の細部までは認識できなかったため、分子の嵩高さを識別することから始められた。ただし早い時期から、他の分子と静電相互作用で結合する包摂化合物︵シクロデキストリンやクラウンエーテルなど︶は知られていた。そこで最初の人工酵素として、リング状の構造を持つシクロデキストリンに活性中心を模倣した側鎖構造を修飾することで、中心空洞に嵌まり込む化合物に対してのみ反応する化学物質が設計された。今日では分子を認識すると蛍光を発するような超分子化合物も設計されている。
また、活性中心で生じている遷移状態を作り出す方法論は反応場理論として体系付けられている。反応場理論の1つの応用が、2001年にノーベル化学賞を受賞した野依良治やバリー・シャープレスらの不斉触媒として成果を挙げている。
代表的な酵素の一覧
酵素に関する年表
出典
(一)^ "amylase", The Columbia Encyclopedia. 6th ed. Bartleby.com.
(二)^ Theodor Schwann. Encyclopædia Britannica. 2007. Encyclopædia Britannica Online.
(三)^ Harper, D (2001). "enzyme". Online Etymology Dictionary.
(四)^ 徳重正信、﹁酵素﹂﹃世界大百科事典﹄(CD-ROM版、第2版)、日立デジタル平凡社、1998年。
(五)^ Fischer E (1894). “Einfluss der Configuration auf die Wirkung der Enzyme”. Ber Dt Chem Ges 27: 2985-93. http://gallica.bnf.fr/ark:/12148/bpt6k90736r/f364.chemindefer.
(六)^ 1946 Nobel prize for Chemistry laureates at http://nobelprize.org
(七)^ 1989 Nobel prize for Chemistrylaureates at http://nobelprize.org
(八)^ Eisenmesser EZ, Bosco DA, Akke M, Kern D (2002). “Enzyme dynamics during catalysis”. Science 295: 1520-3. PMID: 11859194.
(九)^ Agarwal PK (2005). “Role of protein dynamics in reaction rate enhancement by enzymes.”. J Am Chem Soc 127: 15248-56. PMID: 16248667.
(十)^ Eisenmesser EZ, Millet O, Labeikovsky W, Korzhnev DM, Wolf-Watz M, Bosco DA, Skalicky JJ, Kay LE, Kern D (2005). “Intrinsic dynamics of an enzyme underlies catalysis”. Nature 438: 117-21. PMID: 16267559.
(11)^ "E.C."や"EC."と表記される例もある。
(12)^ EC番号は酵素の特性により分類されるので、同じEC番号であっても異なる配列のタンパク質の酵素が含まれる。
(13)^ ﹁補酵素﹂﹃微生物学・分子生物学辞典﹄2版、朝倉書店、1997年。
(14)^ 吉岡 政七, 遠藤 克己﹃新生化学ガイドブック﹄南江堂、1969年、82-119ページ。
(15)^ abcd一島英治 ﹃酵素の化学﹄ 朝倉書店、1995年。ISBN 4-254-14555-1
(16)^ Sang-Hwan Oh, Ganther HE, Hoekstra WG (1974). “Selenium as a Component of Glutathione Peroxidase Isolated
from Ovine Erythrocytest”. Biochemistry 13: 1825-9. [1]
(17)^ abcdefghijklmnopqrstu"Molecule of the Month" RCSB PDB
(18)^ 長倉三郎他編﹁補欠分子族﹂﹃理化学辞典﹄5版、岩波書店、1998年。
(19)^ 長倉三郎他編﹁補酵素﹂﹃理化学辞典﹄5版、岩波書店、1998年。
(20)^ 吉岡 政七, 遠藤 克己﹃新生化学ガイドブック﹄ 南江堂、1969年、89ページ。
(21)^ 1,000cal/molが約4.2kJ/molに相当する。
(22)^ 長倉三郎他編﹁代謝調節﹂ ﹃理化学辞典﹄5版、岩波書店、1998年。
(23)^ 高機能バイオセンサー事業部会編﹃高機能バイオセンサー﹄化学工業日報社、2003年。 ISBN 4873264294
(24)^ T.A. Brown﹃ゲノム 新しい生命情報システムへのアプローチ﹄村松正實監訳、メディカル・サイエンス・インターナショナル、2000年。ISBN 4895922375
(25)^ Nitta I, Ueda T, Watanabe K (1998). “Possivble involvement of Escherichia coli 23S ribosomal RNA in peptide bond formation”. RNA 4: 257–67.
(26)^ Nitta I, Kamada Y, Noda H, Ueda T, Watanabe K (1998). “Reconstitution of peptide bond formation with Escherichia coli 23S ribosomal RNA domains”. Science 281: 666–9. PMID: 9685252.
(27)^ Scott WG, Klug A (1996). “Ribozymes: structure and mechanism in RNA catalysis”. Trends Biochem Sci 21: 351–5.
(28)^ Szathmary E, Smith JM (1993). “The evolution of chromosomes. II. Molecular mechanisms”. J Theoret Biol 164: 447–54.
(29)^ Csermely P (1997). “Proteins, RNAs and chaperones in enzyme evolution: a folding perspective”. Trends Biochem Sci 22: 147–9.
(30)^ Giovanna Ghirlanda, "Old enzymes, new tricks", Nature 453, 164-166 (2008). doi:10.1038/453164a
関連項目

 第一に酵素反応の場合、基質濃度[S]が高くなると反応速度が飽和する現象が見られる。酵素の場合、基質濃度を高く変えると、反応速度は飽和最大速度 Vmaxへと至る双曲線を描く。一方、金属触媒の場合、反応初速度[ν]は触媒濃度に依存せず基質濃度[S]の一次式で決定される。
このことは、酵素と金属触媒との粒子状態の違いによって説明できる。金属触媒の場合、触媒粒子の表面は金属原子で覆われており、無数の触媒部位が存在する。それに対して酵素の場合、酵素分子が基質に比べて巨大な場合が多く、活性中心を高々1か所程度しか持たない。そのため金属触媒に比べて、基質と触媒︵酵素︶とが衝突頻度しても︵活性中心に適合し︶反応を起こす頻度が小さい。そして基質濃度が高まると、少ない酵素の活性中心を基質が取り合うようになるので、飽和現象が生じる。このように酵素反応では、酵素と基質が組み合った基質複合体を作る過程が反応速度を決める律速過程になっていると考えられる。
第一に酵素反応の場合、基質濃度[S]が高くなると反応速度が飽和する現象が見られる。酵素の場合、基質濃度を高く変えると、反応速度は飽和最大速度 Vmaxへと至る双曲線を描く。一方、金属触媒の場合、反応初速度[ν]は触媒濃度に依存せず基質濃度[S]の一次式で決定される。
このことは、酵素と金属触媒との粒子状態の違いによって説明できる。金属触媒の場合、触媒粒子の表面は金属原子で覆われており、無数の触媒部位が存在する。それに対して酵素の場合、酵素分子が基質に比べて巨大な場合が多く、活性中心を高々1か所程度しか持たない。そのため金属触媒に比べて、基質と触媒︵酵素︶とが衝突頻度しても︵活性中心に適合し︶反応を起こす頻度が小さい。そして基質濃度が高まると、少ない酵素の活性中心を基質が取り合うようになるので、飽和現象が生じる。このように酵素反応では、酵素と基質が組み合った基質複合体を作る過程が反応速度を決める律速過程になっていると考えられる。
 酵素基質複合体 (ES) → 酵素 (E) + 生産物 (P)
すなわち、酵素反応は、酵素と基質が一時的に結びついて酵素基質複合体を形成する第1の過程と、酵素基質複合体が酵素と生産物とに分離する第2の過程とに分けられる。
極めて分子活性の高い酵素に炭酸脱水酵素があるが、この酵素は1秒当たり百万個の二酸化炭素を炭酸イオンに変化させる (
酵素基質複合体 (ES) → 酵素 (E) + 生産物 (P)
すなわち、酵素反応は、酵素と基質が一時的に結びついて酵素基質複合体を形成する第1の過程と、酵素基質複合体が酵素と生産物とに分離する第2の過程とに分けられる。
極めて分子活性の高い酵素に炭酸脱水酵素があるが、この酵素は1秒当たり百万個の二酸化炭素を炭酸イオンに変化させる ( )。
)。




 ●1913年 ミカエリス、メンテンらがブラウンとルシャトリエの結果を受けて﹁ミカエリス・メンテン式﹂を発表。
●1925年 G・E・ブリッグスとJ・B・S・ホールデンがミカエリス・メンテン式を発展させた﹁ブリッグス・ホールデンの速度論﹂を発表。
●1926年 アメリカのジェームズ・サムナーがナタ豆から﹁ウレアーゼ﹂と呼ばれる酵素を結晶化して、酵素の本体がタンパク質であることを突き止めた︵ただしこの実験は当時評価されなかった︶。
●1930年 アメリカのジョン・ノースロップがペプシン、トリプシン、キモトリプシンをタンパク質の結晶として抽出した。
●1931年 ドイツのオットー・ワールブルクが、呼吸酵素の特性および作用機構の発見により ノーベル生理学・医学賞を受賞 。
●1913年 ミカエリス、メンテンらがブラウンとルシャトリエの結果を受けて﹁ミカエリス・メンテン式﹂を発表。
●1925年 G・E・ブリッグスとJ・B・S・ホールデンがミカエリス・メンテン式を発展させた﹁ブリッグス・ホールデンの速度論﹂を発表。
●1926年 アメリカのジェームズ・サムナーがナタ豆から﹁ウレアーゼ﹂と呼ばれる酵素を結晶化して、酵素の本体がタンパク質であることを突き止めた︵ただしこの実験は当時評価されなかった︶。
●1930年 アメリカのジョン・ノースロップがペプシン、トリプシン、キモトリプシンをタンパク質の結晶として抽出した。
●1931年 ドイツのオットー・ワールブルクが、呼吸酵素の特性および作用機構の発見により ノーベル生理学・医学賞を受賞 。 ●1945年 アメリカのジョージ・ウェルズ・ビードルとエドワード・ローリー・タータムは1つの遺伝子が1つの酵素に対応することを発表した︵一遺伝子一酵素説︶。
●1946年 サムナーとノースロップは酵素の本体がタンパク質であることを証明し、ノーベル化学賞を受賞した。
●1945年 アメリカのジョージ・ウェルズ・ビードルとエドワード・ローリー・タータムは1つの遺伝子が1つの酵素に対応することを発表した︵一遺伝子一酵素説︶。
●1946年 サムナーとノースロップは酵素の本体がタンパク質であることを証明し、ノーベル化学賞を受賞した。 ●1955年 サンガーらはインスリンの一次構造を決定した。
●1955年 スウェーデンのヒューゴ・テオレルが、酸化酵素の研究により ノーベル生理学・医学賞を受賞 。
●1955年 サンガーらはインスリンの一次構造を決定した。
●1955年 スウェーデンのヒューゴ・テオレルが、酸化酵素の研究により ノーベル生理学・医学賞を受賞 。 ●1960年 アメリカのウィリアム・スタインとスタンフォード・ムーアによって、リボヌクレアーゼのアミノ酸配列が決定された。
●1965年 イギリスのデビッド・フィリップスはリゾチームと基質の複合体の立体構造を明らかにした︵酵素として立体構造が決定されたのはこれが初めて︶。
●1965年 フランスのフランソワ・ジャコブ、アンドレ・ルウォフ、ジャック・モノーが、酵素およびウイルスの合成の遺伝的調節に関する研究により ノーベル生理学・医学賞を受賞 。
●1960年 アメリカのウィリアム・スタインとスタンフォード・ムーアによって、リボヌクレアーゼのアミノ酸配列が決定された。
●1965年 イギリスのデビッド・フィリップスはリゾチームと基質の複合体の立体構造を明らかにした︵酵素として立体構造が決定されたのはこれが初めて︶。
●1965年 フランスのフランソワ・ジャコブ、アンドレ・ルウォフ、ジャック・モノーが、酵素およびウイルスの合成の遺伝的調節に関する研究により ノーベル生理学・医学賞を受賞 。 ●1965年 高崎義幸らが、グルコースイソメラーゼを用いて異性化糖の製造法を発明。
●1968年 H.O.Smith, K.W.ウィルコックスらがDNAの制限酵素を発見した。
●1969年 アメリカのロバート・メリフィールドが、ペプチド固相合成法を用いて、化学的にリポヌクレアーゼを合成した。
●1972年 スタインとムーアは酵素の一次構造決定によりノーベル化学賞を得た。
●1965年 高崎義幸らが、グルコースイソメラーゼを用いて異性化糖の製造法を発明。
●1968年 H.O.Smith, K.W.ウィルコックスらがDNAの制限酵素を発見した。
●1969年 アメリカのロバート・メリフィールドが、ペプチド固相合成法を用いて、化学的にリポヌクレアーゼを合成した。
●1972年 スタインとムーアは酵素の一次構造決定によりノーベル化学賞を得た。 ●1975年 オーストラリア のジョン・コーンフォースが、酵素による触媒反応の立体化学的研究によりノーベル化学賞を受賞 。
●1975年 オーストラリア のジョン・コーンフォースが、酵素による触媒反応の立体化学的研究によりノーベル化学賞を受賞 。 ●1978年、アメリカのダニエル・ネーサンズ 、ハミルトン・スミス、スイスのヴェルナー・アーバーが制限酵素の発見と分子遺伝学への応用により ノーベル生理学・医学賞を受賞 。
●1978年、アメリカのダニエル・ネーサンズ 、ハミルトン・スミス、スイスのヴェルナー・アーバーが制限酵素の発見と分子遺伝学への応用により ノーベル生理学・医学賞を受賞 。 ●1986年 アメリカのトーマス・チェックらによって触媒作用を有するRNAである﹁リボザイム﹂が発見された。これにより、触媒作用はタンパク質によらないという概念ができた。更に生命の起源はRNAから始まったとする﹁RNAワールド仮説﹂の元になっている。
●1986年 アメリカのトラモンタノらは抗体酵素 (abzyme) を発見した。
●1989年 チェックらはリボザイムの発見によりノーベル化学賞を受賞した。
●1986年 アメリカのトーマス・チェックらによって触媒作用を有するRNAである﹁リボザイム﹂が発見された。これにより、触媒作用はタンパク質によらないという概念ができた。更に生命の起源はRNAから始まったとする﹁RNAワールド仮説﹂の元になっている。
●1986年 アメリカのトラモンタノらは抗体酵素 (abzyme) を発見した。
●1989年 チェックらはリボザイムの発見によりノーベル化学賞を受賞した。 ●1992年、スイスのエドモンド・フィッシャー、アメリカのエドヴィン・クレープスが生体制御機構としての可逆的タンパク質リン酸化の発見により︵タンパク質キナーゼ︶ ノーベル生理学・医学賞を受賞 。
●1992年、スイスのエドモンド・フィッシャー、アメリカのエドヴィン・クレープスが生体制御機構としての可逆的タンパク質リン酸化の発見により︵タンパク質キナーゼ︶ ノーベル生理学・医学賞を受賞 。 ●1997年 アメリカのポール・ボイヤー、イギリスのジョン・ウォーカー が、アデノシン三リン酸︵ATP︶の合成の基礎となる酵素機構の解明により︵ATPシンターゼ︶ 、デンマークのイェンス・スコウがイオン輸送酵素、Na+, K+-ATPアーゼの最初の発見によりノーベル化学賞を受賞 。
●1997年 アメリカのポール・ボイヤー、イギリスのジョン・ウォーカー が、アデノシン三リン酸︵ATP︶の合成の基礎となる酵素機構の解明により︵ATPシンターゼ︶ 、デンマークのイェンス・スコウがイオン輸送酵素、Na+, K+-ATPアーゼの最初の発見によりノーベル化学賞を受賞 。