遺伝子
| 遺伝学 |
|---|
 |
生物学において、遺伝子︵いでんし、英: gene、ギリシア語: γένος︶という言葉には2つの意味がある。メンデル遺伝子は、遺伝の基本単位である。分子遺伝子は、DNA内のヌクレオチド配列であり、転写されて機能的なRNAを生成する。この分子遺伝子にはタンパク質コード遺伝子と非コード遺伝子の2種類がある[1][2][3][4]。
遺伝子が発現するとき、まずDNAがRNAに転写される。RNAには直接機能するものもあれば、タンパク質合成の中間鋳型となるものもある。
生物の子孫へ遺伝子を伝達することは、ある世代から次の世代へ表現型形質を継承する基礎をなす。これらの遺伝子は、特定の種の集団からなる遺伝子供給源で、個体ごとに特異的な遺伝型と呼ばれるDNA配列を構成する。遺伝型は、環境因子や発達因子とともに、最終的には個体の表現型を決定する。ほとんどの生物学的な形質は、多遺伝子︵異なる遺伝子の集合︶と遺伝子-環境相互作用が関わる複合的な影響下で発生する。遺伝形質には、花の色や背の高さのようにすぐに分かるものもあれば、血液型や特定の病気のリスク、あるいは生命を構成する何千もの基本的な生化学的過程など、そうでないものもある。
遺伝子はその配列内に変異を獲得し、集団の中でアレルと呼ばれる多様体︵変異体︶をもたらすことがある。これらのアレルは遺伝子のわずかに異なる改版をコード︵符号化︶しており、異なる表現型形質を引き起こす可能性がある[5]。遺伝子は自然選択/適者生存とアレルによる遺伝的浮動によって進化する。
遺伝子という用語は、1909年にデンマークの植物学者、植物生理学者、遺伝学者であるウィルヘルム・ヨハンセンによって導入された[6]。これは、子孫や生殖を意味する古代ギリシャ語の γόνος︵génos︶に由来する[7][8]。
定義[編集]
一口に﹁遺伝子﹂といっても、遺伝、選択、生物学的機能、あるいは分子構造など、さまざまな側面に基づいて、さまざまな用途で使われているが、これらの定義のほとんどはメンデル遺伝子または分子遺伝子の2つに区分される[1][9][10][11][12]。
メンデル遺伝子︵英: Mendelian gene︶は遺伝学の古典的な遺伝子であり、あらゆる遺伝性の形質を指す。これはドーキンスの著書﹃利己的な遺伝子﹄に記述された遺伝子である[13]。
一方、分子遺伝子︵英: molecular gene︶の定義は、生化学、分子生物学、そして遺伝学の大部分にわたって、より一般的に使用されており、DNA配列の観点から説明される遺伝子である[1]。この遺伝子にはさまざまな定義があり、その中には誤解を招くものや不正確なものもある[9][14]。
分子遺伝学となったこの分野のごく初期の研究は、1つの遺伝子が1つのタンパク質を作るという概念を示唆した︵もとは﹁一遺伝子一酵素説﹂︶[15][16]。しかし、1950年代にはリプレッサーRNAを産生する遺伝子が提案され[17]、1960年代の教科書では既に、タンパク質をコードする遺伝子だけでなく、リボソームRNAやtRNAのような機能的RNA分子︵非コード遺伝子︶を含めた分子遺伝子の定義が用いられるようになった[18]。
﹁2種類の遺伝子﹂というこの考え方は、今でもほとんどの教科書で遺伝子の定義の一部となっている。たとえば次のように説明されている。
﹃ゲノムの主な機能はRNA分子を作り出すことである。DNAヌクレオチド配列の選択された部分が、対応するRNAヌクレオチド配列に複写され、タンパク質をコードするか︵mRNAの場合︶、あるいはトランスファーRNA︵tRNA︶やリボソームRNA︵rRNA︶分子のような﹁構造的RNA﹂を形成する。DNAらせんの各領域が、機能的なRNA分子を生成する遺伝子を構成している。﹄[19]
﹃遺伝子を﹁転写されるDNA配列と定義する。この定義には、タンパク質をコードしない遺伝子も含まれる︵すべての転写産物がメッセンジャーRNAというわけではない︶。この定義では通常、転写を制御してもそれ自体は転写されないゲノムの領域は除外される。この遺伝子の定義に対していくつかの例外が見つかるが、驚くことに、完全に満足のゆく定義は存在しない。﹄[20]
﹃遺伝子とは、拡散性の産物をコードするDNA配列である。この産物はタンパク質であったり︵大半の遺伝子が該当︶、RNAである場合もある︵tRNAやrRNAをコードする遺伝子が該当︶。重要な特徴は、その産物が合成部位から拡散して、別の場所で作用することである。﹄[21]
このような定義で重要な部分は︵1︶遺伝子は転写単位に対応すること、︵2︶遺伝子はmRNAと非コードRNAの両方を生成すること、︵3︶調節配列は遺伝子発現を制御するが、遺伝子自体の一部ではないことである。しかし、この定義にはもう一つ重要な部分があり、Kostas Kampourakisの著書﹁Making Sense of Genes﹂で強調されている。
﹃したがって、本書では遺伝子を、タンパク質であれRNA分子であれ、機能的産物の情報をコードするDNA配列として考える。﹁情報をコードする﹂とは、DNAの塩基配列が、ある機能を果たすRNA分子やタンパク質を生成するための鋳型として使われるという意味である。﹄[9]
機能性を強調することが重要なのは、DNAの中には機能的でない転写産物を生成する領域が存在し、それらは遺伝子と見なされないからである。これらには、転写された偽遺伝子のように明白な例だけでなく、転写エラーによってノイズとして生成されるジャンクRNAのような、あまり明白でない例も含まれる。この定義に従うと、真の遺伝子と見なされるためには、その転写物が生物学的機能を持つことが示されなくてはならない[9]。
典型的な遺伝子のサイズに関する初期の推測は、高解像度の遺伝子マッピングとタンパク質やRNA分子のサイズに基づいていた。1965年当時は、1500塩基対という長さが妥当だと考えられた[18]。この数字は、遺伝子は機能性産物の生成に直接関与するDNAであるという考えに基づいていた。1970年代のイントロンの発見は、多くの真核生物の遺伝子が、機能性産物のサイズから推定されるよりもはるかに大きいことを意味した。たとえば、典型的な哺乳類のタンパク質をコードする遺伝子は、長さ︵転写領域︶が約62,000塩基対で、その数は約20,000個であるため、哺乳類ゲノム︵ヒトゲノムを含む︶の約35-40%を占める[22][23][24]。
タンパク質コード遺伝子も、非コード遺伝子も、50年以上前から知られているにもかかわらず、遺伝子をタンパク質を特定するDNA配列として定義している教科書、ウェブサイト、科学出版物がいまだに数多く存在している。言い換えれば、その定義はタンパク質コード遺伝子に限定されたものである。次の引用は、アメリカン・サイエンティスト 誌の最近の記事からの一例である。
... de novo遺伝子の潜在的な重要性を真に評価するために、我々はほぼすべての専門家が同意できる﹁遺伝子﹂という言葉の厳密な定義に頼った。まず、ヌクレオチド配列が真の遺伝子とみなされるには、オープンリーディングフレーム︵ORF︶が存在しなければならない。ORFは﹁遺伝子そのもの﹂と考えることができる。ORFは、すべての遺伝子に共通する開始マーカーで始まり、3種類ある終了マーカーのうちの1つで終わる。この過程における重要な酵素の一つであるRNAポリメラーゼは、モノレールの列車のようにDNA鎖に沿って走行し、DNAをメッセンジャーRNAの形式に転写する。この点が2つ目の重要な基準である。真の遺伝子とは、転写と翻訳の両方が行われるものをいう。つまり、真の遺伝子は、まず一時的なメッセンジャーRNAを作るための鋳型として使われ、次にそれがタンパク質に翻訳されるものである[25]。
この限定された定義は一般的であり、そのためこの﹁標準的な定義﹂を批判し、非コード遺伝子を含む新しい拡張された定義を求める多くの最近の論文が発表されるほどであった[26][27][28]。しかし、このいわゆる﹁新しい﹂定義は半世紀以上も前から存在するものであり、なぜ現代の著者が非コード遺伝子を無視しているのか明確に説明されていない。[独自研究?]
一部の定義は他の定義よりも広範に適用できるかもしれないが、生物学の基本的な複雑さは、遺伝子の定義がすべての側面を完璧に捉えることができないことを意味する。すべてのゲノムがDNAであるわけではない︵たとえばRNAウイルスを参照︶[29]。細菌のオペロンは単一の大きなmRNAに転写される複数のタンパク質コード領域であり、選択的スプライシングは一つのゲノム領域で複数の区域産物をコードすることを可能とし、トランススプライシングによりゲノム全体に分割された短いコード配列からmRNAが連結される[30][31][32]。分子的定義では、イントロン、プロモーター、その他の調節領域などの要素は除外されているため、これらの要素は遺伝子と﹁関連﹂し、その機能に影響を与えるものと考えられている。
このような多様で複雑な現象を包含するために、より広範な実用的定義が用いられることもある。この場合、遺伝子は、潜在的に重畳する機能産物がまとまった集まりをコードするゲノム配列の連合体として定義される[33]。この定義は、遺伝子を特定のDNA座位ではなく、その機能的産物︵タンパク質またはRNA︶によって分類し、調節エレメントは遺伝子関連領域として分類する[33]。
歴史[編集]
詳細は「遺伝学の歴史」を参照
離散的遺伝単位の発見[編集]

離散的遺伝単位︵英: discrete inheritable units︶の存在は、グレゴール・メンデル︵1822-1884︶によって初めて提案された[34]。1857年から1864年にかけて、オーストリア帝国のブルノ︵現在のチェコ共和国︶で、彼は8,000株の一般的な食用エンドウ豆の遺伝パターンを研究し、親から子への明確な形質を追跡した。彼はこれらの遺伝パターンを2n 個の組み合わせとして数学的に説明した︵ここで、nは元のエンドウ豆の異なる特徴の数︶。彼は遺伝子という言葉は使用しなかったが、観察可能な物理的特徴をもたらす離散的な遺伝単位という観点から自らの結果を説明した。この説明は、ウィルヘルム・ヨハンセンによる遺伝型︵生物の遺伝物質︶と表現型︵その生物の観察可能な形質︶の区別を先取りしていた。メンデルはまた、独立組合せ︵英: independent assortment︶、顕性形質︵英: dominant traits︶と潜性形質︵英: recessive traits︶の区別、ヘテロ接合体とホモ接合体の区別、および不連続遺伝の現象を初めて実証した。
メンデルの研究以前は、遺伝の主流は融合遺伝説であり、受精の過程でそれぞれの親が体液を提供し、両親の形質が混ざり合って子孫を残すと考えられていた[35]。チャールズ・ダーウィンは、ギリシャ語のパン︵pan、すべて・全体︶とジェネシス︵genesis、誕生︶/ジェノス︵genos、起源︶を語源とするパンゲネシス︵pangenesis、パンゲン説︶と呼ばれる遺伝理論を展開した[36][37]。ダーウィンは、ジェミュール︵gemmule︶という言葉を使って、生殖の際に混ざり合う仮説上の粒子を表現した。
メンデルの研究は1866年に初めて発表されて以来、ほとんど注目されることはなかったが、19世紀後半にユーゴー・ド・フリース、カール・エーリヒ・コレンス、エーリヒ・フォン・チェルマクらによって再発見され、彼らは自らの研究で同様の結論に達した︵と主張した︶[38]。具体的には、1889年、ユーゴー・ド・フリースは著書﹃Intracellular Pangenesis︵細胞内パンゲネシス︶﹄を出版し[39]、その中で、さまざまな性質が個別の遺伝的キャリアを持ち、生物における特定の形質の継承は粒子で行われると仮定した。ド・フリースは、ダーウィンの1868年のパンゲネシス理論にちなんで、これらの単位をパンゲネス︵pangenes、独: pangens︶と呼んだ。
20年後の1909年、ウィルヘルム・ヨハンセンが﹁遺伝子︵gene︶﹂という語句を[6]、1906年にはウィリアム・ベイトソンが﹁遺伝学︵genetics︶﹂という語句を導入したが[40][33]、特にエドゥアルト・シュトラスブルガーなどは、依然として遺伝の基本的な物理的/機能的単位に﹁パンゲン︵pangene︶﹂という語句を使用していた[39]:訳者前書き(viii)。
DNAの発見[編集]
遺伝子と遺伝に対する理解の進歩は20世紀を通して続いた。1940年代から1950年代の実験によって、デオキシリボ核酸︵DNA︶が遺伝情報の分子的記憶装置であることが示された[41][42]。DNAの構造は、ロザリンド・フランクリンとモーリス・ウィルキンスによってX線結晶学を使用して研究され、そのことが、ジェームズ・ワトソンとフランシス・クリックが、対になった核酸塩基による遺伝子複製の機構について説得力のある仮説を示す二本鎖DNA分子モデルを発表するきっかけとなった[43][44]。 1950年代初期、染色体内の遺伝子に対する一般的な見解は、糸で繋がれたビーズのように配置された別個の要素のように機能するというものであった。バクテリオファージT4のrII領域に欠損を持つ変異体を用いたベンザーの実験︵1955-1959︶は、個々の遺伝子が単純な直線構造を持ち、DNAの直線部分に相当する可能性が高いことを示した[45][46]。 この一連の研究により、タンパク質はDNAから転写されたRNAから翻訳されるという分子生物学におけるセントラル・ドグマが確立された。その後、この定説にはレトロウイルスにおける逆転写 (en:英語版) のような例外があることが示されている。DNAレベルでの遺伝学の現代的研究は、分子遺伝学と呼ばれている。 1972年、ウォルター・ファイヤーズと彼のチームは、バクテリオファージMS2のコートタンパク質の遺伝子配列を初めて決定した[47]。その後、1977年に、フレデリック・サンガーによって鎖終結DNAシークエンシング︵サンガー法︶が開発され、配列決定の効率を向上させる日常的な実験ツールになった[48]。サンガー法の自動化バージョンは、ヒトゲノム計画の初期段階で使用された[49]。現代的総合とその後継者たち[編集]
詳細は「現代的総合 (20世紀)」を参照
メンデル遺伝学とダーウィン進化論を統合するために20世紀初頭に開発された理論は、ジュリアン・ハクスリーによって導入された用語で現代的総合︵英: modern synthesis︶と呼ばれている[50]。
このような進化観は、ジョージ・C・ウィリアムズの遺伝子中心の進化観によって強調された。彼は、メンデル遺伝子を﹁認識できる頻度で分離し、組み換わるもの﹂と定義し、それは自然選択の選択単位︵生物学的組織の階層における生物学的実体︶であると提案した[51]:24。メンデル遺伝子の中心性と進化における自然選択の重要性を強調する関連した考えは、リチャード・ドーキンスによって広められた[13]。
1960年代後半に進化中立説が発展したことで、無作為な遺伝的浮動が進化における主要な担い手であり、中立説は分子進化の帰無仮説とあるべきという認識につながった[52]。これが系統樹の構築と、DNA配列を使用したすべての年代測定技術の基礎となる分子時計の開発につながった。これらの技術は分子遺伝子の配列に限定されるものではなく、ゲノムのすべてのDNAセグメントに適用することができる。
分子基盤[編集]
詳細は「デオキシリボ核酸 (DNA)」を参照

DNA[編集]
大半の生物は、DNA︵デオキシリボ核酸︶の長い鎖にその遺伝子をコード化︵符号化︶している。DNAは、4種類のヌクレオチドサブユニットからなる鎖で構成されており、それぞれのサブユニットは、五炭糖︵2-デオキシリボース︶、リン酸基、そしてアデニン、シトシン、グアニン、チミンの4塩基のうちの1つから構成されている[53]:2.1。
DNAの2本の鎖が互いにねじり合わさってDNA二重らせんを形成し、リン酸-糖主鎖が外側をらせん状に回り、塩基は内側に向いてアデニンはチミンと、グアニンはシトシンと塩基対をなしている。塩基対形成による特異性は、アデニンとチミンが2本の水素結合を形成するのに対し、シトシンとグアニンは3本の水素結合を形成することから生じる。したがって、二重らせんの2本の鎖は相補的でなければならず、一方の鎖のアデニンともう一方の鎖のチミンが対になるように、またグアニンとシトシンも同様に、塩基の配列が一致していなければならない:4.1[53]。
塩基のペントース残基の化学組成に起因して、DNA鎖には方向性が生じる。DNAポリマーの一端には、デオキシリボース上に露出したヒドロキシ基があり、これは分子の3'末端と呼ばれている。もう一方の末端には露出したリン酸基があり、これは5'末端と呼ばれる。二重らせんの2本の鎖は反対方向に走っている。DNAの複製や転写を含む核酸合成は5'→3'方向に行われるが、その理由は、露出した3'-ヒドロキシを求核剤とした脱水反応によって新しいヌクレオチドが付加されるからである[54]:27.2。
DNAにコードされた遺伝子の発現は、遺伝子をRNAに転写することから始まる。RNAはDNAと非常によく似た構造を持っている第二の核酸であるが、そのモノマーはデオキシリボースではなくリボース糖を含んでいる。また、RNAには、チミンの代わりにウラシル塩基も含まれている。RNA分子はDNAよりも安定性が低く、一般的に一本鎖で存在する。タンパク質をコードする遺伝子は、コドンと呼ばれる3連のヌクレオチド配列で構成されており、遺伝子の﹁言語﹂における﹁単語﹂として役割を果たす。遺伝暗号は、タンパク質が翻訳される際のコドンとアミノ酸との対応関係を規定する。遺伝暗号はすべての既知の生物でほぼ同じである[53]:4.1。
染色体[編集]


詳細は「核型」を参照
生物体または細胞内の遺伝子の総体をゲノムといい、1本または複数の染色体に保存することができる。染色体は、単一の非常に長いDNAらせんで、その内部には数千もの遺伝子がコード化されている[53]:4.2。特定の遺伝子が存在する染色体の領域を座位︵または遺伝子座︶と呼ぶ。各座位には遺伝子のアレル︵英: allele、対立遺伝子︶が1つずつ存在するが、集団のメンバーの中にはその座位に異なるアレルが存在し、遺伝子の配列がわずかに異なる場合がある。
真核生物の遺伝子の大部分は、一連の大きな線状染色体に保存されている。染色体は、ヒストンと呼ばれる貯蔵タンパク質と複合体となって核内に詰め込まれ、ヌクレオソームと呼ばれる単位を形成する。このようにパッケージ化されて凝縮したDNAはクロマチンと呼ばれる[53]:4.2。DNAがヒストン上に保存される方法や、ヒストン自体の化学的修飾によって、DNAの特定の領域が遺伝子発現に利用可能かどうかが調節される。遺伝子に加えて、真核生物の染色体には、細胞分裂の際にDNAが末端領域を分解することなく複製され、娘細胞に確実に振り分けられるようにするための配列、すなわち複製起点、テロメア、セントロメアが含まれている[53]:4.2。複製起点は、染色体の2つの複製を作成するためにDNA複製が開始される配列領域である。テロメアは、線状染色体の末端を覆う長い反復配列で、DNA複製時にコーディング領域や調節領域の分解を防ぐ役割を担う。テロメアの長さはゲノムが複製されるたびに短縮し、老化プロセスに関与していると考えられている[56]。セントロメアは、細胞分裂の際に姉妹染色分体を娘細胞に分離する紡錘糸を結合するために必要である[53]:18.2。
原核生物︵細菌と古細菌︶は通常、一本の大きな環状染色体にゲノムを格納している。同様に、真核生物の細胞小器官の中には、少数の遺伝子を含む残余の環状染色体を持つものもある[53]:14.4。原核生物は、プラスミドと呼ばれる小さな環状DNAで染色体を補うことがある。プラスミドは通常、少数の遺伝子しかコードしておらず、個体間で転移可能である。たとえば、抗生物質耐性の遺伝子は通常、細菌のプラスミド上にコードされており、遺伝子の水平伝播によって、︵たとえ異なる種の細胞間であっても︶個々の細胞間で受け継ぐことができる[57]。
原核生物の染色体が比較的、遺伝子密度が高いのに対し、真核生物の染色体は明らかな機能を持たないDNA領域を含むことがよくある。単細胞の単純な真核生物にはこのようなDNAは比較的少ないが、ヒトを含む複雑な多細胞生物のゲノムには、機能が特定されていないDNAが絶対的に多く含まれている[58]。このDNAはしばしば﹁ジャンクDNA﹂と呼ばれてきた。しかし、より最近の分析によると、タンパク質をコードするDNAはヒトゲノムの2%を占めるに過ぎないものの、ゲノム中の塩基の約80%が発現している可能性があるため、ジャンクDNAという言葉は誤用である可能性が示唆されている[30]。
構造と機能[編集]
構造[編集]
|
|
タンパク質コード遺伝子は多くの要素から構成されており、実際のタンパク質コード配列は多くの場合、そのごく一部にしか過ぎない。これらには、成熟mRNAのイントロンや非翻訳領域が含まれる。非コード遺伝子にもイントロンが含まれることがあり、それらは機能的の成熟RNAを産生する過程で除去される。
すべての遺伝子は、その発現に必要な調節配列と関連付けられている。まず、遺伝子にはプロモーター配列が必要である。プロモーターは転写因子によって認識・結合される。転写因子はさらにRNAポリメラーゼを動員して、その領域に結合して転写を開始するのを助ける[53]:7.1。この認識は通常、TATAボックスのようなコンセンサス配列として行われる。遺伝子は複数のプロモーターを持つことができ、その結果、5'末端の長さが異なるメッセンジャーRNA︵mRNA︶が生成する[60]。高度転写遺伝子は、転写因子と強い結合を形成する﹁強い﹂プロモーター配列があり、それによって転写が高速に開始される。他の遺伝子は、転写因子との結合が弱く、転写を開始する頻度が低い﹁弱い﹂プロモーターを持っている[53]:7.2。真核生物のプロモーター領域は、原核生物のプロモーターよりもはるかに複雑で、同定するのは難しい[53]:7.3。
さらに遺伝子は、発現を変化させる調節領域が、その遺伝子の何1,000塩基も上流または下流に存在することがある。これらの領域は転写因子と結合することによって作用し、DNAをループさせて、調節配列︵および結合した転写因子︶がRNAポリメラーゼ結合部位に近づくようにする[61]。たとえば、エンハンサーはアクチベータータンパク質と結合することで転写を増加させ、RNAポリメラーゼをプロモーターに動員するのを助ける。逆にサイレンサーはリプレッサータンパク質と結合し、DNAがRNAポリメラーゼに利用されにくくする[62]。
タンパク質コード遺伝子から産生される成熟メッセンジャーRNAの両端には、リボソーム、RNA結合タンパク質、マイクロRNA、ターミネーター、開始・終止コドンなどの結合部位を含む非翻訳領域がある[63]。さらに、ほとんどの真核生物のオープンリーディングフレームには、RNAスプライシングという過程で除去される非翻訳イントロンと、一緒に連結されるエクソンが含まれている。最後に、遺伝子転写産物の末端が切断およびポリアデニル化︵CPA︶部位によって規定され、そこで新たに生成されたpre-mRNAが切断され、約200個のアデノシン一リン酸が3'末端に付加される。ポリ(A)テールは成熟mRNAを分解から保護し、翻訳、局在化、および核からの転写物の輸送に影響する役割を持つ。スプライシングと、それに続くポリアデニル化により、タンパク質またはRNA産物をコードする最終的な成熟mRNAが生成する[64]。ヒト遺伝子の位置を決定する一般的な機構は知られているが、これらの細胞プロセスを調節する正確な因子を同定することは、活発な研究分野である。たとえば、3'-UTRにおける既知の配列特徴では、ヒト遺伝子末端の半分しか説明できない[65]。
真核生物の非コード遺伝子の多くは、異なる転写終結機構を持ち、ポリ(A)テールを持たない。
原核生物の遺伝子の多くは、いくつかのタンパク質コード遺伝子が一団として転写されるオペロンに組織化されている[66][67]。オペロン内の遺伝子は、ポリシストロン型mRNAと呼ばれる連続したメッセンジャーRNAとして転写される。この文脈でのシストロンという用語は遺伝子に相当する。オペロンのmRNAへの転写は、多くの場合、特定の代謝産物の存在によって活性化または不活性するリプレッサーによって制御される[68]。リプレッサーが活性化すると、オペレーター領域と呼ばれるオペロンの先頭にあるDNA配列に結合し、オペロンの転写を抑制する。逆にリプレッサーが不活性化した場合、オペロンの転写を起こすことができるようになる︵たとえば、lacオペロンを参照︶。オペロン遺伝子の産物は通常、関連した機能を持ち、同じ遺伝子制御ネットワークに関与している[53]:7.3。
複雑性[編集]
多くの遺伝子は単純な構造を持っているが、生物学の多くと同様に、非常に複雑な構造をしていたり、珍しい極端な例を示すものもある。真核生物の遺伝子は、しばしばエクソンよりもはるかに大きなイントロンを持ち[69][70]、これらのイントロンは他の遺伝子を内包することさえある[71]。関連するエンハンサーは、何1,000塩基も離れていたり、あるいは、2つの染色体間の物理的接触を介して作用する、まったく別の染色体上に存在することさえある[72][73]。一つの遺伝子が選択的スプライシングによって複数の異なる機能的産物をコードすることもできるし、逆に一つの遺伝子が染色体間をまたがって分割しても、それらの転写産物はトランススプライシングによって再び機能的配列に連結される[74]。また、オーバーラップ遺伝子が、DNA配列の一部を、反対側の鎖上または同じ鎖上︵異なるリーディングフレーム、あるいは同じリーディングフレーム︶で共有することも可能である[75]。遺伝子発現[編集]
詳細は「遺伝子発現」を参照
すべての生物において、遺伝子のDNAにコードされた情報を読み取り、それによって規定されるタンパク質を生成するためには、2つの段階を経る必要がある。まず、遺伝子のDNAがメッセンジャーRNA︵mRNA︶に転写される[53]:6.1。次に、そのmRNAがタンパク質に翻訳される[53]:6.2。RNAコード遺伝子の場合は、最初の段階を経なくてはならないが、タンパク質には翻訳されない[76]。RNAやタンパク質のように、生物学的に機能する分子を産生する過程は遺伝子発現と呼ばれ、その結果として生じる分子は遺伝子産物と呼ばれる。

一連の3塩基コドンが見える一本鎖RNA分子の模式図。それぞれの3 塩基コドンは、タンパク質に翻訳されるときの1つのアミノ酸に対応する。
遺伝子のDNAのヌクレオチド配列は、遺伝暗号を通じてタンパク質のアミノ酸配列を規定する。コドンと呼ばれる三連のヌクレオチドの集合は、それぞれが特定のアミノ酸に対応している[53]:6。DNAの3つの連続した塩基が各アミノ酸をコードするという原理は、1961年にバクテリオファージT4のrIIB遺伝子のフレームシフト変異を使用して実証された[77]︵クリック-ブレナーらの実験を参照︶。
さらに、1つの﹁開始コドン﹂と3種類の﹁終止コドン﹂がタンパク質コード領域の開始と終了を示す。64種類のコドンが可能で︵3つの位置に4種のヌクレオチドで43通りの組合せが可能︶、標準的なアミノ酸は20種類しかない。したがってコードは冗長であり、複数のコドンが同じアミノ酸を指定することができる。コドンとアミノ酸の対応は、既知のすべての生物においてほぼ共通である[78]。

タンパク質コード遺伝子は中間体のmRNAに転写され、その後、機能 的なタンパク質に翻訳される。RNAコード遺伝子は、機能的な非コードRNAに転写される。PDB: 3BSE, 1OBB, 3TRA
翻訳は、成熟mRNAを鋳型として、新しいタンパク質を合成する過程である[53]:6.2。翻訳は、RNAとタンパク質からなる大きな複合体であるリボソームによって行われ、これはペプチド結合を形成することで、伸長するポリペプチド鎖に新しいアミノ酸を付加する化学反応を行う役割を担っている。遺伝暗号は、トランスファーRNA︵tRNA︶と呼ばれる特殊なRNA分子との相互作用を通じて、コドンと呼ばれる単位で一度に3塩基ずつ読み取られる。それぞれのtRNAは、mRNA上で読み取られるコドンと相補的なアンチコドンという3つの不対塩基を持っている。また、tRNAは相補的コドンによって指定されるアミノ酸とも共有結合している。tRNAがmRNA鎖の相補的コドンに結合すると、リボソームはそのアミノ酸を新しいポリペプチド鎖に結合させ、ポリペプチド鎖がアミノ末端からカルボキシル末端まで合成される。合成中および合成後も、ほとんどの新しいタンパク質はその細胞機能を発揮する前に、活性を持った三次元構造に折り畳まれなければならない[53]:3。
遺伝暗号[編集]

転写[編集]
転写により、メッセンジャーRNAと呼ばれる一本鎖RNA分子が生成する。そのヌクレオチド配列は、転写元のDNAと相補的である[53]:6.1。mRNAは、DNA遺伝子とその最終産物であるタンパク質との中間体として機能する。遺伝子のDNAを鋳型として相補的なmRNAが生成される。mRNAは、鋳型鎖と相補的に合成されるため、遺伝子のDNAコード鎖の配列と一致する。転写はRNAポリメラーゼと呼ばれる酵素によって行われ、鋳型鎖を3'から5'の方向に読み取り、RNAを5'から3'の方向に合成する。転写を開始するために、ポリメラーゼがまず遺伝子のプロモーター領域を認識して結合する。したがって遺伝子調節の主要な機構は、ポリメラーゼを物理的に遮断するリプレッサー分子による強固な結合か、プロモーター領域に接近できないようにDNAを組織化することにより、プロモーター領域を遮断または隔離することである[53]:7。 原核生物では、転写は細胞質で行われる。非常に長い転写産物の場合、RNAの3'末端が転写されている間に、RNAの5'末端から翻訳が始まることがある。真核生物では、転写は細胞のDNAが保存されている核で行われる。ポリメラーゼによって生成されたRNA分子は一次転写産物と呼ばれ、翻訳のために細胞質に輸送される前に転写後修飾を受ける。こうした修飾の一つに、転写領域にあるタンパク質をコードしない配列であるイントロンのスプライシング︵除去︶がある。選択的スプライシング機構により、同じ遺伝子から異なる配列を持つ︵したがって異なるタンパク質をコードする︶成熟転写産物が生成することもある。これは真核細胞における調節の主要な形態であり、一部の原核生物でも見られる[53]:7.5[79]。翻訳[編集]

調節[編集]
遺伝子の発現は限られた資源を利用するので、その産物が必要な時だけ発現が行われるように調節されている[53]:7。細胞は、その外部環境︵利用可能な栄養素、温度、その他のストレス︶、その内部環境︵細胞分裂周期、代謝、感染状態など︶、および多細胞生物であればその特定の役割に応じて遺伝子発現を調節する。遺伝子発現は、転写の開始からRNAプロセシング、タンパク質の翻訳後修飾に至るまで、あらゆる段階で制御することができる。大腸菌における乳糖代謝遺伝子の調節︵lacオペロン︶は、1961年に初めて報告されたそうした機構である[80]。RNA遺伝子[編集]
典型的なタンパク質コード遺伝子は、最終タンパク質産物の生成過程における中間体として、まずRNAに転写される[53]:6.1。また、リボソームRNAやトランスファーRNAの合成のように、RNA分子が実際の機能産物となる場合もある。リボザイムとして知られるRNAの中には酵素機能を持つものもあれば、マイクロRNAやリボスイッチのように制御的な役割を持つものもある。このようなRNAが転写されるDNA配列は、非コードRNA遺伝子として知られている[76]。 ウイルスの中には、ゲノム全体をRNAの形で保存し、DNAをまったく含まないものもある[81][82]。遺伝子の保存にRNAを使用するため、宿主細胞は感染するとすぐに、転写を待つことなくタンパク質を合成することができる[83]。対照的に、HIVのようなRNAレトロウイルスは、タンパク質が合成される前に、ゲノムをRNAからDNAに逆転写する必要がある。遺伝[編集]

生物は両親から遺伝子を受け継ぐ。無性生物は、親のゲノムの完全な写しを継承するだけである。有性生物は、それぞれの親から完全な染色体を1セットずつ継承するので、各染色体の写しを2つ持っている[53]:1。
メンデル遺伝[編集]
メンデル遺伝によれば、生物の表現型︵観察可能な身体的/行動的特徴︶の変異は、遺伝型︵特定の遺伝子の集合︶の変異に部分的に起因している。各遺伝子は特定の表現型形質を規定し、遺伝子の異なる配列︵アレル︶が異なる表現型を引き起こす。ほとんどの真核生物︵メンデルが研究したエンドウ豆の植物など︶は、それぞれの形質ごとに2つのアレルを持ち、それぞれの親から1つずつ受け継がれる[53]:20。 座位︵遺伝子座とも︶におけるアレルには顕性アレルと潜性アレルがあり、顕性アレルは同じ形質について他のどのアレルとも対になっても対応する表現型を生じるが、潜性アレルは同じアレルと対になったときにのみ表現型を生じる。もし生物の遺伝型がわかっていれば、どのアレルが顕性で、どのアレルが潜性かを決定することができる。たとえば、エンドウの高い背丈を規定するアレルが、低い背丈を規定するアレルより顕性であれば、一方の親から背丈の高いアレルを、他方の親から背丈の低いアレルを受け継いだエンドウも高い背丈を持つことになる。メンデルの研究は、配偶子︵生殖細胞︶の生成においてアレルが独立に組み入れられて、次世代における変異が保証されることを示した。メンデル遺伝は、単一遺伝子によって決定される多くの形質︵よく知られた遺伝性疾患の多くを含む︶に対して、依然として優れたモデルであるが、DNA複製と細胞分裂の物理的過程は含まれていない[84][85]。DNA複製と細胞分裂[編集]
生物の成長、発達、繁殖は細胞分裂に依存している。細胞分裂とは、一つの細胞が︵通常は同等の︶二つの娘細胞に分裂する過程である。そのためにはまず、DNA複製と呼ばれる過程で、ゲノム中のすべての遺伝子の写しを作る必要がある[53]:5.2。複製はDNAポリメラーゼと呼ばれる特殊な酵素によって行われ、二重らせんDNAの一方の鎖︵鋳型鎖という︶を﹁読み取り﹂、新しい相補鎖を合成する。DNAの二重らせんは塩基対を形成して相互に結合されているため、一方の鎖の塩基配列はその相補鎖の塩基配列を明確に規定する。したがって、正確な写しを作るためには、一方の鎖だけを酵素が読み取ればよい。DNA複製の過程は半保存的であり、各娘細胞に受け継がれるゲノムの2本のDNA鎖には、元のDNA鎖と新しく合成されたDNA鎖が1つずつ含まれている[53]:5.2。 生きた細胞におけるDNA複製の速度は、ファージに感染した大腸菌におけるファージT4 DNAの伸長速度で初めて測定され、驚くほど速いことがわかった[86]。37 °Cでの指数関数的なDNA増加期間において、伸長速度は749ヌクレオチド/秒であった。 DNA複製が完了すると、細胞はゲノムの2つの写しを物理的に分離し、2つの異なる膜結合した細胞に分裂しなければならない[53]:18.2。原核生物︵細菌と古細菌︶では、これは通常、二分裂と呼ばれる比較的単純な過程で行われる。二分裂では、それぞれの環状ゲノムが細胞膜に付着し、細胞膜が陥入して細胞質を2つの膜結合部分に分割するときに娘細胞に分離される。二分裂は、真核生物の細胞分裂の速度と比べると極めて速い。真核生物の細胞分裂は、細胞周期として知られるより複雑な過程を経て行われる。DNAの複製は、細胞周期のS期という段階で起こり、染色体の分離と細胞質の分裂はM期で起こる[53]:18.1。分子遺伝[編集]
ある世代の細胞から次の世代の細胞への遺伝物質の複製と伝達は、分子遺伝の基礎であり、遺伝子の古典的な像と分子的な像との接点でもある。生物が両親の特徴を受け継ぐのは、子孫の細胞には両親のそれぞれの細胞に由来する遺伝子の写しが含まれているからである。無性生殖をする生物では、子孫は親の遺伝的複製あるいはクローンとなる。有性生殖をする生物では、減数分裂と呼ばれる特殊な細胞分裂によって、配偶子または生殖細胞と呼ばれる単数体、すなわち各遺伝子の写しを1つだけ含む細胞が作られる[53]:20.2。雌性︵しせい︶配偶子は卵または卵子と呼ばれ、雄性︵ゆうせい︶配偶子は精細胞または精子と呼ばれる。2つの配偶子が融合して二倍体の受精卵が形成される。受精卵は、2組の遺伝子を持つ単一の細胞であり、それぞれの遺伝子の写しは母親から1つ、父親から1つである[53]:20。 減数分裂の細胞分裂の過程で、遺伝的組換え、あるいは乗換えと呼ばれる現象が起こることがある。この現象では、一方の染色分体上のDNA断片が、対応する相同な非姉妹染色分体上のDNA断片と入れ替わる。この結果、アレルが再集合する可能性がある[53]:5.5。メンデルの独立組合せの法則は、両親の各形質に関する2つの遺伝子が、それぞれ独立して配偶子に組み入れられるというもので、ある形質について生物がどのアレルを受け継ぐかは、別の形質についてどのアレルを受け継ぐかとは無関係である。実際には、このことは、同じ染色体上に存在しない遺伝子、あるいは同じ染色体上で互いに非常に離れた位置にある遺伝子にのみ当てはまる。2つの遺伝子が同じ染色体上で近くに存在するほど、配偶子においてより密接に関連し、一緒に現れる頻度も高くなる︵遺伝的連鎖と呼ばれる︶[87]。互いに非常に近い遺伝子は、その間に交叉︵こうさ︶点が生じる可能性が極めて低いため、基本的に分離することはない[87]。分子進化[編集]
詳細は「分子進化」を参照
変異[編集]
DNAの複製は、ほとんどの場合非常に正確であるが、変異︵突然変異とも︶と呼ばれるエラーが起こることもある[53]:7.6。真核細胞におけるエラー率は、1回の複製につきヌクレオチドあたり10−8と低いこともあるが[88][89]、一部のRNAウイルスでは10−3と高いことがある[90]。このことは、ヒトゲノムの場合、世代ごとに約30個の新しい変異が蓄積されることを意味する[91]。小さな変異は、DNA複製やDNA損傷の結果として引き起こされる場合があり、単一の塩基が変更される点変異や、単一の塩基が挿入または欠失するフレームシフト変異が含まれる。これらの変異はいずれも、ミスセンス︵コドンを変更して別のアミノ酸をコードする︶またはナンセンス︵途中で終止コドンが生じる︶によって遺伝子を変化させる場合がある[92]。より大きな変異は、組換えエラーによって引き起こされる可能性があり、染色体の大きな部分の重複、欠失、再配列または逆位などの染色体異常が生じる。さらに、DNA修復機構が分子の物理的損傷を修復する際に変異エラーを引き起こすこともある。生存という観点においては、変異があっても修復する方が︵たとえば二本鎖切断の修復︶、正確な写しを復元するよりも重要である[53]:5.4。
ある種の集団において、複数の異なる遺伝子のアレルが存在する場合、その遺伝子は多型と呼ばれる。ほとんどの異なるアレルは機能的に同等であるが、アレルの中には異なる表現型形質を引き起こすこともある。遺伝子の最も一般的なアレルは野生型と呼ばれ、稀なアレルは変異体と呼ばれる。集団内における異なるアレル頻度の遺伝的変異は、自然選択と遺伝的浮動の両方によるものである[93]。野生型アレルは、必ずしも稀なアレルの祖先であるとは限らないし、より適応しているとも限らない。
遺伝子内の変異のほとんどは中立で、生物の表現型に影響を与えない︵サイレント変異︶。複数のコドンが同じアミノ酸をコードするため、アミノ酸配列が変化しない変異もある︵同義置換︶。その他の変異には中立的なものもあり、たとえば保存的置換は、アミノ酸配列が変化しても、タンパク質が新しいアミノ酸でも同様に機能する。しかし、多くの変異は有害または致死的であり、自然選択によって集団から取り除かれる。遺伝性疾患は有害突然変異の結果であり、罹患者︵りかんじゃ︶の自然変異が原因で起こることもあれば、遺伝することもある。最後に、ごく一部の変異は生物の適応を向上させる有益なものであり、その定方向選択が適応進化をもたらすため、進化にとって極めて重要である[53]:7.6。
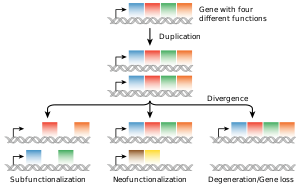
重複遺伝子の3つの進化的運命
真核生物の系統において、新しい遺伝子が生まれる最も一般的な原因は、ゲノム上に存在する遺伝子のコピー数に変異が起こる遺伝子重複である[100][101]。この結果として生じる遺伝子︵パラログ︶は、配列や機能が分岐する可能性がある。このようにして形成された遺伝子の集合が遺伝子ファミリーを構成する。ファミリー内での遺伝子の重複や消失は一般的で、進化における生物多様性の重要な要因となっている[102]。遺伝子重複の結果、ある遺伝子の複製が機能しなくなることや、機能する複製が変異を受けて機能を失うこともある。このような機能しない遺伝子は偽遺伝子と呼ばれる[53]:7.6。
オーファン遺伝子は、その配列が既存の遺伝子と類似性を示さないもので、遺伝子重複よりも稀である。ヒトゲノムには、ヒト以外の生物で同定可能な相同遺伝子を持たない遺伝子が18個-60個含まれていると推定されている[103][104]。オーファン遺伝子は主に、以前は非コード配列であったものから出現したde novo遺伝子か、あるいは遺伝子重複の後に急速に配列が変化して元の関係が検出できなくなったもののいずれかとして生じる[105]。de novo遺伝子は一般的に、ほとんどの真核生物遺伝子よりも短く単純な構造をしており、イントロンはあったとしてもほとんどない[100]。長い進化の過程においては、de novo遺伝子の誕生が、分類学的に制限された遺伝子ファミリーのかなりの部分に関与している可能性がある[106]。
遺伝子の水平伝播とは、生殖以外の機構による遺伝物質の伝達を指す。この機構は、原核生物において新しい遺伝子が生まれる一般的な原因であり、遺伝子重複よりも遺伝的変異により多く寄与していると考えられることもある[107]。これは、抗生物質耐性、病原性、適応的代謝機能を広めるための一般的な手段である[57][108]。遺伝子の水平伝播は真核生物では稀であるが、原生生物や藻類のゲノムでは、細菌の遺伝子が含まれている可能性が高い例が同定されている[109][110]。

代表的な植物 (緑色)、脊椎動物 (青色)、無脊椎動物 (オレン ジ色)、真菌 (黄色)、細菌 (紫色)、およびウイルス (灰色) の遺伝子数を面積で比較した。右の挿入図は小さなゲノムを100倍に拡大した。[113][114][115][116][117][118][119][120]
ゲノムの大きさと、それがコードする遺伝子の数は、生物によって大きく異なる。最も小さなゲノムはウイルス[121]やウイロイド[122]︵単一の非コードRNA遺伝子として機能する︶に見られる。対照的に、植物は非常に大きなゲノムを持つことがあり[123]、イネは46,000を超えるタンパク質コード遺伝子を含んでいる[117]。タンパク質コード遺伝子の総数︵地球上のプロテオーム︶は500万配列と推定されている[124]。
ヒトゲノムを構成するDNA塩基対の数は1950年代から知られていたが、遺伝子の定義や検出方法の改良に伴い、遺伝子の推定数は時代とともに変化してきた。1960年代から1970年代にかけてのヒト遺伝子の数に関する初期の理論的予測は、変異負荷量の推定値とmRNAの数に基づいており、タンパク質コード遺伝子の数を約30,000個と推定していた[125][126][127]。1990年代には、最大で100,000個の遺伝子が推定され、mRNA︵発現配列タグ︶の検出に関する初期のデータは、1980年代に教科書に記述されていた従来の値である30,000遺伝子よりも多いことを示唆していた[128]。
ヒトゲノム配列の初期草案は、タンパク質コード遺伝子が約30,000個という初期の予測を裏付けたが、進行中のGENCODEアノテーション・プロジェクトによる予測では約19,000個に減少した︵2014年︶[129][130]。非コード遺伝子の数は確実には分かっていないが、Ensemblプロジェクトによる推定では、26,462個の非コード遺伝子が存在すると示唆されている︵2024年︶[131]。
配列相同性[編集]
遺伝子間の関係は、DNAの配列を比較することによって測定することができる。類似性の程度が最低線を上回れば、その遺伝子は共通の祖先の子孫であると結論づけることができる。それらは相同であるという[94][95]。共通の祖先からの直接の子孫によって関連している遺伝子はオルソロガス遺伝子であり、それらは通常、異なる種の同じ座位に見いだされる。遺伝子重複の結果として関連する遺伝子はパラロガス遺伝子と呼ばれる[96][97]。 オルソロガス遺伝子の機能は、パラロガス遺伝子よりも類似しているとしばしば考えられるが、その違いはわずかである[98][99]。新しい遺伝子の起源[編集]

ゲノム[編集]
ゲノムは生物の遺伝物質の全体であり、遺伝子と非コード配列の両方を含む[111]。真核生物の遺伝子の構造や機能を分析するために、アノテーション︵注釈︶をするFINDERなどのツールが使用される[112]。遺伝子数[編集]

必須遺伝子[編集]
詳細は「必須遺伝子」を参照
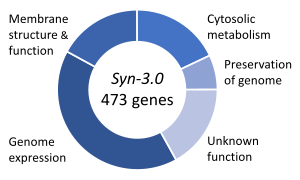
必須遺伝子とは、生物の生存に不可欠であると考えられる遺伝子のことである[133]。この定義は、すべての関連する栄養素が豊富に利用可能で、環境ストレスがないことを前提としている。生物の遺伝子のごく一部だけが必須である。細菌において、大腸菌︵Escherichia coli︶と枯草菌︵Bacillus subtilis︶に必須な遺伝子は250-400個と推定され、これはその生物の遺伝子の10%未満である[134][135][136]。これらの遺伝子の半分は両方の生物のオルソログ︵相同︶であり、タンパク質合成に大きく関与している[136]。出芽酵母︵Saccharomyces cerevisiae︶では、必須遺伝子の数はわずかに多く、1,000遺伝子︵生物の遺伝子の約20%︶である[137]。高等真核生物でその数を測定するのはさらに難しいが、マウスとヒトは約2,000の必須遺伝子があると推定されている︵生物の遺伝子の10%程度︶[138]。合成生物であるSyn 3は、473の必須遺伝子と準必須遺伝子︵急速な成長に必要︶からなる最小限ゲノムを持っているが、そのうち149は機能不明である[132]。
必須遺伝子には、ハウスキーピング遺伝子︵基本的な細胞機能に不可欠[139]︶や、生物の発生やライフサイクルのさまざまな時期に発現する遺伝子も含まれている[140]。ハウスキーピング遺伝子は、比較的一定のレベルで構成的に発現するため、遺伝子発現を分析する際の対照群として使用される。
遺伝子およびゲノムの命名法[編集]
遺伝子命名法は、ヒト遺伝子解析機構の委員会であるHUGO遺伝子命名法委員会︵HGNC︶によって、既知のヒト遺伝子ごとに、承認された遺伝子名とシンボル︵短縮形略語︶の形式で確立されており、HGNCが管理するデータベースを通じてアクセスすることができる。シンボルは一意であるように選ばれ、各遺伝子は1つのシンボルしか持たない︵ただし承認されたシンボルが変更されることもある︶。シンボルは遺伝子ファミリーの他のメンバーや、他の生物種︵特にマウスは共通のモデル生物としての役割を担うため︶の相同遺伝子と一貫性を保つことが好ましい[141]。遺伝子工学[編集]
詳細は「遺伝子工学」を参照
遺伝子工学とは、バイオテクノロジーによって生物のゲノムを改変することである。1970年代以来、生物の遺伝子を特異的に追加、削除、編集するためのさまざまな技術が開発されてきた[142]。さらに最近開発されたゲノム工学技術は、人工ヌクレアーゼ酵素を使用して、染色体に標的DNA修復を作成し、切断が修復されるときに遺伝子を破壊または編集するものである[143][144][145][146]。関連用語の合成生物学は、生物の広範な遺伝子工学を指すために使用されることがある[147]。
遺伝子工学は今では、モデル生物を用いた日常的な研究ツールとなっている。たとえば、細菌に遺伝子を付加することは容易で[148]、特定の遺伝子の機能が破壊されたノックアウトマウスの系統がその遺伝子の研究に使用されている[149][150]。農業、産業用バイオテクノロジー、医学への応用のために、多くの生物が遺伝子組換えされてきた。
多細胞生物の場合、通常は胚が操作されて、遺伝子組換え成体へと成長する[151]。けれども、遺伝子疾患を治療するために、遺伝子治療技術を用いて成体生物の細胞のゲノムを編集することもできる。
関連項目[編集]
- エピジェネティクス - DNA塩基配列の変化を伴わない遺伝的形質や細胞機能の安定した変化を研究する学問領域
- 全ゲノム配列 - 生物のゲノムのDNA配列のすべて(またはほぼすべて)を一度に決定するプロセス
- 遺伝子中心の進化観 - 利己的遺伝子の理論
- 遺伝子量 - ゲノム中に存在する特定の遺伝子のコピー数
- 生物学的特許 - 生物学分野の発明に関する特許
- 遺伝子プール - 集団に含まれるすべての遺伝子の集合
- 遺伝子重剰性 - ゲノム上に同じ機能を果たす複数の遺伝子が存在すること
- 遺伝子サイレンシング - 細胞内で遺伝子の発現を制御/阻止すること
- 遺伝的アルゴリズム - コンピュータサイエンス分野で自然選択から着想を得た解の探索手法
- ハプロタイプ - 単一の親から一緒に受け継がれるアレル群
- 遺伝子予測ソフトウェアの一覧
- ヒト遺伝子の一覧
- 予測医学 - 疾病の発生確率を予測し、予防あるいは影響の軽減措置を講じる医学分野
- 量的形質座位 - 生物集団の表現型形質における量的形質の変異と相関する座位
- 利己的遺伝因子 - ゲノム内の他の遺伝子を犠牲にして自らの伝達を強化できる遺伝子断片
脚注[編集]
出典[編集]
(一)^ abcOrgogozo V, Peluffo AE, Morizot B (2016). “The "Mendelian Gene" and the "Molecular Gene": Two Relevant Concepts of Genetic Units”. Current Topics in Developmental Biology 119: 1–26. doi:10.1016/bs.ctdb.2016.03.002. PMID 27282022.
(二)^ “What is a gene?: MedlinePlus Genetics”. MedlinePlus (2020年9月17日). 2021年1月4日閲覧。
(三)^ Hirsch ED (2002). The new dictionary of cultural literacy. Boston: Houghton Mifflin. ISBN 0-618-22647-8. OCLC 50166721
(四)^ “Studying Genes”. nigms.nih.gov. 2021年1月15日閲覧。
(五)^ Elston RC, Satagopan JM, Sun S (2012). “Genetic terminology”. Statistical Human Genetics. Methods in Molecular Biology. 850. Humana Press. pp. 1–9. doi:10.1007/978-1-61779-555-8_1. ISBN 978-1-61779-554-1. PMC 4450815. PMID 22307690
(六)^ abJohannsen W (1909) (German). Elemente der exakten Erblichkeitslehre [Elements of the exact theory of heredity]. Jena, Germany: Gustav Fischer. p. 124 From p. 124: "Dieses "etwas" in den Gameten bezw. in der Zygote, … – kurz, was wir eben Gene nennen wollen – bedingt sind." (This "something" in the gametes or in the zygote, which has crucial importance for the character of the organism, is usually called by the quite ambiguous term Anlagen [primordium, from the German word Anlage for "plan, arrangement ; rough sketch"]. Many other terms have been suggested, mostly unfortunately in closer connection with certain hypothetical opinions. The word "pangene", which was introduced by Darwin, is perhaps used most frequently in place of Anlagen. However, the word "pangene" was not well chosen, as it is a compound word containing the roots pan (the neuter form of Πας all, every) and gen (from γί-γ(ε)ν-ομαι, to become). Only the meaning of this latter [i.e., gen] comes into consideration here ; just the basic idea – [namely,] that a trait in the developing organism can be determined or is influenced by "something" in the gametes – should find expression. No hypothesis about the nature of this "something" should be postulated or supported by it. For that reason it seems simplest to use in isolation the last syllable gen from Darwin's well-known word, which alone is of interest to us, in order to replace, with it, the poor, ambiguous word Anlage. Thus we will say simply "gene" and "genes" for "pangene" and "pangenes". The word gene is completely free of any hypothesis ; it expresses only the established fact that in any case many traits of the organism are determined by specific, separable, and thus independent "conditions", "foundations", "plans" – in short, precisely what we want to call genes.)
(七)^ “1909: The Word Gene Coined”. genome.gov. 2021年3月8日閲覧。 "...Wilhelm Johannsen coined the word gene to describe the Mendelian units of heredity..."
(八)^ Roth SC (July 2019). “What is genomic medicine?”. Journal of the Medical Library Association (University Library System, University of Pittsburgh) 107 (3): 442–448. doi:10.5195/jmla.2019.604. PMC 6579593. PMID 31258451.
(九)^ abcdKampourakis K (2017). Making Sense of Genes. Cambridge, UK: Cambridge University Press
(十)^ Gericke N, Hagberg M (5 December 2006). “Definition of historical models of gene function and their relation to students' understanding of genetics”. Science & Education 16 (7–8): 849–881. Bibcode: 2007Sc&Ed..16..849G. doi:10.1007/s11191-006-9064-4.
(11)^ Meunier R (2022年). “Stanford Encyclopedia of Philosophy: Gene”. Stanford Encyclopedia of Philosophy. 2023年2月28日閲覧。
(12)^ Kellis M, Wold B, Snyder MP, Bernstein BE, Kundaje A, Marinov GK, Ward LD, Birney E, Crawford GE, Dekker J, Dunham I, Elnitski LL, Farnham PJ, Feingold EA, Gerstein M, Giddings MC, Gilbert DM, Gingeras TR, Green ED, Guigo R, Hubbard T, Kent J, Lieb JD, Myers RM, Pazin MJ, Ren B, Stamatoyannopoulos JA, Weng Z, White KP, Hardison RC (April 2014). “Defining functional DNA elements in the human genome”. Proceedings of the National Academy of Sciences of the United States of America 111 (17): 6131–8. Bibcode: 2014PNAS..111.6131K. doi:10.1073/pnas.1318948111. PMC 4035993. PMID 24753594.
(13)^ abドーキンス,リチャード 著、日高敏隆/岸由二/羽田節子/垂水雄二 訳﹃利己的な遺伝子40周年記念版﹄紀伊國屋書店、2018年2月15日。ISBN 978-4314011532。
(14)^ Stoltz K, Griffiths P (2004). “Genes: Philosophical Analyses Put to the Test”. History and Philosophy of the Life Sciences 26 (1): 5–28. doi:10.1080/03919710412331341621. JSTOR 23333378. PMID 15791804.
(15)^ Beadle GW, Tatum EL (November 1941). “Genetic Control of Biochemical Reactions in Neurospora”. Proceedings of the National Academy of Sciences of the United States of America 27 (11): 499–506. Bibcode: 1941PNAS...27..499B. doi:10.1073/pnas.27.11.499. PMC 1078370. PMID 16588492.
(16)^ Horowitz NH, Berg P, Singer M, Lederberg J, Susman M, Doebley J, Crow JF (January 2004). “A centennial: George W. Beadle, 1903-1989”. Genetics 166 (1): 1–10. doi:10.1534/genetics.166.1.1. PMC 1470705. PMID 15020400.
(17)^ Judson HF (1996). The Eight Day of Creation (Expanded ed.). Plainview, NY (US): Cold Spring Harbor Laboratory Press
(18)^ abWatson JD (1965). Molecular Biology of the Gene. New York, NY, US: W.A. Benjamin, Inc.
(19)^ Alberts B, Bray D, Lewis J, Raff M, Roberts K, Watson JD (1994). Molecular Biology of the Cell: Third Edition. London, UK: Garland Publishing, Inc.. ISBN 0-8153-1619-4
(20)^ Moran LA, Horton HR, Scrimgeour KG, Perry MD (2012). Principles of Biochemistry: Fifth Edition. Upper Saddle River, NJ, US: Pearson
(21)^ Lewin B (2004). Genes VIII. Upper Saddle River, NJ, US: Pearson/Prentice Hall
(22)^ Piovesan A, Pelleri MC, Antonaros F, Strippoli P, Caracausi M, and Vitale L (2019). “On the length, weight and GC content of the human genome”. BMC Research Notes 12 (1): 106–173. doi:10.1186/s13104-019-4137-z. PMC 6391780. PMID 30813969.
(23)^ Hubé F, and Francastel C (2015). “Mammalian Introns: When the Junk Generates Molecular Diversity”. International Journal of Molecular Sciences 16 (3): 4429–4452. doi:10.3390/ijms16034429. PMC 4394429. PMID 25710723.
(24)^ Francis WR, and Wörheide G (2017). “Similar ratios of introns to intergenic sequence across animal genomes”. Genome Biology and Evolution 9 (6): 1582–1598. doi:10.1093/gbe/evx103. PMC 5534336. PMID 28633296.
(25)^ Mortola E, Long M (2021). “Turning Junk into Us: How Genes Are Born”. American Scientist 109: 174–182.
(26)^ Hopkin K (2009). “The Evolving Definition of a Gene: With the discovery that nearly all of the genome is transcribed, the definition of a "gene" needs another revision”. BioScience 59: 928–931. doi:10.1525/bio.2009.59.11.3.
(27)^ Pearson H (2006). “What Is a Gene?”. Nature 441 (7092): 399–401. Bibcode: 2006Natur.441..398P. doi:10.1038/441398a. PMID 16724031.
(28)^ Pennisi E (2007). “DNA study forces rethink of what it means to be a gene”. Science 316 (5831): 1556–1557. doi:10.1126/science.316.5831.1556. PMID 17569836.
(29)^ Wolf YI, Kazlauskas D, Iranzo J, Lucía-Sanz A, Kuhn JH, Krupovic M, Dolja VV, Koonin EV (November 2018). Eric Delwart, Luis Enjuanes. “Origins and Evolution of the Global RNA Virome”. mBio 9 (6): e02329–18. doi:10.1128/mBio.02329-18. PMC 6282212. PMID 30482837.
(30)^ abPennisi E (June 2007). “Genomics. DNA study forces rethink of what it means to be a gene”. Science 316 (5831): 1556–7. doi:10.1126/science.316.5831.1556. PMID 17569836.
(31)^ Marande W, Burger G (October 2007). “Mitochondrial DNA as a genomic jigsaw puzzle”. Science (AAAS) 318 (5849): 415. Bibcode: 2007Sci...318..415M. doi:10.1126/science.1148033. PMID 17947575.
(32)^ Parra G, Reymond A, Dabbouseh N, Dermitzakis ET, Castelo R, Thomson TM, Antonarakis SE, Guigó R (January 2006). “Tandem chimerism as a means to increase protein complexity in the human genome”. Genome Research 16 (1): 37–44. doi:10.1101/gr.4145906. PMC 1356127. PMID 16344564.
(33)^ abcGerstein MB, Bruce C, Rozowsky JS, Zheng D, Du J, Korbel JO, Emanuelsson O, Zhang ZD, Weissman S, Snyder M (June 2007). “What is a gene, post-ENCODE? History and updated definition”. Genome Research 17 (6): 669–81. doi:10.1101/gr.6339607. PMID 17567988.
(34)^ Noble D (September 2008). “Genes and causation”. Philosophical Transactions. Series A, Mathematical, Physical, and Engineering Sciences 366 (1878): 3001–15. Bibcode: 2008RSPTA.366.3001N. doi:10.1098/rsta.2008.0086. PMID 18559318.
(35)^ “Blending Inheritance - an overview | ScienceDirect Topics”. 2021年7月3日閲覧。
(36)^ "genesis". Oxford English Dictionary (3rd ed.). Oxford University Press. September 2005. (要購読、またはイギリス公立図書館への会員加入。)
(37)^ Magner LN (2002). A History of the Life Sciences (Third ed.). Marcel Dekker, CRC Press. p. 371. ISBN 978-0-203-91100-6
(38)^ Henig RM (2000). The Monk in the Garden: The Lost and Found Genius of Gregor Mendel, the Father of Genetics. Boston: Houghton Mifflin. pp. 1–9. ISBN 978-0395-97765-1
(39)^ abde Vries H (1889) (ドイツ語). Intracellulare Pangenese [Intracellular Pangenesis]. Jena: Verlag von Gustav Fischer Translated in 1908 from German to English by Open Court Publishing Co., Chicago, 1910
(40)^ Bateson W (1906). “The progress of genetic research”. Report of the Third International Conference 1906 on Genetics. London, England: Royal Horticultural Society. pp. 90–97. "… the science itself [i.e. the study of the breeding and hybridisation of plants] is still nameless, and we can only describe our pursuit by cumbrous and often misleading periphrasis. To meet this difficulty I suggest for the consideration of this Congress the term Genetics, which sufficiently indicates that our labors are devoted to the elucidation of the phenomena of heredity and variation: in other words, to the physiology of Descent, with implied bearing on the theoretical problems of the evolutionist and the systematist, and application to the practical problems of breeders, whether of animals or plants."
(41)^ Avery OT, Macleod CM, McCarty M (February 1944). “Studies on the Chemical Nature of the Substance Inducing Transformation of Pneumococcal Types : Induction of Transformation by a Desoxyribonucleic Acid Fraction Isolated From Pneumococcus Type III”. The Journal of Experimental Medicine 79 (2): 137–58. doi:10.1084/jem.79.2.137. PMC 2135445. PMID 19871359. Reprint: Avery OT, MacLeod CM, McCarty M (February 1979). “Studies on the chemical nature of the substance inducing transformation of pneumococcal types. Inductions of transformation by a desoxyribonucleic acid fraction isolated from pneumococcus type III”. The Journal of Experimental Medicine 149 (2): 297–326. doi:10.1084/jem.149.2.297. PMC 2184805. PMID 33226.
(42)^ Hershey AD, Chase M (May 1952). “Independent functions of viral protein and nucleic acid in growth of bacteriophage”. The Journal of General Physiology 36 (1): 39–56. doi:10.1085/jgp.36.1.39. PMC 2147348. PMID 12981234.
(43)^ Judson H (1979). The Eighth Day of Creation: Makers of the Revolution in Biology. Cold Spring Harbor Laboratory Press. pp. 51–169. ISBN 978-0-87969-477-7
(44)^ Watson JD, Crick FH (April 1953). “Molecular structure of nucleic acids; a structure for deoxyribose nucleic acid”. Nature 171 (4356): 737–8. Bibcode: 1953Natur.171..737W. doi:10.1038/171737a0. PMID 13054692.
(45)^ Benzer S (June 1955). “Fine Structure of a Genetic Region in Bacteriophage”. Proceedings of the National Academy of Sciences of the United States of America 41 (6): 344–54. Bibcode: 1955PNAS...41..344B. doi:10.1073/pnas.41.6.344. PMC 528093. PMID 16589677.
(46)^ Benzer S (November 1959). “On the Topology of the Genetic Fine Structure”. Proceedings of the National Academy of Sciences of the United States of America 45 (11): 1607–20. Bibcode: 1959PNAS...45.1607B. doi:10.1073/pnas.45.11.1607. PMC 222769. PMID 16590553.
(47)^ Min Jou W, Haegeman G, Ysebaert M, Fiers W (May 1972). “Nucleotide sequence of the gene coding for the bacteriophage MS2 coat protein”. Nature 237 (5350): 82–8. Bibcode: 1972Natur.237...82J. doi:10.1038/237082a0. PMID 4555447.
(48)^ Sanger F, Nicklen S, Coulson AR (December 1977). “DNA sequencing with chain-terminating inhibitors”. Proceedings of the National Academy of Sciences of the United States of America 74 (12): 5463–7. Bibcode: 1977PNAS...74.5463S. doi:10.1073/pnas.74.12.5463. PMC 431765. PMID 271968.
(49)^ Adams JU (2008). “DNA Sequencing Technologies”. Nature Education Knowledge. SciTable (Nature Publishing Group) 1 (1): 193.
(50)^ Huxley J (1942). Evolution: the Modern Synthesis. Cambridge, Massachusetts: MIT Press. ISBN 978-0262513661
(51)^ Williams GC (2001). Adaptation and Natural Selection a Critique of Some Current Evolutionary Thought (Online ed.). Princeton: Princeton University Press. ISBN 9781400820108
(52)^ Duret L (2008). “Neutral Theory: The Null Hypothesis of Molecular Evolution”. Nature Education 1: 218.
(53)^ abcdefghijklmnopqrstuvwxyzaaabacadaeafagahaiAlberts B, Johnson A, Lewis J, Raff M, Roberts K, Walter P﹃Molecular Biology of the Cell﹄︵Fourth︶Garland Science、New York、2002年、(#主要教科書の章番号を脚注リンク脇の数字 n.m に示す)。ISBN 978-0-8153-3218-3。
(54)^ Stryer L, Berg JM, Tymoczko JL (2002). Biochemistry (5th ed.). San Francisco: W.H. Freeman. ISBN 978-0-7167-4955-4
(55)^ Bolzer A, Kreth G, Solovei I, Koehler D, Saracoglu K, Fauth C, Müller S, Eils R, Cremer C, Speicher MR, Cremer T (May 2005). “Three-dimensional maps of all chromosomes in human male fibroblast nuclei and prometaphase rosettes”. PLOS Biology 3 (5): e157. doi:10.1371/journal.pbio.0030157. PMC 1084335. PMID 15839726.  (56)^ Braig M, Schmitt CA (March 2006). “Oncogene-induced senescence: putting the brakes on tumor development”. Cancer Research 66 (6): 2881–4. doi:10.1158/0008-5472.CAN-05-4006. PMID 16540631.
(57)^ abBennett PM (March 2008). “Plasmid encoded antibiotic resistance: acquisition and transfer of antibiotic resistance genes in bacteria”. British Journal of Pharmacology 153 (Suppl 1): S347-57. doi:10.1038/sj.bjp.0707607. PMC 2268074. PMID 18193080.
(58)^ International Human Genome Sequencing Consortium (October 2004). “Finishing the euchromatic sequence of the human genome”. Nature 431 (7011): 931–45. Bibcode: 2004Natur.431..931H. doi:10.1038/nature03001. PMID 15496913.
(59)^ abShafee, Thomas; Lowe, Rohan (2017). “Eukaryotic and prokaryotic gene structure”. WikiJournal of Medicine 4 (1). doi:10.15347/wjm/2017.002. ISSN 2002-4436.
(60)^ Mortazavi A, Williams BA, McCue K, Schaeffer L, Wold B (July 2008). “Mapping and quantifying mammalian transcriptomes by RNA-Seq”. Nature Methods 5 (7): 621–8. doi:10.1038/nmeth.1226. PMID 18516045.
(61)^ Pennacchio LA, Bickmore W, Dean A, Nobrega MA, Bejerano G (April 2013). “Enhancers: five essential questions”. Nature Reviews. Genetics 14 (4): 288–95. doi:10.1038/nrg3458. PMC 4445073. PMID 23503198.
(62)^ Maston GA, Evans SK, Green MR (2006). “Transcriptional regulatory elements in the human genome”. Annual Review of Genomics and Human Genetics 7: 29–59. doi:10.1146/annurev.genom.7.080505.115623. PMID 16719718.
(63)^ Mignone F, Gissi C, Liuni S, Pesole G (2002-02-28). “Untranslated regions of mRNAs”. Genome Biology 3 (3): REVIEWS0004. doi:10.1186/gb-2002-3-3-reviews0004. PMC 139023. PMID 11897027.
(64)^ Bicknell AA, Cenik C, Chua HN, Roth FP, Moore MJ (December 2012). “Introns in UTRs: why we should stop ignoring them”. BioEssays 34 (12): 1025–34. doi:10.1002/bies.201200073. PMID 23108796.
(65)^ Shkurin A, Pour SE, Hughes TR (April 2023). “Known sequence features explain half of all human gene ends”. NAR Genomics and Bioinformatics 5 (2): lqad031. doi:10.1093/nargab/lqad031. PMC 10072996. PMID 37035540.
(66)^ Salgado H, Moreno-Hagelsieb G, Smith TF, Collado-Vides J (June 2000). “Operons in Escherichia coli: genomic analyses and predictions”. Proceedings of the National Academy of Sciences of the United States of America 97 (12): 6652–7. Bibcode: 2000PNAS...97.6652S. doi:10.1073/pnas.110147297. PMC 18690. PMID 10823905.
(67)^ Blumenthal T (November 2004). “Operons in eukaryotes”. Briefings in Functional Genomics & Proteomics 3 (3): 199–211. doi:10.1093/bfgp/3.3.199. PMID 15642184.
(68)^ Jacob F, Monod J (June 1961). “Genetic regulatory mechanisms in the synthesis of proteins”. Journal of Molecular Biology 3 (3): 318–56. doi:10.1016/S0022-2836(61)80072-7. PMID 13718526.
(69)^ Pozzoli U, Menozzi G, Comi GP, Cagliani R, Bresolin N, Sironi M (January 2007). “Intron size in mammals: complexity comes to terms with economy”. Trends in Genetics 23 (1): 20–24. doi:10.1016/j.tig.2006.10.003. PMID 17070957.
(70)^ Marais G, Nouvellet P, Keightley PD, Charlesworth B (May 2005). “Intron size and exon evolution in Drosophila”. Genetics 170 (1): 481–485. doi:10.1534/genetics.104.037333. PMC 1449718. PMID 15781704.
(71)^ Kumar A (September 2009). “An overview of nested genes in eukaryotic genomes”. Eukaryotic Cell 8 (9): 1321–1329. doi:10.1128/EC.00143-09. PMC 2747821. PMID 19542305..
(72)^ Spilianakis CG, Lalioti MD, Town T, Lee GR, Flavell RA (June 2005). “Interchromosomal associations between alternatively expressed loci”. Nature 435 (7042): 637–645. Bibcode: 2005Natur.435..637S. doi:10.1038/nature03574. PMID 15880101.
(73)^ Williams A, Spilianakis CG, Flavell RA (April 2010). “Interchromosomal association and gene regulation in trans”. Trends in Genetics 26 (4): 188–197. doi:10.1016/j.tig.2010.01.007. PMC 2865229. PMID 20236724.
(74)^ Lei Q, Li C, Zuo Z, Huang C, Cheng H, Zhou R (March 2016). “Evolutionary Insights into RNA trans-Splicing in Vertebrates”. Genome Biology and Evolution 8 (3): 562–577. doi:10.1093/gbe/evw025. PMC 4824033. PMID 26966239.
(75)^ Wright BW, Molloy MP, Jaschke PR (March 2022). “Overlapping genes in natural and engineered genomes”. Nature Reviews. Genetics 23 (3): 154–168. doi:10.1038/s41576-021-00417-w. PMC 8490965. PMID 34611352.
(76)^ abEddy SR (December 2001). “Non-coding RNA genes and the modern RNA world”. Nature Reviews. Genetics 2 (12): 919–29. doi:10.1038/35103511. PMID 11733745.
(77)^ Crick FH, Barnett L, Brenner S, Watts-Tobin RJ (December 1961). “General nature of the genetic code for proteins”. Nature 192 (4809): 1227–32. Bibcode: 1961Natur.192.1227C. doi:10.1038/1921227a0. PMID 13882203.
(78)^ Crick FH (October 1962). “The genetic code”. Scientific American (WH Freeman and Company) 207 (4): 66–74. Bibcode: 1962SciAm.207d..66C. doi:10.1038/scientificamerican1062-66. PMID 13882204.
(79)^ Woodson SA (May 1998). “Ironing out the kinks: splicing and translation in bacteria”. Genes & Development 12 (9): 1243–7. doi:10.1101/gad.12.9.1243. PMID 9573040.
(80)^ Jacob F, Monod J (June 1961). “Genetic regulatory mechanisms in the synthesis of proteins”. Journal of Molecular Biology 3 (3): 318–56. doi:10.1016/S0022-2836(61)80072-7. PMID 13718526.
(81)^ Koonin EV, Dolja VV (January 1993). “Evolution and taxonomy of positive-strand RNA viruses: implications of comparative analysis of amino acid sequences”. Critical Reviews in Biochemistry and Molecular Biology 28 (5): 375–430. doi:10.3109/10409239309078440. PMID 8269709.
(82)^ Domingo E (2001). “RNA Virus Genomes”. eLS. doi:10.1002/9780470015902.a0001488.pub2. ISBN 978-0470016176.
(83)^ Domingo E, Escarmís C, Sevilla N, Moya A, Elena SF, Quer J, Novella IS, Holland JJ (June 1996). “Basic concepts in RNA virus evolution”. FASEB Journal 10 (8): 859–64. doi:10.1096/fasebj.10.8.8666162. PMID 8666162.
(84)^ Miko I (2008). “Gregor Mendel and the Principles of Inheritance”. Nature Education Knowledge. SciTable (Nature Publishing Group) 1 (1): 134.
(85)^ Chial H (2008). “Mendelian Genetics: Patterns of Inheritance and Single-Gene Disorders”. Nature Education Knowledge. SciTable (Nature Publishing Group) 1 (1): 63.
(86)^ McCarthy D, Minner C, Bernstein H, Bernstein C (October 1976). “DNA elongation rates and growing point distributions of wild-type phage T4 and a DNA-delay amber mutant”. Journal of Molecular Biology 106 (4): 963–81. doi:10.1016/0022-2836(76)90346-6. PMID 789903.
(87)^ abLobo I, Shaw K (2008). “Discovery and Types of Genetic Linkage”. Nature Education Knowledge. SciTable (Nature Publishing Group) 1 (1): 139.
(88)^ Nachman MW, Crowell SL (September 2000). “Estimate of the mutation rate per nucleotide in humans”. Genetics 156 (1): 297–304. doi:10.1093/genetics/156.1.297. PMC 1461236. PMID 10978293.
(89)^ Roach JC, Glusman G, Smit AF, Huff CD, Hubley R, Shannon PT, Rowen L, Pant KP, Goodman N, Bamshad M, Shendure J, Drmanac R, Jorde LB, Hood L, Galas DJ (April 2010). “Analysis of genetic inheritance in a family quartet by whole-genome sequencing”. Science 328 (5978): 636–9. Bibcode: 2010Sci...328..636R. doi:10.1126/science.1186802. PMC 3037280. PMID 20220176.
(90)^ Drake JW, Charlesworth B, Charlesworth D, Crow JF (April 1998). “Rates of spontaneous mutation”. Genetics 148 (4): 1667–86. doi:10.1093/genetics/148.4.1667. PMC 1460098. PMID 9560386.
(91)^ Pyeritz, Reed E., Bruce R. Korf, and Wayne W. Grody, eds. Emery and Rimoin’s principles and practice of medical genetics and genomics: foundations. Academic Press, 2018.
(92)^ “What kinds of gene mutations are possible?”. Genetics Home Reference. United States National Library of Medicine (2015年5月11日). 2015年5月19日閲覧。
(93)^ Andrews CA (2010). “Natural Selection, Genetic Drift, and Gene Flow Do Not Act in Isolation in Natural Populations”. Nature Education Knowledge. SciTable (Nature Publishing Group) 3 (10): 5.
(94)^ Patterson C (November 1988). “Homology in classical and molecular biology”. Molecular Biology and Evolution 5 (6): 603–25. doi:10.1093/oxfordjournals.molbev.a040523. PMID 3065587.
(95)^ Graur D (2016). Molecular and Genome Evolution. Sunderland MA (US): Sinauer Associates, Inc.. ISBN 9781605354699
(96)^ Graur D (2016). Molecular and Genome Evolution. Sunderland MA (US): Sinauer Associates, Inc.. ISBN 9781605354699
(97)^ Jensen RA (2001). “Orthologs and paralogs - we need to get it right”. Genome Biology 2 (8): INTERACTIONS1002. doi:10.1186/gb-2001-2-8-interactions1002. PMC 138949. PMID 11532207.
(98)^ Studer RA, Robinson-Rechavi M (May 2009). “How confident can we be that orthologs are similar, but paralogs differ?”. Trends in Genetics 25 (5): 210–6. doi:10.1016/j.tig.2009.03.004. PMID 19368988.
(99)^ Altenhoff AM, Studer RA, Robinson-Rechavi M, Dessimoz C (2012). “Resolving the ortholog conjecture: orthologs tend to be weakly, but significantly, more similar in function than paralogs”. PLOS Computational Biology 8 (5): e1002514. Bibcode: 2012PLSCB...8E2514A. doi:10.1371/journal.pcbi.1002514. PMC 3355068. PMID 22615551.
(56)^ Braig M, Schmitt CA (March 2006). “Oncogene-induced senescence: putting the brakes on tumor development”. Cancer Research 66 (6): 2881–4. doi:10.1158/0008-5472.CAN-05-4006. PMID 16540631.
(57)^ abBennett PM (March 2008). “Plasmid encoded antibiotic resistance: acquisition and transfer of antibiotic resistance genes in bacteria”. British Journal of Pharmacology 153 (Suppl 1): S347-57. doi:10.1038/sj.bjp.0707607. PMC 2268074. PMID 18193080.
(58)^ International Human Genome Sequencing Consortium (October 2004). “Finishing the euchromatic sequence of the human genome”. Nature 431 (7011): 931–45. Bibcode: 2004Natur.431..931H. doi:10.1038/nature03001. PMID 15496913.
(59)^ abShafee, Thomas; Lowe, Rohan (2017). “Eukaryotic and prokaryotic gene structure”. WikiJournal of Medicine 4 (1). doi:10.15347/wjm/2017.002. ISSN 2002-4436.
(60)^ Mortazavi A, Williams BA, McCue K, Schaeffer L, Wold B (July 2008). “Mapping and quantifying mammalian transcriptomes by RNA-Seq”. Nature Methods 5 (7): 621–8. doi:10.1038/nmeth.1226. PMID 18516045.
(61)^ Pennacchio LA, Bickmore W, Dean A, Nobrega MA, Bejerano G (April 2013). “Enhancers: five essential questions”. Nature Reviews. Genetics 14 (4): 288–95. doi:10.1038/nrg3458. PMC 4445073. PMID 23503198.
(62)^ Maston GA, Evans SK, Green MR (2006). “Transcriptional regulatory elements in the human genome”. Annual Review of Genomics and Human Genetics 7: 29–59. doi:10.1146/annurev.genom.7.080505.115623. PMID 16719718.
(63)^ Mignone F, Gissi C, Liuni S, Pesole G (2002-02-28). “Untranslated regions of mRNAs”. Genome Biology 3 (3): REVIEWS0004. doi:10.1186/gb-2002-3-3-reviews0004. PMC 139023. PMID 11897027.
(64)^ Bicknell AA, Cenik C, Chua HN, Roth FP, Moore MJ (December 2012). “Introns in UTRs: why we should stop ignoring them”. BioEssays 34 (12): 1025–34. doi:10.1002/bies.201200073. PMID 23108796.
(65)^ Shkurin A, Pour SE, Hughes TR (April 2023). “Known sequence features explain half of all human gene ends”. NAR Genomics and Bioinformatics 5 (2): lqad031. doi:10.1093/nargab/lqad031. PMC 10072996. PMID 37035540.
(66)^ Salgado H, Moreno-Hagelsieb G, Smith TF, Collado-Vides J (June 2000). “Operons in Escherichia coli: genomic analyses and predictions”. Proceedings of the National Academy of Sciences of the United States of America 97 (12): 6652–7. Bibcode: 2000PNAS...97.6652S. doi:10.1073/pnas.110147297. PMC 18690. PMID 10823905.
(67)^ Blumenthal T (November 2004). “Operons in eukaryotes”. Briefings in Functional Genomics & Proteomics 3 (3): 199–211. doi:10.1093/bfgp/3.3.199. PMID 15642184.
(68)^ Jacob F, Monod J (June 1961). “Genetic regulatory mechanisms in the synthesis of proteins”. Journal of Molecular Biology 3 (3): 318–56. doi:10.1016/S0022-2836(61)80072-7. PMID 13718526.
(69)^ Pozzoli U, Menozzi G, Comi GP, Cagliani R, Bresolin N, Sironi M (January 2007). “Intron size in mammals: complexity comes to terms with economy”. Trends in Genetics 23 (1): 20–24. doi:10.1016/j.tig.2006.10.003. PMID 17070957.
(70)^ Marais G, Nouvellet P, Keightley PD, Charlesworth B (May 2005). “Intron size and exon evolution in Drosophila”. Genetics 170 (1): 481–485. doi:10.1534/genetics.104.037333. PMC 1449718. PMID 15781704.
(71)^ Kumar A (September 2009). “An overview of nested genes in eukaryotic genomes”. Eukaryotic Cell 8 (9): 1321–1329. doi:10.1128/EC.00143-09. PMC 2747821. PMID 19542305..
(72)^ Spilianakis CG, Lalioti MD, Town T, Lee GR, Flavell RA (June 2005). “Interchromosomal associations between alternatively expressed loci”. Nature 435 (7042): 637–645. Bibcode: 2005Natur.435..637S. doi:10.1038/nature03574. PMID 15880101.
(73)^ Williams A, Spilianakis CG, Flavell RA (April 2010). “Interchromosomal association and gene regulation in trans”. Trends in Genetics 26 (4): 188–197. doi:10.1016/j.tig.2010.01.007. PMC 2865229. PMID 20236724.
(74)^ Lei Q, Li C, Zuo Z, Huang C, Cheng H, Zhou R (March 2016). “Evolutionary Insights into RNA trans-Splicing in Vertebrates”. Genome Biology and Evolution 8 (3): 562–577. doi:10.1093/gbe/evw025. PMC 4824033. PMID 26966239.
(75)^ Wright BW, Molloy MP, Jaschke PR (March 2022). “Overlapping genes in natural and engineered genomes”. Nature Reviews. Genetics 23 (3): 154–168. doi:10.1038/s41576-021-00417-w. PMC 8490965. PMID 34611352.
(76)^ abEddy SR (December 2001). “Non-coding RNA genes and the modern RNA world”. Nature Reviews. Genetics 2 (12): 919–29. doi:10.1038/35103511. PMID 11733745.
(77)^ Crick FH, Barnett L, Brenner S, Watts-Tobin RJ (December 1961). “General nature of the genetic code for proteins”. Nature 192 (4809): 1227–32. Bibcode: 1961Natur.192.1227C. doi:10.1038/1921227a0. PMID 13882203.
(78)^ Crick FH (October 1962). “The genetic code”. Scientific American (WH Freeman and Company) 207 (4): 66–74. Bibcode: 1962SciAm.207d..66C. doi:10.1038/scientificamerican1062-66. PMID 13882204.
(79)^ Woodson SA (May 1998). “Ironing out the kinks: splicing and translation in bacteria”. Genes & Development 12 (9): 1243–7. doi:10.1101/gad.12.9.1243. PMID 9573040.
(80)^ Jacob F, Monod J (June 1961). “Genetic regulatory mechanisms in the synthesis of proteins”. Journal of Molecular Biology 3 (3): 318–56. doi:10.1016/S0022-2836(61)80072-7. PMID 13718526.
(81)^ Koonin EV, Dolja VV (January 1993). “Evolution and taxonomy of positive-strand RNA viruses: implications of comparative analysis of amino acid sequences”. Critical Reviews in Biochemistry and Molecular Biology 28 (5): 375–430. doi:10.3109/10409239309078440. PMID 8269709.
(82)^ Domingo E (2001). “RNA Virus Genomes”. eLS. doi:10.1002/9780470015902.a0001488.pub2. ISBN 978-0470016176.
(83)^ Domingo E, Escarmís C, Sevilla N, Moya A, Elena SF, Quer J, Novella IS, Holland JJ (June 1996). “Basic concepts in RNA virus evolution”. FASEB Journal 10 (8): 859–64. doi:10.1096/fasebj.10.8.8666162. PMID 8666162.
(84)^ Miko I (2008). “Gregor Mendel and the Principles of Inheritance”. Nature Education Knowledge. SciTable (Nature Publishing Group) 1 (1): 134.
(85)^ Chial H (2008). “Mendelian Genetics: Patterns of Inheritance and Single-Gene Disorders”. Nature Education Knowledge. SciTable (Nature Publishing Group) 1 (1): 63.
(86)^ McCarthy D, Minner C, Bernstein H, Bernstein C (October 1976). “DNA elongation rates and growing point distributions of wild-type phage T4 and a DNA-delay amber mutant”. Journal of Molecular Biology 106 (4): 963–81. doi:10.1016/0022-2836(76)90346-6. PMID 789903.
(87)^ abLobo I, Shaw K (2008). “Discovery and Types of Genetic Linkage”. Nature Education Knowledge. SciTable (Nature Publishing Group) 1 (1): 139.
(88)^ Nachman MW, Crowell SL (September 2000). “Estimate of the mutation rate per nucleotide in humans”. Genetics 156 (1): 297–304. doi:10.1093/genetics/156.1.297. PMC 1461236. PMID 10978293.
(89)^ Roach JC, Glusman G, Smit AF, Huff CD, Hubley R, Shannon PT, Rowen L, Pant KP, Goodman N, Bamshad M, Shendure J, Drmanac R, Jorde LB, Hood L, Galas DJ (April 2010). “Analysis of genetic inheritance in a family quartet by whole-genome sequencing”. Science 328 (5978): 636–9. Bibcode: 2010Sci...328..636R. doi:10.1126/science.1186802. PMC 3037280. PMID 20220176.
(90)^ Drake JW, Charlesworth B, Charlesworth D, Crow JF (April 1998). “Rates of spontaneous mutation”. Genetics 148 (4): 1667–86. doi:10.1093/genetics/148.4.1667. PMC 1460098. PMID 9560386.
(91)^ Pyeritz, Reed E., Bruce R. Korf, and Wayne W. Grody, eds. Emery and Rimoin’s principles and practice of medical genetics and genomics: foundations. Academic Press, 2018.
(92)^ “What kinds of gene mutations are possible?”. Genetics Home Reference. United States National Library of Medicine (2015年5月11日). 2015年5月19日閲覧。
(93)^ Andrews CA (2010). “Natural Selection, Genetic Drift, and Gene Flow Do Not Act in Isolation in Natural Populations”. Nature Education Knowledge. SciTable (Nature Publishing Group) 3 (10): 5.
(94)^ Patterson C (November 1988). “Homology in classical and molecular biology”. Molecular Biology and Evolution 5 (6): 603–25. doi:10.1093/oxfordjournals.molbev.a040523. PMID 3065587.
(95)^ Graur D (2016). Molecular and Genome Evolution. Sunderland MA (US): Sinauer Associates, Inc.. ISBN 9781605354699
(96)^ Graur D (2016). Molecular and Genome Evolution. Sunderland MA (US): Sinauer Associates, Inc.. ISBN 9781605354699
(97)^ Jensen RA (2001). “Orthologs and paralogs - we need to get it right”. Genome Biology 2 (8): INTERACTIONS1002. doi:10.1186/gb-2001-2-8-interactions1002. PMC 138949. PMID 11532207.
(98)^ Studer RA, Robinson-Rechavi M (May 2009). “How confident can we be that orthologs are similar, but paralogs differ?”. Trends in Genetics 25 (5): 210–6. doi:10.1016/j.tig.2009.03.004. PMID 19368988.
(99)^ Altenhoff AM, Studer RA, Robinson-Rechavi M, Dessimoz C (2012). “Resolving the ortholog conjecture: orthologs tend to be weakly, but significantly, more similar in function than paralogs”. PLOS Computational Biology 8 (5): e1002514. Bibcode: 2012PLSCB...8E2514A. doi:10.1371/journal.pcbi.1002514. PMC 3355068. PMID 22615551.  (100)^ abGuerzoni D, McLysaght A (November 2011). “De novo origins of human genes”. PLOS Genetics 7 (11): e1002381. doi:10.1371/journal.pgen.1002381. PMC 3213182. PMID 22102832.
(100)^ abGuerzoni D, McLysaght A (November 2011). “De novo origins of human genes”. PLOS Genetics 7 (11): e1002381. doi:10.1371/journal.pgen.1002381. PMC 3213182. PMID 22102832.  (101)^ Reams AB, Roth JR (February 2015). “Mechanisms of gene duplication and amplification”. Cold Spring Harbor Perspectives in Biology 7 (2): a016592. doi:10.1101/cshperspect.a016592. PMC 4315931. PMID 25646380.
(102)^ Demuth JP, De Bie T, Stajich JE, Cristianini N, Hahn MW (December 2006). “The evolution of mammalian gene families”. PLOS ONE 1 (1): e85. Bibcode: 2006PLoSO...1...85D. doi:10.1371/journal.pone.0000085. PMC 1762380. PMID 17183716.
(101)^ Reams AB, Roth JR (February 2015). “Mechanisms of gene duplication and amplification”. Cold Spring Harbor Perspectives in Biology 7 (2): a016592. doi:10.1101/cshperspect.a016592. PMC 4315931. PMID 25646380.
(102)^ Demuth JP, De Bie T, Stajich JE, Cristianini N, Hahn MW (December 2006). “The evolution of mammalian gene families”. PLOS ONE 1 (1): e85. Bibcode: 2006PLoSO...1...85D. doi:10.1371/journal.pone.0000085. PMC 1762380. PMID 17183716.  (103)^ Knowles DG, McLysaght A (October 2009). “Recent de novo origin of human protein-coding genes”. Genome Research 19 (10): 1752–9. doi:10.1101/gr.095026.109. PMC 2765279. PMID 19726446.
(104)^ Wu DD, Irwin DM, Zhang YP (November 2011). “De novo origin of human protein-coding genes”. PLOS Genetics 7 (11): e1002379. doi:10.1371/journal.pgen.1002379. PMC 3213175. PMID 22102831.
(103)^ Knowles DG, McLysaght A (October 2009). “Recent de novo origin of human protein-coding genes”. Genome Research 19 (10): 1752–9. doi:10.1101/gr.095026.109. PMC 2765279. PMID 19726446.
(104)^ Wu DD, Irwin DM, Zhang YP (November 2011). “De novo origin of human protein-coding genes”. PLOS Genetics 7 (11): e1002379. doi:10.1371/journal.pgen.1002379. PMC 3213175. PMID 22102831.  (105)^ McLysaght A, Guerzoni D (September 2015). “New genes from non-coding sequence: the role of de novo protein-coding genes in eukaryotic evolutionary innovation”. Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences 370 (1678): 20140332. doi:10.1098/rstb.2014.0332. PMC 4571571. PMID 26323763.
(106)^ Neme R, Tautz D (February 2013). “Phylogenetic patterns of emergence of new genes support a model of frequent de novo evolution”. BMC Genomics 14 (1): 117. doi:10.1186/1471-2164-14-117. PMC 3616865. PMID 23433480.
(107)^ Treangen TJ, Rocha EP (January 2011). “Horizontal transfer, not duplication, drives the expansion of protein families in prokaryotes”. PLOS Genetics 7 (1): e1001284. doi:10.1371/journal.pgen.1001284. PMC 3029252. PMID 21298028.
(105)^ McLysaght A, Guerzoni D (September 2015). “New genes from non-coding sequence: the role of de novo protein-coding genes in eukaryotic evolutionary innovation”. Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences 370 (1678): 20140332. doi:10.1098/rstb.2014.0332. PMC 4571571. PMID 26323763.
(106)^ Neme R, Tautz D (February 2013). “Phylogenetic patterns of emergence of new genes support a model of frequent de novo evolution”. BMC Genomics 14 (1): 117. doi:10.1186/1471-2164-14-117. PMC 3616865. PMID 23433480.
(107)^ Treangen TJ, Rocha EP (January 2011). “Horizontal transfer, not duplication, drives the expansion of protein families in prokaryotes”. PLOS Genetics 7 (1): e1001284. doi:10.1371/journal.pgen.1001284. PMC 3029252. PMID 21298028.  (108)^ Ochman H, Lawrence JG, Groisman EA (May 2000). “Lateral gene transfer and the nature of bacterial innovation”. Nature 405 (6784): 299–304. Bibcode: 2000Natur.405..299O. doi:10.1038/35012500. PMID 10830951.
(109)^ Keeling PJ, Palmer JD (August 2008). “Horizontal gene transfer in eukaryotic evolution”. Nature Reviews. Genetics 9 (8): 605–18. doi:10.1038/nrg2386. PMID 18591983.
(110)^ Schönknecht G, Chen WH, Ternes CM, Barbier GG, Shrestha RP, Stanke M, Bräutigam A, Baker BJ, Banfield JF, Garavito RM, Carr K, Wilkerson C, Rensing SA, Gagneul D, Dickenson NE, Oesterhelt C, Lercher MJ, Weber AP (March 2013). “Gene transfer from bacteria and archaea facilitated evolution of an extremophilic eukaryote”. Science 339 (6124): 1207–10. Bibcode: 2013Sci...339.1207S. doi:10.1126/science.1231707. PMID 23471408.
(111)^ Ridley, M. (2006). Genome. New York, NY: Harper Perennial. ISBN 0-06-019497-9
(112)^ Banerjee S, Bhandary P, Woodhouse M, Sen TZ, Wise RP, Andorf CM (Apr 2021). “FINDER: an automated software package to annotate eukaryotic genes from RNA-Seq data and associated protein sequences”. BMC Bioinformatics 44 (9): e89. doi:10.1186/s12859-021-04120-9. PMC 8056616. PMID 33879057.
(113)^ Watson, JD, Baker TA, Bell SP, Gann A, Levine M, Losick R. (2004). "Ch9-10", Molecular Biology of the Gene, 5th ed., Peason Benjamin Cummings; CSHL Press.
(114)^ “Integr8 – A.thaliana Genome Statistics”. 2015年4月26日閲覧。
(115)^ “Understanding the Basics”. The Human Genome Project. 2015年4月26日閲覧。
(116)^ “WS227 Release Letter”. WormBase (2011年8月10日). 2013年11月28日時点のオリジナルよりアーカイブ。2013年11月19日閲覧。
(117)^ abYu J, Hu S, Wang J, Wong GK, Li S, Liu B, Deng Y, Dai L, Zhou Y, Zhang X, Cao M, Liu J, Sun J, Tang J, Chen Y, Huang X, Lin W, Ye C, Tong W, Cong L, Geng J, Han Y, Li L, Li W, Hu G, Huang X, Li W, Li J, Liu Z, Li L, Liu J, Qi Q, Liu J, Li L, Li T, Wang X, Lu H, Wu T, Zhu M, Ni P, Han H, Dong W, Ren X, Feng X, Cui P, Li X, Wang H, Xu X, Zhai W, Xu Z, Zhang J, He S, Zhang J, Xu J, Zhang K, Zheng X, Dong J, Zeng W, Tao L, Ye J, Tan J, Ren X, Chen X, He J, Liu D, Tian W, Tian C, Xia H, Bao Q, Li G, Gao H, Cao T, Wang J, Zhao W, Li P, Chen W, Wang X, Zhang Y, Hu J, Wang J, Liu S, Yang J, Zhang G, Xiong Y, Li Z, Mao L, Zhou C, Zhu Z, Chen R, Hao B, Zheng W, Chen S, Guo W, Li G, Liu S, Tao M, Wang J, Zhu L, Yuan L, Yang H (April 2002). “A draft sequence of the rice genome (Oryza sativa L. ssp. indica)”. Science 296 (5565): 79–92. Bibcode: 2002Sci...296...79Y. doi:10.1126/science.1068037. PMID 11935017.
(118)^ Anderson S, Bankier AT, Barrell BG, de Bruijn MH, Coulson AR, Drouin J, Eperon IC, Nierlich DP, Roe BA, Sanger F, Schreier PH, Smith AJ, Staden R, Young IG (April 1981). “Sequence and organization of the human mitochondrial genome”. Nature 290 (5806): 457–65. Bibcode: 1981Natur.290..457A. doi:10.1038/290457a0. PMID 7219534.
(119)^ Adams MD, Celniker SE, Holt RA, Evans CA, Gocayne JD, Amanatides PG, Scherer SE, Li PW, Hoskins RA, Galle RF, George RA, Lewis SE, Richards S, Ashburner M, Henderson SN, Sutton GG, Wortman JR, Yandell MD, Zhang Q, Chen LX, Brandon RC, Rogers YH, Blazej RG, Champe M, Pfeiffer BD, Wan KH, Doyle C, Baxter EG, Helt G, Nelson CR, Gabor GL, Abril JF, Agbayani A, An HJ, Andrews-Pfannkoch C, Baldwin D, Ballew RM, Basu A, Baxendale J, Bayraktaroglu L, Beasley EM, Beeson KY, Benos PV, Berman BP, Bhandari D, Bolshakov S, Borkova D, Botchan MR, Bouck J, Brokstein P, Brottier P, Burtis KC, Busam DA, Butler H, Cadieu E, Center A, Chandra I, Cherry JM, Cawley S, Dahlke C, Davenport LB, Davies P, de Pablos B, Delcher A, Deng Z, Mays AD, Dew I, Dietz SM, Dodson K, Doup LE, Downes M, Dugan-Rocha S, Dunkov BC, Dunn P, Durbin KJ, Evangelista CC, Ferraz C, Ferriera S, Fleischmann W, Fosler C, Gabrielian AE, Garg NS, Gelbart WM, Glasser K, Glodek A, Gong F, Gorrell JH, Gu Z, Guan P, Harris M, Harris NL, Harvey D, Heiman TJ, Hernandez JR, Houck J, Hostin D, Houston KA, Howland TJ, Wei MH, Ibegwam C, Jalali M, Kalush F, Karpen GH, Ke Z, Kennison JA, Ketchum KA, Kimmel BE, Kodira CD, Kraft C, Kravitz S, Kulp D, Lai Z, Lasko P, Lei Y, Levitsky AA, Li J, Li Z, Liang Y, Lin X, Liu X, Mattei B, McIntosh TC, McLeod MP, McPherson D, Merkulov G, Milshina NV, Mobarry C, Morris J, Moshrefi A, Mount SM, Moy M, Murphy B, Murphy L, Muzny DM, Nelson DL, Nelson DR, Nelson KA, Nixon K, Nusskern DR, Pacleb JM, Palazzolo M, Pittman GS, Pan S, Pollard J, Puri V, Reese MG, Reinert K, Remington K, Saunders RD, Scheeler F, Shen H, Shue BC, Sidén-Kiamos I, Simpson M, Skupski MP, Smith T, Spier E, Spradling AC, Stapleton M, Strong R, Sun E, Svirskas R, Tector C, Turner R, Venter E, Wang AH, Wang X, Wang ZY, Wassarman DA, Weinstock GM, Weissenbach J, Williams SM, Worley KC, Wu D, Yang S, Yao QA, Ye J, Yeh RF, Zaveri JS, Zhan M, Zhang G, Zhao Q, Zheng L, Zheng XH, Zhong FN, Zhong W, Zhou X, Zhu S, Zhu X, Smith HO, Gibbs RA, Myers EW, Rubin GM, Venter JC (March 2000). “The genome sequence of Drosophila melanogaster”. Science 287 (5461): 2185–95. Bibcode: 2000Sci...287.2185.. doi:10.1126/science.287.5461.2185. PMID 10731132.
(120)^ Pertea M, Salzberg SL (2010). “Between a chicken and a grape: estimating the number of human genes”. Genome Biology 11 (5): 206. doi:10.1186/gb-2010-11-5-206. PMC 2898077. PMID 20441615.
(121)^ Belyi VA, Levine AJ, Skalka AM (December 2010). “Sequences from ancestral single-stranded DNA viruses in vertebrate genomes: the parvoviridae and circoviridae are more than 40 to 50 million years old”. Journal of Virology 84 (23): 12458–62. doi:10.1128/JVI.01789-10. PMC 2976387. PMID 20861255.
(122)^ Flores R, Di Serio F, Hernández C (February 1997). “Viroids: The Noncoding Genomes”. Seminars in Virology 8 (1): 65–73. doi:10.1006/smvy.1997.0107.
(123)^ Zonneveld BJ (2010). “New Record Holders for Maximum Genome Size in Eudicots and Monocots”. Journal of Botany 2010: 1–4. doi:10.1155/2010/527357.
(124)^ Perez-Iratxeta C, Palidwor G, Andrade-Navarro MA (December 2007). “Towards completion of the Earth's proteome”. EMBO Reports 8 (12): 1135–41. doi:10.1038/sj.embor.7401117. PMC 2267224. PMID 18059312.
(125)^ Muller HJ (1966). “The gene material as the initiator and the organizing basis of life”. American Naturalist 100 (915): 493–517. doi:10.1086/282445. JSTOR 2459205.
(126)^ Ohno S (1972). “So much "junk" DNA in our genome”. Brookhaven Symposia in Biology 23: 366–370. PMID 5065367.
(127)^ Hatje K, Mühlhausen S, Simm D, Killmar M (2019). “The Protein-Coding Human Genome: Annotating High-Hanging Fruits.”. BioEssays 41 (11): 1900066. doi:10.1002/bies.201900066. PMID 31544971.
(128)^ Schuler GD, Boguski MS, Stewart EA, Stein LD, Gyapay G, Rice K, White RE, Rodriguez-Tomé P, Aggarwal A, Bajorek E, Bentolila S, Birren BB, Butler A, Castle AB, Chiannilkulchai N, Chu A, Clee C, Cowles S, Day PJ, Dibling T, Drouot N, Dunham I, Duprat S, East C, Edwards C, Fan JB, Fang N, Fizames C, Garrett C, Green L, Hadley D, Harris M, Harrison P, Brady S, Hicks A, Holloway E, Hui L, Hussain S, Louis-Dit-Sully C, Ma J, MacGilvery A, Mader C, Maratukulam A, Matise TC, McKusick KB, Morissette J, Mungall A, Muselet D, Nusbaum HC, Page DC, Peck A, Perkins S, Piercy M, Qin F, Quackenbush J, Ranby S, Reif T, Rozen S, Sanders C, She X, Silva J, Slonim DK, Soderlund C, Sun WL, Tabar P, Thangarajah T, Vega-Czarny N, Vollrath D, Voyticky S, Wilmer T, Wu X, Adams MD, Auffray C, Walter NA, Brandon R, Dehejia A, Goodfellow PN, Houlgatte R, Hudson JR, Ide SE, Iorio KR, Lee WY, Seki N, Nagase T, Ishikawa K, Nomura N, Phillips C, Polymeropoulos MH, Sandusky M, Schmitt K, Berry R, Swanson K, Torres R, Venter JC, Sikela JM, Beckmann JS, Weissenbach J, Myers RM, Cox DR, James MR, Bentley D, Deloukas P, Lander ES, Hudson TJ (October 1996). “A gene map of the human genome”. Science 274 (5287): 540–6. Bibcode: 1996Sci...274..540S. doi:10.1126/science.274.5287.540. PMID 8849440.
(129)^ Chi KR (October 2016). “The dark side of the human genome” (英語). Nature 538 (7624): 275–277. Bibcode: 2016Natur.538..275C. doi:10.1038/538275a. PMID 27734873.
(130)^ Ezkurdia, Iakes; Juan, David; Rodriguez, Jose Manuel; Frankish, Adam; Diekhans, Mark; Harrow, Jennifer; Vazquez, Jesus; Valencia, Alfonso et al. (2014-11-15). “Multiple evidence strands suggest that there may be as few as 19 000 human protein-coding genes” (英語). Human Molecular Genetics 23 (22): 5866–5878. doi:10.1093/hmg/ddu309. ISSN 0964-6906. PMC PMC4204768. PMID 24939910.
(131)^ “Human assembly and gene annotation”. Ensembl (2022年). 2023年2月28日閲覧。
(132)^ abHutchison CA, Chuang RY, Noskov VN, Assad-Garcia N, Deerinck TJ, Ellisman MH, Gill J, Kannan K, Karas BJ, Ma L, Pelletier JF, Qi ZQ, Richter RA, Strychalski EA, Sun L, Suzuki Y, Tsvetanova B, Wise KS, Smith HO, Glass JI, Merryman C, Gibson DG, Venter JC (March 2016). “Design and synthesis of a minimal bacterial genome”. Science 351 (6280): aad6253. Bibcode: 2016Sci...351.....H. doi:10.1126/science.aad6253. PMID 27013737.
(133)^ Glass JI, Assad-Garcia N, Alperovich N, Yooseph S, Lewis MR, Maruf M, Hutchison CA, Smith HO, Venter JC (January 2006). “Essential genes of a minimal bacterium”. Proceedings of the National Academy of Sciences of the United States of America 103 (2): 425–30. Bibcode: 2006PNAS..103..425G. doi:10.1073/pnas.0510013103. PMC 1324956. PMID 16407165.
(134)^ Gerdes SY, Scholle MD, Campbell JW, Balázsi G, Ravasz E, Daugherty MD, Somera AL, Kyrpides NC, Anderson I, Gelfand MS, Bhattacharya A, Kapatral V, D'Souza M, Baev MV, Grechkin Y, Mseeh F, Fonstein MY, Overbeek R, Barabási AL, Oltvai ZN, Osterman AL (October 2003). “Experimental determination and system level analysis of essential genes in Escherichia coli MG1655”. Journal of Bacteriology 185 (19): 5673–84. doi:10.1128/jb.185.19.5673-5684.2003. PMC 193955. PMID 13129938.
(135)^ Baba T, Ara T, Hasegawa M, Takai Y, Okumura Y, Baba M, Datsenko KA, Tomita M, Wanner BL, Mori H (2006). “Construction of Escherichia coli K-12 in-frame, single-gene knockout mutants: the Keio collection”. Molecular Systems Biology 2: 2006.0008. doi:10.1038/msb4100050. PMC 1681482. PMID 16738554.
(136)^ abJuhas M, Reuß DR, Zhu B, Commichau FM (November 2014). “Bacillus subtilis and Escherichia coli essential genes and minimal cell factories after one decade of genome engineering”. Microbiology 160 (Pt 11): 2341–2351. doi:10.1099/mic.0.079376-0. PMID 25092907.
(137)^ Tu Z, Wang L, Xu M, Zhou X, Chen T, Sun F (February 2006). “Further understanding human disease genes by comparing with housekeeping genes and other genes”. BMC Genomics 7: 31. doi:10.1186/1471-2164-7-31. PMC 1397819. PMID 16504025.
(108)^ Ochman H, Lawrence JG, Groisman EA (May 2000). “Lateral gene transfer and the nature of bacterial innovation”. Nature 405 (6784): 299–304. Bibcode: 2000Natur.405..299O. doi:10.1038/35012500. PMID 10830951.
(109)^ Keeling PJ, Palmer JD (August 2008). “Horizontal gene transfer in eukaryotic evolution”. Nature Reviews. Genetics 9 (8): 605–18. doi:10.1038/nrg2386. PMID 18591983.
(110)^ Schönknecht G, Chen WH, Ternes CM, Barbier GG, Shrestha RP, Stanke M, Bräutigam A, Baker BJ, Banfield JF, Garavito RM, Carr K, Wilkerson C, Rensing SA, Gagneul D, Dickenson NE, Oesterhelt C, Lercher MJ, Weber AP (March 2013). “Gene transfer from bacteria and archaea facilitated evolution of an extremophilic eukaryote”. Science 339 (6124): 1207–10. Bibcode: 2013Sci...339.1207S. doi:10.1126/science.1231707. PMID 23471408.
(111)^ Ridley, M. (2006). Genome. New York, NY: Harper Perennial. ISBN 0-06-019497-9
(112)^ Banerjee S, Bhandary P, Woodhouse M, Sen TZ, Wise RP, Andorf CM (Apr 2021). “FINDER: an automated software package to annotate eukaryotic genes from RNA-Seq data and associated protein sequences”. BMC Bioinformatics 44 (9): e89. doi:10.1186/s12859-021-04120-9. PMC 8056616. PMID 33879057.
(113)^ Watson, JD, Baker TA, Bell SP, Gann A, Levine M, Losick R. (2004). "Ch9-10", Molecular Biology of the Gene, 5th ed., Peason Benjamin Cummings; CSHL Press.
(114)^ “Integr8 – A.thaliana Genome Statistics”. 2015年4月26日閲覧。
(115)^ “Understanding the Basics”. The Human Genome Project. 2015年4月26日閲覧。
(116)^ “WS227 Release Letter”. WormBase (2011年8月10日). 2013年11月28日時点のオリジナルよりアーカイブ。2013年11月19日閲覧。
(117)^ abYu J, Hu S, Wang J, Wong GK, Li S, Liu B, Deng Y, Dai L, Zhou Y, Zhang X, Cao M, Liu J, Sun J, Tang J, Chen Y, Huang X, Lin W, Ye C, Tong W, Cong L, Geng J, Han Y, Li L, Li W, Hu G, Huang X, Li W, Li J, Liu Z, Li L, Liu J, Qi Q, Liu J, Li L, Li T, Wang X, Lu H, Wu T, Zhu M, Ni P, Han H, Dong W, Ren X, Feng X, Cui P, Li X, Wang H, Xu X, Zhai W, Xu Z, Zhang J, He S, Zhang J, Xu J, Zhang K, Zheng X, Dong J, Zeng W, Tao L, Ye J, Tan J, Ren X, Chen X, He J, Liu D, Tian W, Tian C, Xia H, Bao Q, Li G, Gao H, Cao T, Wang J, Zhao W, Li P, Chen W, Wang X, Zhang Y, Hu J, Wang J, Liu S, Yang J, Zhang G, Xiong Y, Li Z, Mao L, Zhou C, Zhu Z, Chen R, Hao B, Zheng W, Chen S, Guo W, Li G, Liu S, Tao M, Wang J, Zhu L, Yuan L, Yang H (April 2002). “A draft sequence of the rice genome (Oryza sativa L. ssp. indica)”. Science 296 (5565): 79–92. Bibcode: 2002Sci...296...79Y. doi:10.1126/science.1068037. PMID 11935017.
(118)^ Anderson S, Bankier AT, Barrell BG, de Bruijn MH, Coulson AR, Drouin J, Eperon IC, Nierlich DP, Roe BA, Sanger F, Schreier PH, Smith AJ, Staden R, Young IG (April 1981). “Sequence and organization of the human mitochondrial genome”. Nature 290 (5806): 457–65. Bibcode: 1981Natur.290..457A. doi:10.1038/290457a0. PMID 7219534.
(119)^ Adams MD, Celniker SE, Holt RA, Evans CA, Gocayne JD, Amanatides PG, Scherer SE, Li PW, Hoskins RA, Galle RF, George RA, Lewis SE, Richards S, Ashburner M, Henderson SN, Sutton GG, Wortman JR, Yandell MD, Zhang Q, Chen LX, Brandon RC, Rogers YH, Blazej RG, Champe M, Pfeiffer BD, Wan KH, Doyle C, Baxter EG, Helt G, Nelson CR, Gabor GL, Abril JF, Agbayani A, An HJ, Andrews-Pfannkoch C, Baldwin D, Ballew RM, Basu A, Baxendale J, Bayraktaroglu L, Beasley EM, Beeson KY, Benos PV, Berman BP, Bhandari D, Bolshakov S, Borkova D, Botchan MR, Bouck J, Brokstein P, Brottier P, Burtis KC, Busam DA, Butler H, Cadieu E, Center A, Chandra I, Cherry JM, Cawley S, Dahlke C, Davenport LB, Davies P, de Pablos B, Delcher A, Deng Z, Mays AD, Dew I, Dietz SM, Dodson K, Doup LE, Downes M, Dugan-Rocha S, Dunkov BC, Dunn P, Durbin KJ, Evangelista CC, Ferraz C, Ferriera S, Fleischmann W, Fosler C, Gabrielian AE, Garg NS, Gelbart WM, Glasser K, Glodek A, Gong F, Gorrell JH, Gu Z, Guan P, Harris M, Harris NL, Harvey D, Heiman TJ, Hernandez JR, Houck J, Hostin D, Houston KA, Howland TJ, Wei MH, Ibegwam C, Jalali M, Kalush F, Karpen GH, Ke Z, Kennison JA, Ketchum KA, Kimmel BE, Kodira CD, Kraft C, Kravitz S, Kulp D, Lai Z, Lasko P, Lei Y, Levitsky AA, Li J, Li Z, Liang Y, Lin X, Liu X, Mattei B, McIntosh TC, McLeod MP, McPherson D, Merkulov G, Milshina NV, Mobarry C, Morris J, Moshrefi A, Mount SM, Moy M, Murphy B, Murphy L, Muzny DM, Nelson DL, Nelson DR, Nelson KA, Nixon K, Nusskern DR, Pacleb JM, Palazzolo M, Pittman GS, Pan S, Pollard J, Puri V, Reese MG, Reinert K, Remington K, Saunders RD, Scheeler F, Shen H, Shue BC, Sidén-Kiamos I, Simpson M, Skupski MP, Smith T, Spier E, Spradling AC, Stapleton M, Strong R, Sun E, Svirskas R, Tector C, Turner R, Venter E, Wang AH, Wang X, Wang ZY, Wassarman DA, Weinstock GM, Weissenbach J, Williams SM, Worley KC, Wu D, Yang S, Yao QA, Ye J, Yeh RF, Zaveri JS, Zhan M, Zhang G, Zhao Q, Zheng L, Zheng XH, Zhong FN, Zhong W, Zhou X, Zhu S, Zhu X, Smith HO, Gibbs RA, Myers EW, Rubin GM, Venter JC (March 2000). “The genome sequence of Drosophila melanogaster”. Science 287 (5461): 2185–95. Bibcode: 2000Sci...287.2185.. doi:10.1126/science.287.5461.2185. PMID 10731132.
(120)^ Pertea M, Salzberg SL (2010). “Between a chicken and a grape: estimating the number of human genes”. Genome Biology 11 (5): 206. doi:10.1186/gb-2010-11-5-206. PMC 2898077. PMID 20441615.
(121)^ Belyi VA, Levine AJ, Skalka AM (December 2010). “Sequences from ancestral single-stranded DNA viruses in vertebrate genomes: the parvoviridae and circoviridae are more than 40 to 50 million years old”. Journal of Virology 84 (23): 12458–62. doi:10.1128/JVI.01789-10. PMC 2976387. PMID 20861255.
(122)^ Flores R, Di Serio F, Hernández C (February 1997). “Viroids: The Noncoding Genomes”. Seminars in Virology 8 (1): 65–73. doi:10.1006/smvy.1997.0107.
(123)^ Zonneveld BJ (2010). “New Record Holders for Maximum Genome Size in Eudicots and Monocots”. Journal of Botany 2010: 1–4. doi:10.1155/2010/527357.
(124)^ Perez-Iratxeta C, Palidwor G, Andrade-Navarro MA (December 2007). “Towards completion of the Earth's proteome”. EMBO Reports 8 (12): 1135–41. doi:10.1038/sj.embor.7401117. PMC 2267224. PMID 18059312.
(125)^ Muller HJ (1966). “The gene material as the initiator and the organizing basis of life”. American Naturalist 100 (915): 493–517. doi:10.1086/282445. JSTOR 2459205.
(126)^ Ohno S (1972). “So much "junk" DNA in our genome”. Brookhaven Symposia in Biology 23: 366–370. PMID 5065367.
(127)^ Hatje K, Mühlhausen S, Simm D, Killmar M (2019). “The Protein-Coding Human Genome: Annotating High-Hanging Fruits.”. BioEssays 41 (11): 1900066. doi:10.1002/bies.201900066. PMID 31544971.
(128)^ Schuler GD, Boguski MS, Stewart EA, Stein LD, Gyapay G, Rice K, White RE, Rodriguez-Tomé P, Aggarwal A, Bajorek E, Bentolila S, Birren BB, Butler A, Castle AB, Chiannilkulchai N, Chu A, Clee C, Cowles S, Day PJ, Dibling T, Drouot N, Dunham I, Duprat S, East C, Edwards C, Fan JB, Fang N, Fizames C, Garrett C, Green L, Hadley D, Harris M, Harrison P, Brady S, Hicks A, Holloway E, Hui L, Hussain S, Louis-Dit-Sully C, Ma J, MacGilvery A, Mader C, Maratukulam A, Matise TC, McKusick KB, Morissette J, Mungall A, Muselet D, Nusbaum HC, Page DC, Peck A, Perkins S, Piercy M, Qin F, Quackenbush J, Ranby S, Reif T, Rozen S, Sanders C, She X, Silva J, Slonim DK, Soderlund C, Sun WL, Tabar P, Thangarajah T, Vega-Czarny N, Vollrath D, Voyticky S, Wilmer T, Wu X, Adams MD, Auffray C, Walter NA, Brandon R, Dehejia A, Goodfellow PN, Houlgatte R, Hudson JR, Ide SE, Iorio KR, Lee WY, Seki N, Nagase T, Ishikawa K, Nomura N, Phillips C, Polymeropoulos MH, Sandusky M, Schmitt K, Berry R, Swanson K, Torres R, Venter JC, Sikela JM, Beckmann JS, Weissenbach J, Myers RM, Cox DR, James MR, Bentley D, Deloukas P, Lander ES, Hudson TJ (October 1996). “A gene map of the human genome”. Science 274 (5287): 540–6. Bibcode: 1996Sci...274..540S. doi:10.1126/science.274.5287.540. PMID 8849440.
(129)^ Chi KR (October 2016). “The dark side of the human genome” (英語). Nature 538 (7624): 275–277. Bibcode: 2016Natur.538..275C. doi:10.1038/538275a. PMID 27734873.
(130)^ Ezkurdia, Iakes; Juan, David; Rodriguez, Jose Manuel; Frankish, Adam; Diekhans, Mark; Harrow, Jennifer; Vazquez, Jesus; Valencia, Alfonso et al. (2014-11-15). “Multiple evidence strands suggest that there may be as few as 19 000 human protein-coding genes” (英語). Human Molecular Genetics 23 (22): 5866–5878. doi:10.1093/hmg/ddu309. ISSN 0964-6906. PMC PMC4204768. PMID 24939910.
(131)^ “Human assembly and gene annotation”. Ensembl (2022年). 2023年2月28日閲覧。
(132)^ abHutchison CA, Chuang RY, Noskov VN, Assad-Garcia N, Deerinck TJ, Ellisman MH, Gill J, Kannan K, Karas BJ, Ma L, Pelletier JF, Qi ZQ, Richter RA, Strychalski EA, Sun L, Suzuki Y, Tsvetanova B, Wise KS, Smith HO, Glass JI, Merryman C, Gibson DG, Venter JC (March 2016). “Design and synthesis of a minimal bacterial genome”. Science 351 (6280): aad6253. Bibcode: 2016Sci...351.....H. doi:10.1126/science.aad6253. PMID 27013737.
(133)^ Glass JI, Assad-Garcia N, Alperovich N, Yooseph S, Lewis MR, Maruf M, Hutchison CA, Smith HO, Venter JC (January 2006). “Essential genes of a minimal bacterium”. Proceedings of the National Academy of Sciences of the United States of America 103 (2): 425–30. Bibcode: 2006PNAS..103..425G. doi:10.1073/pnas.0510013103. PMC 1324956. PMID 16407165.
(134)^ Gerdes SY, Scholle MD, Campbell JW, Balázsi G, Ravasz E, Daugherty MD, Somera AL, Kyrpides NC, Anderson I, Gelfand MS, Bhattacharya A, Kapatral V, D'Souza M, Baev MV, Grechkin Y, Mseeh F, Fonstein MY, Overbeek R, Barabási AL, Oltvai ZN, Osterman AL (October 2003). “Experimental determination and system level analysis of essential genes in Escherichia coli MG1655”. Journal of Bacteriology 185 (19): 5673–84. doi:10.1128/jb.185.19.5673-5684.2003. PMC 193955. PMID 13129938.
(135)^ Baba T, Ara T, Hasegawa M, Takai Y, Okumura Y, Baba M, Datsenko KA, Tomita M, Wanner BL, Mori H (2006). “Construction of Escherichia coli K-12 in-frame, single-gene knockout mutants: the Keio collection”. Molecular Systems Biology 2: 2006.0008. doi:10.1038/msb4100050. PMC 1681482. PMID 16738554.
(136)^ abJuhas M, Reuß DR, Zhu B, Commichau FM (November 2014). “Bacillus subtilis and Escherichia coli essential genes and minimal cell factories after one decade of genome engineering”. Microbiology 160 (Pt 11): 2341–2351. doi:10.1099/mic.0.079376-0. PMID 25092907.
(137)^ Tu Z, Wang L, Xu M, Zhou X, Chen T, Sun F (February 2006). “Further understanding human disease genes by comparing with housekeeping genes and other genes”. BMC Genomics 7: 31. doi:10.1186/1471-2164-7-31. PMC 1397819. PMID 16504025.  (138)^ Georgi B, Voight BF, Bućan M (May 2013). “From mouse to human: evolutionary genomics analysis of human orthologs of essential genes”. PLOS Genetics 9 (5): e1003484. doi:10.1371/journal.pgen.1003484. PMC 3649967. PMID 23675308.
(138)^ Georgi B, Voight BF, Bućan M (May 2013). “From mouse to human: evolutionary genomics analysis of human orthologs of essential genes”. PLOS Genetics 9 (5): e1003484. doi:10.1371/journal.pgen.1003484. PMC 3649967. PMID 23675308.  (139)^ Eisenberg E, Levanon EY (October 2013). “Human housekeeping genes, revisited”. Trends in Genetics 29 (10): 569–74. doi:10.1016/j.tig.2013.05.010. PMID 23810203.
(140)^ Amsterdam A, Hopkins N (September 2006). “Mutagenesis strategies in zebrafish for identifying genes involved in development and disease”. Trends in Genetics 22 (9): 473–8. doi:10.1016/j.tig.2006.06.011. PMID 16844256.
(141)^ “About the HGNC”. HGNC Database of Human Gene Names. HUGO Gene Nomenclature Committee. 2015年5月14日閲覧。
(142)^ Cohen SN, Chang AC (May 1973). “Recircularization and autonomous replication of a sheared R-factor DNA segment in Escherichia coli transformants”. Proceedings of the National Academy of Sciences of the United States of America 70 (5): 1293–7. Bibcode: 1973PNAS...70.1293C. doi:10.1073/pnas.70.5.1293. PMC 433482. PMID 4576014.
(143)^ Esvelt KM, Wang HH (2013). “Genome-scale engineering for systems and synthetic biology”. Molecular Systems Biology 9 (1): 641. doi:10.1038/msb.2012.66. PMC 3564264. PMID 23340847.
(144)^ Tan WS, Carlson DF, Walton MW, Fahrenkrug SC, Hackett PB (2012). “Precision editing of large animal genomes”. Advances in Genetics Volume 80. 80. pp. 37–97. doi:10.1016/B978-0-12-404742-6.00002-8. ISBN 9780124047426. PMC 3683964. PMID 23084873
(145)^ Puchta H, Fauser F (2013). “Gene targeting in plants: 25 years later”. The International Journal of Developmental Biology 57 (6–8): 629–37. doi:10.1387/ijdb.130194hp. PMID 24166445.
(146)^ Ran FA, Hsu PD, Wright J, Agarwala V, Scott DA, Zhang F (November 2013). “Genome engineering using the CRISPR-Cas9 system”. Nature Protocols 8 (11): 2281–2308. doi:10.1038/nprot.2013.143. PMC 3969860. PMID 24157548.
(147)^ Kittleson JT, Wu GC, Anderson JC (August 2012). “Successes and failures in modular genetic engineering”. Current Opinion in Chemical Biology 16 (3–4): 329–36. doi:10.1016/j.cbpa.2012.06.009. PMID 22818777.
(148)^ Berg P, Mertz JE (January 2010). “Personal reflections on the origins and emergence of recombinant DNA technology”. Genetics 184 (1): 9–17. doi:10.1534/genetics.109.112144. PMC 2815933. PMID 20061565.
(149)^ Guan C, Ye C, Yang X, Gao J (February 2010). “A review of current large-scale mouse knockout efforts”. Genesis 48 (2): 73–85. doi:10.1002/dvg.20594. PMID 20095055.
(150)^ Austin CP, Battey JF, Bradley A, Bucan M, Capecchi M, Collins FS, Dove WF, Duyk G, Dymecki S, Eppig JT, Grieder FB, Heintz N, Hicks G, Insel TR, Joyner A, Koller BH, Lloyd KC, Magnuson T, Moore MW, Nagy A, Pollock JD, Roses AD, Sands AT, Seed B, Skarnes WC, Snoddy J, Soriano P, Stewart DJ, Stewart F, Stillman B, Varmus H, Varticovski L, Verma IM, Vogt TF, von Melchner H, Witkowski J, Woychik RP, Wurst W, Yancopoulos GD, Young SG, Zambrowicz B (September 2004). “The knockout mouse project”. Nature Genetics 36 (9): 921–4. doi:10.1038/ng0904-921. PMC 2716027. PMID 15340423.
(151)^ Deng C (October 2007). “In celebration of Dr. Mario R. Capecchi's Nobel Prize”. International Journal of Biological Sciences 3 (7): 417–9. doi:10.7150/ijbs.3.417. PMC 2043165. PMID 17998949.
(139)^ Eisenberg E, Levanon EY (October 2013). “Human housekeeping genes, revisited”. Trends in Genetics 29 (10): 569–74. doi:10.1016/j.tig.2013.05.010. PMID 23810203.
(140)^ Amsterdam A, Hopkins N (September 2006). “Mutagenesis strategies in zebrafish for identifying genes involved in development and disease”. Trends in Genetics 22 (9): 473–8. doi:10.1016/j.tig.2006.06.011. PMID 16844256.
(141)^ “About the HGNC”. HGNC Database of Human Gene Names. HUGO Gene Nomenclature Committee. 2015年5月14日閲覧。
(142)^ Cohen SN, Chang AC (May 1973). “Recircularization and autonomous replication of a sheared R-factor DNA segment in Escherichia coli transformants”. Proceedings of the National Academy of Sciences of the United States of America 70 (5): 1293–7. Bibcode: 1973PNAS...70.1293C. doi:10.1073/pnas.70.5.1293. PMC 433482. PMID 4576014.
(143)^ Esvelt KM, Wang HH (2013). “Genome-scale engineering for systems and synthetic biology”. Molecular Systems Biology 9 (1): 641. doi:10.1038/msb.2012.66. PMC 3564264. PMID 23340847.
(144)^ Tan WS, Carlson DF, Walton MW, Fahrenkrug SC, Hackett PB (2012). “Precision editing of large animal genomes”. Advances in Genetics Volume 80. 80. pp. 37–97. doi:10.1016/B978-0-12-404742-6.00002-8. ISBN 9780124047426. PMC 3683964. PMID 23084873
(145)^ Puchta H, Fauser F (2013). “Gene targeting in plants: 25 years later”. The International Journal of Developmental Biology 57 (6–8): 629–37. doi:10.1387/ijdb.130194hp. PMID 24166445.
(146)^ Ran FA, Hsu PD, Wright J, Agarwala V, Scott DA, Zhang F (November 2013). “Genome engineering using the CRISPR-Cas9 system”. Nature Protocols 8 (11): 2281–2308. doi:10.1038/nprot.2013.143. PMC 3969860. PMID 24157548.
(147)^ Kittleson JT, Wu GC, Anderson JC (August 2012). “Successes and failures in modular genetic engineering”. Current Opinion in Chemical Biology 16 (3–4): 329–36. doi:10.1016/j.cbpa.2012.06.009. PMID 22818777.
(148)^ Berg P, Mertz JE (January 2010). “Personal reflections on the origins and emergence of recombinant DNA technology”. Genetics 184 (1): 9–17. doi:10.1534/genetics.109.112144. PMC 2815933. PMID 20061565.
(149)^ Guan C, Ye C, Yang X, Gao J (February 2010). “A review of current large-scale mouse knockout efforts”. Genesis 48 (2): 73–85. doi:10.1002/dvg.20594. PMID 20095055.
(150)^ Austin CP, Battey JF, Bradley A, Bucan M, Capecchi M, Collins FS, Dove WF, Duyk G, Dymecki S, Eppig JT, Grieder FB, Heintz N, Hicks G, Insel TR, Joyner A, Koller BH, Lloyd KC, Magnuson T, Moore MW, Nagy A, Pollock JD, Roses AD, Sands AT, Seed B, Skarnes WC, Snoddy J, Soriano P, Stewart DJ, Stewart F, Stillman B, Varmus H, Varticovski L, Verma IM, Vogt TF, von Melchner H, Witkowski J, Woychik RP, Wurst W, Yancopoulos GD, Young SG, Zambrowicz B (September 2004). “The knockout mouse project”. Nature Genetics 36 (9): 921–4. doi:10.1038/ng0904-921. PMC 2716027. PMID 15340423.
(151)^ Deng C (October 2007). “In celebration of Dr. Mario R. Capecchi's Nobel Prize”. International Journal of Biological Sciences 3 (7): 417–9. doi:10.7150/ijbs.3.417. PMC 2043165. PMID 17998949.
 (56)^ Braig M, Schmitt CA (March 2006). “Oncogene-induced senescence: putting the brakes on tumor development”. Cancer Research 66 (6): 2881–4. doi:10.1158/0008-5472.CAN-05-4006. PMID 16540631.
(57)^ abBennett PM (March 2008). “Plasmid encoded antibiotic resistance: acquisition and transfer of antibiotic resistance genes in bacteria”. British Journal of Pharmacology 153 (Suppl 1): S347-57. doi:10.1038/sj.bjp.0707607. PMC 2268074. PMID 18193080.
(58)^ International Human Genome Sequencing Consortium (October 2004). “Finishing the euchromatic sequence of the human genome”. Nature 431 (7011): 931–45. Bibcode: 2004Natur.431..931H. doi:10.1038/nature03001. PMID 15496913.
(59)^ abShafee, Thomas; Lowe, Rohan (2017). “Eukaryotic and prokaryotic gene structure”. WikiJournal of Medicine 4 (1). doi:10.15347/wjm/2017.002. ISSN 2002-4436.
(60)^ Mortazavi A, Williams BA, McCue K, Schaeffer L, Wold B (July 2008). “Mapping and quantifying mammalian transcriptomes by RNA-Seq”. Nature Methods 5 (7): 621–8. doi:10.1038/nmeth.1226. PMID 18516045.
(61)^ Pennacchio LA, Bickmore W, Dean A, Nobrega MA, Bejerano G (April 2013). “Enhancers: five essential questions”. Nature Reviews. Genetics 14 (4): 288–95. doi:10.1038/nrg3458. PMC 4445073. PMID 23503198.
(62)^ Maston GA, Evans SK, Green MR (2006). “Transcriptional regulatory elements in the human genome”. Annual Review of Genomics and Human Genetics 7: 29–59. doi:10.1146/annurev.genom.7.080505.115623. PMID 16719718.
(63)^ Mignone F, Gissi C, Liuni S, Pesole G (2002-02-28). “Untranslated regions of mRNAs”. Genome Biology 3 (3): REVIEWS0004. doi:10.1186/gb-2002-3-3-reviews0004. PMC 139023. PMID 11897027.
(64)^ Bicknell AA, Cenik C, Chua HN, Roth FP, Moore MJ (December 2012). “Introns in UTRs: why we should stop ignoring them”. BioEssays 34 (12): 1025–34. doi:10.1002/bies.201200073. PMID 23108796.
(65)^ Shkurin A, Pour SE, Hughes TR (April 2023). “Known sequence features explain half of all human gene ends”. NAR Genomics and Bioinformatics 5 (2): lqad031. doi:10.1093/nargab/lqad031. PMC 10072996. PMID 37035540.
(66)^ Salgado H, Moreno-Hagelsieb G, Smith TF, Collado-Vides J (June 2000). “Operons in Escherichia coli: genomic analyses and predictions”. Proceedings of the National Academy of Sciences of the United States of America 97 (12): 6652–7. Bibcode: 2000PNAS...97.6652S. doi:10.1073/pnas.110147297. PMC 18690. PMID 10823905.
(67)^ Blumenthal T (November 2004). “Operons in eukaryotes”. Briefings in Functional Genomics & Proteomics 3 (3): 199–211. doi:10.1093/bfgp/3.3.199. PMID 15642184.
(68)^ Jacob F, Monod J (June 1961). “Genetic regulatory mechanisms in the synthesis of proteins”. Journal of Molecular Biology 3 (3): 318–56. doi:10.1016/S0022-2836(61)80072-7. PMID 13718526.
(69)^ Pozzoli U, Menozzi G, Comi GP, Cagliani R, Bresolin N, Sironi M (January 2007). “Intron size in mammals: complexity comes to terms with economy”. Trends in Genetics 23 (1): 20–24. doi:10.1016/j.tig.2006.10.003. PMID 17070957.
(70)^ Marais G, Nouvellet P, Keightley PD, Charlesworth B (May 2005). “Intron size and exon evolution in Drosophila”. Genetics 170 (1): 481–485. doi:10.1534/genetics.104.037333. PMC 1449718. PMID 15781704.
(71)^ Kumar A (September 2009). “An overview of nested genes in eukaryotic genomes”. Eukaryotic Cell 8 (9): 1321–1329. doi:10.1128/EC.00143-09. PMC 2747821. PMID 19542305..
(72)^ Spilianakis CG, Lalioti MD, Town T, Lee GR, Flavell RA (June 2005). “Interchromosomal associations between alternatively expressed loci”. Nature 435 (7042): 637–645. Bibcode: 2005Natur.435..637S. doi:10.1038/nature03574. PMID 15880101.
(73)^ Williams A, Spilianakis CG, Flavell RA (April 2010). “Interchromosomal association and gene regulation in trans”. Trends in Genetics 26 (4): 188–197. doi:10.1016/j.tig.2010.01.007. PMC 2865229. PMID 20236724.
(74)^ Lei Q, Li C, Zuo Z, Huang C, Cheng H, Zhou R (March 2016). “Evolutionary Insights into RNA trans-Splicing in Vertebrates”. Genome Biology and Evolution 8 (3): 562–577. doi:10.1093/gbe/evw025. PMC 4824033. PMID 26966239.
(75)^ Wright BW, Molloy MP, Jaschke PR (March 2022). “Overlapping genes in natural and engineered genomes”. Nature Reviews. Genetics 23 (3): 154–168. doi:10.1038/s41576-021-00417-w. PMC 8490965. PMID 34611352.
(76)^ abEddy SR (December 2001). “Non-coding RNA genes and the modern RNA world”. Nature Reviews. Genetics 2 (12): 919–29. doi:10.1038/35103511. PMID 11733745.
(77)^ Crick FH, Barnett L, Brenner S, Watts-Tobin RJ (December 1961). “General nature of the genetic code for proteins”. Nature 192 (4809): 1227–32. Bibcode: 1961Natur.192.1227C. doi:10.1038/1921227a0. PMID 13882203.
(78)^ Crick FH (October 1962). “The genetic code”. Scientific American (WH Freeman and Company) 207 (4): 66–74. Bibcode: 1962SciAm.207d..66C. doi:10.1038/scientificamerican1062-66. PMID 13882204.
(79)^ Woodson SA (May 1998). “Ironing out the kinks: splicing and translation in bacteria”. Genes & Development 12 (9): 1243–7. doi:10.1101/gad.12.9.1243. PMID 9573040.
(80)^ Jacob F, Monod J (June 1961). “Genetic regulatory mechanisms in the synthesis of proteins”. Journal of Molecular Biology 3 (3): 318–56. doi:10.1016/S0022-2836(61)80072-7. PMID 13718526.
(81)^ Koonin EV, Dolja VV (January 1993). “Evolution and taxonomy of positive-strand RNA viruses: implications of comparative analysis of amino acid sequences”. Critical Reviews in Biochemistry and Molecular Biology 28 (5): 375–430. doi:10.3109/10409239309078440. PMID 8269709.
(82)^ Domingo E (2001). “RNA Virus Genomes”. eLS. doi:10.1002/9780470015902.a0001488.pub2. ISBN 978-0470016176.
(83)^ Domingo E, Escarmís C, Sevilla N, Moya A, Elena SF, Quer J, Novella IS, Holland JJ (June 1996). “Basic concepts in RNA virus evolution”. FASEB Journal 10 (8): 859–64. doi:10.1096/fasebj.10.8.8666162. PMID 8666162.
(84)^ Miko I (2008). “Gregor Mendel and the Principles of Inheritance”. Nature Education Knowledge. SciTable (Nature Publishing Group) 1 (1): 134.
(85)^ Chial H (2008). “Mendelian Genetics: Patterns of Inheritance and Single-Gene Disorders”. Nature Education Knowledge. SciTable (Nature Publishing Group) 1 (1): 63.
(86)^ McCarthy D, Minner C, Bernstein H, Bernstein C (October 1976). “DNA elongation rates and growing point distributions of wild-type phage T4 and a DNA-delay amber mutant”. Journal of Molecular Biology 106 (4): 963–81. doi:10.1016/0022-2836(76)90346-6. PMID 789903.
(87)^ abLobo I, Shaw K (2008). “Discovery and Types of Genetic Linkage”. Nature Education Knowledge. SciTable (Nature Publishing Group) 1 (1): 139.
(88)^ Nachman MW, Crowell SL (September 2000). “Estimate of the mutation rate per nucleotide in humans”. Genetics 156 (1): 297–304. doi:10.1093/genetics/156.1.297. PMC 1461236. PMID 10978293.
(89)^ Roach JC, Glusman G, Smit AF, Huff CD, Hubley R, Shannon PT, Rowen L, Pant KP, Goodman N, Bamshad M, Shendure J, Drmanac R, Jorde LB, Hood L, Galas DJ (April 2010). “Analysis of genetic inheritance in a family quartet by whole-genome sequencing”. Science 328 (5978): 636–9. Bibcode: 2010Sci...328..636R. doi:10.1126/science.1186802. PMC 3037280. PMID 20220176.
(90)^ Drake JW, Charlesworth B, Charlesworth D, Crow JF (April 1998). “Rates of spontaneous mutation”. Genetics 148 (4): 1667–86. doi:10.1093/genetics/148.4.1667. PMC 1460098. PMID 9560386.
(91)^ Pyeritz, Reed E., Bruce R. Korf, and Wayne W. Grody, eds. Emery and Rimoin’s principles and practice of medical genetics and genomics: foundations. Academic Press, 2018.
(92)^ “What kinds of gene mutations are possible?”. Genetics Home Reference. United States National Library of Medicine (2015年5月11日). 2015年5月19日閲覧。
(93)^ Andrews CA (2010). “Natural Selection, Genetic Drift, and Gene Flow Do Not Act in Isolation in Natural Populations”. Nature Education Knowledge. SciTable (Nature Publishing Group) 3 (10): 5.
(94)^ Patterson C (November 1988). “Homology in classical and molecular biology”. Molecular Biology and Evolution 5 (6): 603–25. doi:10.1093/oxfordjournals.molbev.a040523. PMID 3065587.
(95)^ Graur D (2016). Molecular and Genome Evolution. Sunderland MA (US): Sinauer Associates, Inc.. ISBN 9781605354699
(96)^ Graur D (2016). Molecular and Genome Evolution. Sunderland MA (US): Sinauer Associates, Inc.. ISBN 9781605354699
(97)^ Jensen RA (2001). “Orthologs and paralogs - we need to get it right”. Genome Biology 2 (8): INTERACTIONS1002. doi:10.1186/gb-2001-2-8-interactions1002. PMC 138949. PMID 11532207.
(98)^ Studer RA, Robinson-Rechavi M (May 2009). “How confident can we be that orthologs are similar, but paralogs differ?”. Trends in Genetics 25 (5): 210–6. doi:10.1016/j.tig.2009.03.004. PMID 19368988.
(99)^ Altenhoff AM, Studer RA, Robinson-Rechavi M, Dessimoz C (2012). “Resolving the ortholog conjecture: orthologs tend to be weakly, but significantly, more similar in function than paralogs”. PLOS Computational Biology 8 (5): e1002514. Bibcode: 2012PLSCB...8E2514A. doi:10.1371/journal.pcbi.1002514. PMC 3355068. PMID 22615551.
(56)^ Braig M, Schmitt CA (March 2006). “Oncogene-induced senescence: putting the brakes on tumor development”. Cancer Research 66 (6): 2881–4. doi:10.1158/0008-5472.CAN-05-4006. PMID 16540631.
(57)^ abBennett PM (March 2008). “Plasmid encoded antibiotic resistance: acquisition and transfer of antibiotic resistance genes in bacteria”. British Journal of Pharmacology 153 (Suppl 1): S347-57. doi:10.1038/sj.bjp.0707607. PMC 2268074. PMID 18193080.
(58)^ International Human Genome Sequencing Consortium (October 2004). “Finishing the euchromatic sequence of the human genome”. Nature 431 (7011): 931–45. Bibcode: 2004Natur.431..931H. doi:10.1038/nature03001. PMID 15496913.
(59)^ abShafee, Thomas; Lowe, Rohan (2017). “Eukaryotic and prokaryotic gene structure”. WikiJournal of Medicine 4 (1). doi:10.15347/wjm/2017.002. ISSN 2002-4436.
(60)^ Mortazavi A, Williams BA, McCue K, Schaeffer L, Wold B (July 2008). “Mapping and quantifying mammalian transcriptomes by RNA-Seq”. Nature Methods 5 (7): 621–8. doi:10.1038/nmeth.1226. PMID 18516045.
(61)^ Pennacchio LA, Bickmore W, Dean A, Nobrega MA, Bejerano G (April 2013). “Enhancers: five essential questions”. Nature Reviews. Genetics 14 (4): 288–95. doi:10.1038/nrg3458. PMC 4445073. PMID 23503198.
(62)^ Maston GA, Evans SK, Green MR (2006). “Transcriptional regulatory elements in the human genome”. Annual Review of Genomics and Human Genetics 7: 29–59. doi:10.1146/annurev.genom.7.080505.115623. PMID 16719718.
(63)^ Mignone F, Gissi C, Liuni S, Pesole G (2002-02-28). “Untranslated regions of mRNAs”. Genome Biology 3 (3): REVIEWS0004. doi:10.1186/gb-2002-3-3-reviews0004. PMC 139023. PMID 11897027.
(64)^ Bicknell AA, Cenik C, Chua HN, Roth FP, Moore MJ (December 2012). “Introns in UTRs: why we should stop ignoring them”. BioEssays 34 (12): 1025–34. doi:10.1002/bies.201200073. PMID 23108796.
(65)^ Shkurin A, Pour SE, Hughes TR (April 2023). “Known sequence features explain half of all human gene ends”. NAR Genomics and Bioinformatics 5 (2): lqad031. doi:10.1093/nargab/lqad031. PMC 10072996. PMID 37035540.
(66)^ Salgado H, Moreno-Hagelsieb G, Smith TF, Collado-Vides J (June 2000). “Operons in Escherichia coli: genomic analyses and predictions”. Proceedings of the National Academy of Sciences of the United States of America 97 (12): 6652–7. Bibcode: 2000PNAS...97.6652S. doi:10.1073/pnas.110147297. PMC 18690. PMID 10823905.
(67)^ Blumenthal T (November 2004). “Operons in eukaryotes”. Briefings in Functional Genomics & Proteomics 3 (3): 199–211. doi:10.1093/bfgp/3.3.199. PMID 15642184.
(68)^ Jacob F, Monod J (June 1961). “Genetic regulatory mechanisms in the synthesis of proteins”. Journal of Molecular Biology 3 (3): 318–56. doi:10.1016/S0022-2836(61)80072-7. PMID 13718526.
(69)^ Pozzoli U, Menozzi G, Comi GP, Cagliani R, Bresolin N, Sironi M (January 2007). “Intron size in mammals: complexity comes to terms with economy”. Trends in Genetics 23 (1): 20–24. doi:10.1016/j.tig.2006.10.003. PMID 17070957.
(70)^ Marais G, Nouvellet P, Keightley PD, Charlesworth B (May 2005). “Intron size and exon evolution in Drosophila”. Genetics 170 (1): 481–485. doi:10.1534/genetics.104.037333. PMC 1449718. PMID 15781704.
(71)^ Kumar A (September 2009). “An overview of nested genes in eukaryotic genomes”. Eukaryotic Cell 8 (9): 1321–1329. doi:10.1128/EC.00143-09. PMC 2747821. PMID 19542305..
(72)^ Spilianakis CG, Lalioti MD, Town T, Lee GR, Flavell RA (June 2005). “Interchromosomal associations between alternatively expressed loci”. Nature 435 (7042): 637–645. Bibcode: 2005Natur.435..637S. doi:10.1038/nature03574. PMID 15880101.
(73)^ Williams A, Spilianakis CG, Flavell RA (April 2010). “Interchromosomal association and gene regulation in trans”. Trends in Genetics 26 (4): 188–197. doi:10.1016/j.tig.2010.01.007. PMC 2865229. PMID 20236724.
(74)^ Lei Q, Li C, Zuo Z, Huang C, Cheng H, Zhou R (March 2016). “Evolutionary Insights into RNA trans-Splicing in Vertebrates”. Genome Biology and Evolution 8 (3): 562–577. doi:10.1093/gbe/evw025. PMC 4824033. PMID 26966239.
(75)^ Wright BW, Molloy MP, Jaschke PR (March 2022). “Overlapping genes in natural and engineered genomes”. Nature Reviews. Genetics 23 (3): 154–168. doi:10.1038/s41576-021-00417-w. PMC 8490965. PMID 34611352.
(76)^ abEddy SR (December 2001). “Non-coding RNA genes and the modern RNA world”. Nature Reviews. Genetics 2 (12): 919–29. doi:10.1038/35103511. PMID 11733745.
(77)^ Crick FH, Barnett L, Brenner S, Watts-Tobin RJ (December 1961). “General nature of the genetic code for proteins”. Nature 192 (4809): 1227–32. Bibcode: 1961Natur.192.1227C. doi:10.1038/1921227a0. PMID 13882203.
(78)^ Crick FH (October 1962). “The genetic code”. Scientific American (WH Freeman and Company) 207 (4): 66–74. Bibcode: 1962SciAm.207d..66C. doi:10.1038/scientificamerican1062-66. PMID 13882204.
(79)^ Woodson SA (May 1998). “Ironing out the kinks: splicing and translation in bacteria”. Genes & Development 12 (9): 1243–7. doi:10.1101/gad.12.9.1243. PMID 9573040.
(80)^ Jacob F, Monod J (June 1961). “Genetic regulatory mechanisms in the synthesis of proteins”. Journal of Molecular Biology 3 (3): 318–56. doi:10.1016/S0022-2836(61)80072-7. PMID 13718526.
(81)^ Koonin EV, Dolja VV (January 1993). “Evolution and taxonomy of positive-strand RNA viruses: implications of comparative analysis of amino acid sequences”. Critical Reviews in Biochemistry and Molecular Biology 28 (5): 375–430. doi:10.3109/10409239309078440. PMID 8269709.
(82)^ Domingo E (2001). “RNA Virus Genomes”. eLS. doi:10.1002/9780470015902.a0001488.pub2. ISBN 978-0470016176.
(83)^ Domingo E, Escarmís C, Sevilla N, Moya A, Elena SF, Quer J, Novella IS, Holland JJ (June 1996). “Basic concepts in RNA virus evolution”. FASEB Journal 10 (8): 859–64. doi:10.1096/fasebj.10.8.8666162. PMID 8666162.
(84)^ Miko I (2008). “Gregor Mendel and the Principles of Inheritance”. Nature Education Knowledge. SciTable (Nature Publishing Group) 1 (1): 134.
(85)^ Chial H (2008). “Mendelian Genetics: Patterns of Inheritance and Single-Gene Disorders”. Nature Education Knowledge. SciTable (Nature Publishing Group) 1 (1): 63.
(86)^ McCarthy D, Minner C, Bernstein H, Bernstein C (October 1976). “DNA elongation rates and growing point distributions of wild-type phage T4 and a DNA-delay amber mutant”. Journal of Molecular Biology 106 (4): 963–81. doi:10.1016/0022-2836(76)90346-6. PMID 789903.
(87)^ abLobo I, Shaw K (2008). “Discovery and Types of Genetic Linkage”. Nature Education Knowledge. SciTable (Nature Publishing Group) 1 (1): 139.
(88)^ Nachman MW, Crowell SL (September 2000). “Estimate of the mutation rate per nucleotide in humans”. Genetics 156 (1): 297–304. doi:10.1093/genetics/156.1.297. PMC 1461236. PMID 10978293.
(89)^ Roach JC, Glusman G, Smit AF, Huff CD, Hubley R, Shannon PT, Rowen L, Pant KP, Goodman N, Bamshad M, Shendure J, Drmanac R, Jorde LB, Hood L, Galas DJ (April 2010). “Analysis of genetic inheritance in a family quartet by whole-genome sequencing”. Science 328 (5978): 636–9. Bibcode: 2010Sci...328..636R. doi:10.1126/science.1186802. PMC 3037280. PMID 20220176.
(90)^ Drake JW, Charlesworth B, Charlesworth D, Crow JF (April 1998). “Rates of spontaneous mutation”. Genetics 148 (4): 1667–86. doi:10.1093/genetics/148.4.1667. PMC 1460098. PMID 9560386.
(91)^ Pyeritz, Reed E., Bruce R. Korf, and Wayne W. Grody, eds. Emery and Rimoin’s principles and practice of medical genetics and genomics: foundations. Academic Press, 2018.
(92)^ “What kinds of gene mutations are possible?”. Genetics Home Reference. United States National Library of Medicine (2015年5月11日). 2015年5月19日閲覧。
(93)^ Andrews CA (2010). “Natural Selection, Genetic Drift, and Gene Flow Do Not Act in Isolation in Natural Populations”. Nature Education Knowledge. SciTable (Nature Publishing Group) 3 (10): 5.
(94)^ Patterson C (November 1988). “Homology in classical and molecular biology”. Molecular Biology and Evolution 5 (6): 603–25. doi:10.1093/oxfordjournals.molbev.a040523. PMID 3065587.
(95)^ Graur D (2016). Molecular and Genome Evolution. Sunderland MA (US): Sinauer Associates, Inc.. ISBN 9781605354699
(96)^ Graur D (2016). Molecular and Genome Evolution. Sunderland MA (US): Sinauer Associates, Inc.. ISBN 9781605354699
(97)^ Jensen RA (2001). “Orthologs and paralogs - we need to get it right”. Genome Biology 2 (8): INTERACTIONS1002. doi:10.1186/gb-2001-2-8-interactions1002. PMC 138949. PMID 11532207.
(98)^ Studer RA, Robinson-Rechavi M (May 2009). “How confident can we be that orthologs are similar, but paralogs differ?”. Trends in Genetics 25 (5): 210–6. doi:10.1016/j.tig.2009.03.004. PMID 19368988.
(99)^ Altenhoff AM, Studer RA, Robinson-Rechavi M, Dessimoz C (2012). “Resolving the ortholog conjecture: orthologs tend to be weakly, but significantly, more similar in function than paralogs”. PLOS Computational Biology 8 (5): e1002514. Bibcode: 2012PLSCB...8E2514A. doi:10.1371/journal.pcbi.1002514. PMC 3355068. PMID 22615551.  (100)^ abGuerzoni D, McLysaght A (November 2011). “De novo origins of human genes”. PLOS Genetics 7 (11): e1002381. doi:10.1371/journal.pgen.1002381. PMC 3213182. PMID 22102832.
(100)^ abGuerzoni D, McLysaght A (November 2011). “De novo origins of human genes”. PLOS Genetics 7 (11): e1002381. doi:10.1371/journal.pgen.1002381. PMC 3213182. PMID 22102832.  (101)^ Reams AB, Roth JR (February 2015). “Mechanisms of gene duplication and amplification”. Cold Spring Harbor Perspectives in Biology 7 (2): a016592. doi:10.1101/cshperspect.a016592. PMC 4315931. PMID 25646380.
(102)^ Demuth JP, De Bie T, Stajich JE, Cristianini N, Hahn MW (December 2006). “The evolution of mammalian gene families”. PLOS ONE 1 (1): e85. Bibcode: 2006PLoSO...1...85D. doi:10.1371/journal.pone.0000085. PMC 1762380. PMID 17183716.
(101)^ Reams AB, Roth JR (February 2015). “Mechanisms of gene duplication and amplification”. Cold Spring Harbor Perspectives in Biology 7 (2): a016592. doi:10.1101/cshperspect.a016592. PMC 4315931. PMID 25646380.
(102)^ Demuth JP, De Bie T, Stajich JE, Cristianini N, Hahn MW (December 2006). “The evolution of mammalian gene families”. PLOS ONE 1 (1): e85. Bibcode: 2006PLoSO...1...85D. doi:10.1371/journal.pone.0000085. PMC 1762380. PMID 17183716.  (103)^ Knowles DG, McLysaght A (October 2009). “Recent de novo origin of human protein-coding genes”. Genome Research 19 (10): 1752–9. doi:10.1101/gr.095026.109. PMC 2765279. PMID 19726446.
(104)^ Wu DD, Irwin DM, Zhang YP (November 2011). “De novo origin of human protein-coding genes”. PLOS Genetics 7 (11): e1002379. doi:10.1371/journal.pgen.1002379. PMC 3213175. PMID 22102831.
(103)^ Knowles DG, McLysaght A (October 2009). “Recent de novo origin of human protein-coding genes”. Genome Research 19 (10): 1752–9. doi:10.1101/gr.095026.109. PMC 2765279. PMID 19726446.
(104)^ Wu DD, Irwin DM, Zhang YP (November 2011). “De novo origin of human protein-coding genes”. PLOS Genetics 7 (11): e1002379. doi:10.1371/journal.pgen.1002379. PMC 3213175. PMID 22102831.  (105)^ McLysaght A, Guerzoni D (September 2015). “New genes from non-coding sequence: the role of de novo protein-coding genes in eukaryotic evolutionary innovation”. Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences 370 (1678): 20140332. doi:10.1098/rstb.2014.0332. PMC 4571571. PMID 26323763.
(106)^ Neme R, Tautz D (February 2013). “Phylogenetic patterns of emergence of new genes support a model of frequent de novo evolution”. BMC Genomics 14 (1): 117. doi:10.1186/1471-2164-14-117. PMC 3616865. PMID 23433480.
(107)^ Treangen TJ, Rocha EP (January 2011). “Horizontal transfer, not duplication, drives the expansion of protein families in prokaryotes”. PLOS Genetics 7 (1): e1001284. doi:10.1371/journal.pgen.1001284. PMC 3029252. PMID 21298028.
(105)^ McLysaght A, Guerzoni D (September 2015). “New genes from non-coding sequence: the role of de novo protein-coding genes in eukaryotic evolutionary innovation”. Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences 370 (1678): 20140332. doi:10.1098/rstb.2014.0332. PMC 4571571. PMID 26323763.
(106)^ Neme R, Tautz D (February 2013). “Phylogenetic patterns of emergence of new genes support a model of frequent de novo evolution”. BMC Genomics 14 (1): 117. doi:10.1186/1471-2164-14-117. PMC 3616865. PMID 23433480.
(107)^ Treangen TJ, Rocha EP (January 2011). “Horizontal transfer, not duplication, drives the expansion of protein families in prokaryotes”. PLOS Genetics 7 (1): e1001284. doi:10.1371/journal.pgen.1001284. PMC 3029252. PMID 21298028.  (108)^ Ochman H, Lawrence JG, Groisman EA (May 2000). “Lateral gene transfer and the nature of bacterial innovation”. Nature 405 (6784): 299–304. Bibcode: 2000Natur.405..299O. doi:10.1038/35012500. PMID 10830951.
(109)^ Keeling PJ, Palmer JD (August 2008). “Horizontal gene transfer in eukaryotic evolution”. Nature Reviews. Genetics 9 (8): 605–18. doi:10.1038/nrg2386. PMID 18591983.
(110)^ Schönknecht G, Chen WH, Ternes CM, Barbier GG, Shrestha RP, Stanke M, Bräutigam A, Baker BJ, Banfield JF, Garavito RM, Carr K, Wilkerson C, Rensing SA, Gagneul D, Dickenson NE, Oesterhelt C, Lercher MJ, Weber AP (March 2013). “Gene transfer from bacteria and archaea facilitated evolution of an extremophilic eukaryote”. Science 339 (6124): 1207–10. Bibcode: 2013Sci...339.1207S. doi:10.1126/science.1231707. PMID 23471408.
(111)^ Ridley, M. (2006). Genome. New York, NY: Harper Perennial. ISBN 0-06-019497-9
(112)^ Banerjee S, Bhandary P, Woodhouse M, Sen TZ, Wise RP, Andorf CM (Apr 2021). “FINDER: an automated software package to annotate eukaryotic genes from RNA-Seq data and associated protein sequences”. BMC Bioinformatics 44 (9): e89. doi:10.1186/s12859-021-04120-9. PMC 8056616. PMID 33879057.
(113)^ Watson, JD, Baker TA, Bell SP, Gann A, Levine M, Losick R. (2004). "Ch9-10", Molecular Biology of the Gene, 5th ed., Peason Benjamin Cummings; CSHL Press.
(114)^ “Integr8 – A.thaliana Genome Statistics”. 2015年4月26日閲覧。
(115)^ “Understanding the Basics”. The Human Genome Project. 2015年4月26日閲覧。
(116)^ “WS227 Release Letter”. WormBase (2011年8月10日). 2013年11月28日時点のオリジナルよりアーカイブ。2013年11月19日閲覧。
(117)^ abYu J, Hu S, Wang J, Wong GK, Li S, Liu B, Deng Y, Dai L, Zhou Y, Zhang X, Cao M, Liu J, Sun J, Tang J, Chen Y, Huang X, Lin W, Ye C, Tong W, Cong L, Geng J, Han Y, Li L, Li W, Hu G, Huang X, Li W, Li J, Liu Z, Li L, Liu J, Qi Q, Liu J, Li L, Li T, Wang X, Lu H, Wu T, Zhu M, Ni P, Han H, Dong W, Ren X, Feng X, Cui P, Li X, Wang H, Xu X, Zhai W, Xu Z, Zhang J, He S, Zhang J, Xu J, Zhang K, Zheng X, Dong J, Zeng W, Tao L, Ye J, Tan J, Ren X, Chen X, He J, Liu D, Tian W, Tian C, Xia H, Bao Q, Li G, Gao H, Cao T, Wang J, Zhao W, Li P, Chen W, Wang X, Zhang Y, Hu J, Wang J, Liu S, Yang J, Zhang G, Xiong Y, Li Z, Mao L, Zhou C, Zhu Z, Chen R, Hao B, Zheng W, Chen S, Guo W, Li G, Liu S, Tao M, Wang J, Zhu L, Yuan L, Yang H (April 2002). “A draft sequence of the rice genome (Oryza sativa L. ssp. indica)”. Science 296 (5565): 79–92. Bibcode: 2002Sci...296...79Y. doi:10.1126/science.1068037. PMID 11935017.
(118)^ Anderson S, Bankier AT, Barrell BG, de Bruijn MH, Coulson AR, Drouin J, Eperon IC, Nierlich DP, Roe BA, Sanger F, Schreier PH, Smith AJ, Staden R, Young IG (April 1981). “Sequence and organization of the human mitochondrial genome”. Nature 290 (5806): 457–65. Bibcode: 1981Natur.290..457A. doi:10.1038/290457a0. PMID 7219534.
(119)^ Adams MD, Celniker SE, Holt RA, Evans CA, Gocayne JD, Amanatides PG, Scherer SE, Li PW, Hoskins RA, Galle RF, George RA, Lewis SE, Richards S, Ashburner M, Henderson SN, Sutton GG, Wortman JR, Yandell MD, Zhang Q, Chen LX, Brandon RC, Rogers YH, Blazej RG, Champe M, Pfeiffer BD, Wan KH, Doyle C, Baxter EG, Helt G, Nelson CR, Gabor GL, Abril JF, Agbayani A, An HJ, Andrews-Pfannkoch C, Baldwin D, Ballew RM, Basu A, Baxendale J, Bayraktaroglu L, Beasley EM, Beeson KY, Benos PV, Berman BP, Bhandari D, Bolshakov S, Borkova D, Botchan MR, Bouck J, Brokstein P, Brottier P, Burtis KC, Busam DA, Butler H, Cadieu E, Center A, Chandra I, Cherry JM, Cawley S, Dahlke C, Davenport LB, Davies P, de Pablos B, Delcher A, Deng Z, Mays AD, Dew I, Dietz SM, Dodson K, Doup LE, Downes M, Dugan-Rocha S, Dunkov BC, Dunn P, Durbin KJ, Evangelista CC, Ferraz C, Ferriera S, Fleischmann W, Fosler C, Gabrielian AE, Garg NS, Gelbart WM, Glasser K, Glodek A, Gong F, Gorrell JH, Gu Z, Guan P, Harris M, Harris NL, Harvey D, Heiman TJ, Hernandez JR, Houck J, Hostin D, Houston KA, Howland TJ, Wei MH, Ibegwam C, Jalali M, Kalush F, Karpen GH, Ke Z, Kennison JA, Ketchum KA, Kimmel BE, Kodira CD, Kraft C, Kravitz S, Kulp D, Lai Z, Lasko P, Lei Y, Levitsky AA, Li J, Li Z, Liang Y, Lin X, Liu X, Mattei B, McIntosh TC, McLeod MP, McPherson D, Merkulov G, Milshina NV, Mobarry C, Morris J, Moshrefi A, Mount SM, Moy M, Murphy B, Murphy L, Muzny DM, Nelson DL, Nelson DR, Nelson KA, Nixon K, Nusskern DR, Pacleb JM, Palazzolo M, Pittman GS, Pan S, Pollard J, Puri V, Reese MG, Reinert K, Remington K, Saunders RD, Scheeler F, Shen H, Shue BC, Sidén-Kiamos I, Simpson M, Skupski MP, Smith T, Spier E, Spradling AC, Stapleton M, Strong R, Sun E, Svirskas R, Tector C, Turner R, Venter E, Wang AH, Wang X, Wang ZY, Wassarman DA, Weinstock GM, Weissenbach J, Williams SM, Worley KC, Wu D, Yang S, Yao QA, Ye J, Yeh RF, Zaveri JS, Zhan M, Zhang G, Zhao Q, Zheng L, Zheng XH, Zhong FN, Zhong W, Zhou X, Zhu S, Zhu X, Smith HO, Gibbs RA, Myers EW, Rubin GM, Venter JC (March 2000). “The genome sequence of Drosophila melanogaster”. Science 287 (5461): 2185–95. Bibcode: 2000Sci...287.2185.. doi:10.1126/science.287.5461.2185. PMID 10731132.
(120)^ Pertea M, Salzberg SL (2010). “Between a chicken and a grape: estimating the number of human genes”. Genome Biology 11 (5): 206. doi:10.1186/gb-2010-11-5-206. PMC 2898077. PMID 20441615.
(121)^ Belyi VA, Levine AJ, Skalka AM (December 2010). “Sequences from ancestral single-stranded DNA viruses in vertebrate genomes: the parvoviridae and circoviridae are more than 40 to 50 million years old”. Journal of Virology 84 (23): 12458–62. doi:10.1128/JVI.01789-10. PMC 2976387. PMID 20861255.
(122)^ Flores R, Di Serio F, Hernández C (February 1997). “Viroids: The Noncoding Genomes”. Seminars in Virology 8 (1): 65–73. doi:10.1006/smvy.1997.0107.
(123)^ Zonneveld BJ (2010). “New Record Holders for Maximum Genome Size in Eudicots and Monocots”. Journal of Botany 2010: 1–4. doi:10.1155/2010/527357.
(124)^ Perez-Iratxeta C, Palidwor G, Andrade-Navarro MA (December 2007). “Towards completion of the Earth's proteome”. EMBO Reports 8 (12): 1135–41. doi:10.1038/sj.embor.7401117. PMC 2267224. PMID 18059312.
(125)^ Muller HJ (1966). “The gene material as the initiator and the organizing basis of life”. American Naturalist 100 (915): 493–517. doi:10.1086/282445. JSTOR 2459205.
(126)^ Ohno S (1972). “So much "junk" DNA in our genome”. Brookhaven Symposia in Biology 23: 366–370. PMID 5065367.
(127)^ Hatje K, Mühlhausen S, Simm D, Killmar M (2019). “The Protein-Coding Human Genome: Annotating High-Hanging Fruits.”. BioEssays 41 (11): 1900066. doi:10.1002/bies.201900066. PMID 31544971.
(128)^ Schuler GD, Boguski MS, Stewart EA, Stein LD, Gyapay G, Rice K, White RE, Rodriguez-Tomé P, Aggarwal A, Bajorek E, Bentolila S, Birren BB, Butler A, Castle AB, Chiannilkulchai N, Chu A, Clee C, Cowles S, Day PJ, Dibling T, Drouot N, Dunham I, Duprat S, East C, Edwards C, Fan JB, Fang N, Fizames C, Garrett C, Green L, Hadley D, Harris M, Harrison P, Brady S, Hicks A, Holloway E, Hui L, Hussain S, Louis-Dit-Sully C, Ma J, MacGilvery A, Mader C, Maratukulam A, Matise TC, McKusick KB, Morissette J, Mungall A, Muselet D, Nusbaum HC, Page DC, Peck A, Perkins S, Piercy M, Qin F, Quackenbush J, Ranby S, Reif T, Rozen S, Sanders C, She X, Silva J, Slonim DK, Soderlund C, Sun WL, Tabar P, Thangarajah T, Vega-Czarny N, Vollrath D, Voyticky S, Wilmer T, Wu X, Adams MD, Auffray C, Walter NA, Brandon R, Dehejia A, Goodfellow PN, Houlgatte R, Hudson JR, Ide SE, Iorio KR, Lee WY, Seki N, Nagase T, Ishikawa K, Nomura N, Phillips C, Polymeropoulos MH, Sandusky M, Schmitt K, Berry R, Swanson K, Torres R, Venter JC, Sikela JM, Beckmann JS, Weissenbach J, Myers RM, Cox DR, James MR, Bentley D, Deloukas P, Lander ES, Hudson TJ (October 1996). “A gene map of the human genome”. Science 274 (5287): 540–6. Bibcode: 1996Sci...274..540S. doi:10.1126/science.274.5287.540. PMID 8849440.
(129)^ Chi KR (October 2016). “The dark side of the human genome” (英語). Nature 538 (7624): 275–277. Bibcode: 2016Natur.538..275C. doi:10.1038/538275a. PMID 27734873.
(130)^ Ezkurdia, Iakes; Juan, David; Rodriguez, Jose Manuel; Frankish, Adam; Diekhans, Mark; Harrow, Jennifer; Vazquez, Jesus; Valencia, Alfonso et al. (2014-11-15). “Multiple evidence strands suggest that there may be as few as 19 000 human protein-coding genes” (英語). Human Molecular Genetics 23 (22): 5866–5878. doi:10.1093/hmg/ddu309. ISSN 0964-6906. PMC PMC4204768. PMID 24939910.
(131)^ “Human assembly and gene annotation”. Ensembl (2022年). 2023年2月28日閲覧。
(132)^ abHutchison CA, Chuang RY, Noskov VN, Assad-Garcia N, Deerinck TJ, Ellisman MH, Gill J, Kannan K, Karas BJ, Ma L, Pelletier JF, Qi ZQ, Richter RA, Strychalski EA, Sun L, Suzuki Y, Tsvetanova B, Wise KS, Smith HO, Glass JI, Merryman C, Gibson DG, Venter JC (March 2016). “Design and synthesis of a minimal bacterial genome”. Science 351 (6280): aad6253. Bibcode: 2016Sci...351.....H. doi:10.1126/science.aad6253. PMID 27013737.
(133)^ Glass JI, Assad-Garcia N, Alperovich N, Yooseph S, Lewis MR, Maruf M, Hutchison CA, Smith HO, Venter JC (January 2006). “Essential genes of a minimal bacterium”. Proceedings of the National Academy of Sciences of the United States of America 103 (2): 425–30. Bibcode: 2006PNAS..103..425G. doi:10.1073/pnas.0510013103. PMC 1324956. PMID 16407165.
(134)^ Gerdes SY, Scholle MD, Campbell JW, Balázsi G, Ravasz E, Daugherty MD, Somera AL, Kyrpides NC, Anderson I, Gelfand MS, Bhattacharya A, Kapatral V, D'Souza M, Baev MV, Grechkin Y, Mseeh F, Fonstein MY, Overbeek R, Barabási AL, Oltvai ZN, Osterman AL (October 2003). “Experimental determination and system level analysis of essential genes in Escherichia coli MG1655”. Journal of Bacteriology 185 (19): 5673–84. doi:10.1128/jb.185.19.5673-5684.2003. PMC 193955. PMID 13129938.
(135)^ Baba T, Ara T, Hasegawa M, Takai Y, Okumura Y, Baba M, Datsenko KA, Tomita M, Wanner BL, Mori H (2006). “Construction of Escherichia coli K-12 in-frame, single-gene knockout mutants: the Keio collection”. Molecular Systems Biology 2: 2006.0008. doi:10.1038/msb4100050. PMC 1681482. PMID 16738554.
(136)^ abJuhas M, Reuß DR, Zhu B, Commichau FM (November 2014). “Bacillus subtilis and Escherichia coli essential genes and minimal cell factories after one decade of genome engineering”. Microbiology 160 (Pt 11): 2341–2351. doi:10.1099/mic.0.079376-0. PMID 25092907.
(137)^ Tu Z, Wang L, Xu M, Zhou X, Chen T, Sun F (February 2006). “Further understanding human disease genes by comparing with housekeeping genes and other genes”. BMC Genomics 7: 31. doi:10.1186/1471-2164-7-31. PMC 1397819. PMID 16504025.
(108)^ Ochman H, Lawrence JG, Groisman EA (May 2000). “Lateral gene transfer and the nature of bacterial innovation”. Nature 405 (6784): 299–304. Bibcode: 2000Natur.405..299O. doi:10.1038/35012500. PMID 10830951.
(109)^ Keeling PJ, Palmer JD (August 2008). “Horizontal gene transfer in eukaryotic evolution”. Nature Reviews. Genetics 9 (8): 605–18. doi:10.1038/nrg2386. PMID 18591983.
(110)^ Schönknecht G, Chen WH, Ternes CM, Barbier GG, Shrestha RP, Stanke M, Bräutigam A, Baker BJ, Banfield JF, Garavito RM, Carr K, Wilkerson C, Rensing SA, Gagneul D, Dickenson NE, Oesterhelt C, Lercher MJ, Weber AP (March 2013). “Gene transfer from bacteria and archaea facilitated evolution of an extremophilic eukaryote”. Science 339 (6124): 1207–10. Bibcode: 2013Sci...339.1207S. doi:10.1126/science.1231707. PMID 23471408.
(111)^ Ridley, M. (2006). Genome. New York, NY: Harper Perennial. ISBN 0-06-019497-9
(112)^ Banerjee S, Bhandary P, Woodhouse M, Sen TZ, Wise RP, Andorf CM (Apr 2021). “FINDER: an automated software package to annotate eukaryotic genes from RNA-Seq data and associated protein sequences”. BMC Bioinformatics 44 (9): e89. doi:10.1186/s12859-021-04120-9. PMC 8056616. PMID 33879057.
(113)^ Watson, JD, Baker TA, Bell SP, Gann A, Levine M, Losick R. (2004). "Ch9-10", Molecular Biology of the Gene, 5th ed., Peason Benjamin Cummings; CSHL Press.
(114)^ “Integr8 – A.thaliana Genome Statistics”. 2015年4月26日閲覧。
(115)^ “Understanding the Basics”. The Human Genome Project. 2015年4月26日閲覧。
(116)^ “WS227 Release Letter”. WormBase (2011年8月10日). 2013年11月28日時点のオリジナルよりアーカイブ。2013年11月19日閲覧。
(117)^ abYu J, Hu S, Wang J, Wong GK, Li S, Liu B, Deng Y, Dai L, Zhou Y, Zhang X, Cao M, Liu J, Sun J, Tang J, Chen Y, Huang X, Lin W, Ye C, Tong W, Cong L, Geng J, Han Y, Li L, Li W, Hu G, Huang X, Li W, Li J, Liu Z, Li L, Liu J, Qi Q, Liu J, Li L, Li T, Wang X, Lu H, Wu T, Zhu M, Ni P, Han H, Dong W, Ren X, Feng X, Cui P, Li X, Wang H, Xu X, Zhai W, Xu Z, Zhang J, He S, Zhang J, Xu J, Zhang K, Zheng X, Dong J, Zeng W, Tao L, Ye J, Tan J, Ren X, Chen X, He J, Liu D, Tian W, Tian C, Xia H, Bao Q, Li G, Gao H, Cao T, Wang J, Zhao W, Li P, Chen W, Wang X, Zhang Y, Hu J, Wang J, Liu S, Yang J, Zhang G, Xiong Y, Li Z, Mao L, Zhou C, Zhu Z, Chen R, Hao B, Zheng W, Chen S, Guo W, Li G, Liu S, Tao M, Wang J, Zhu L, Yuan L, Yang H (April 2002). “A draft sequence of the rice genome (Oryza sativa L. ssp. indica)”. Science 296 (5565): 79–92. Bibcode: 2002Sci...296...79Y. doi:10.1126/science.1068037. PMID 11935017.
(118)^ Anderson S, Bankier AT, Barrell BG, de Bruijn MH, Coulson AR, Drouin J, Eperon IC, Nierlich DP, Roe BA, Sanger F, Schreier PH, Smith AJ, Staden R, Young IG (April 1981). “Sequence and organization of the human mitochondrial genome”. Nature 290 (5806): 457–65. Bibcode: 1981Natur.290..457A. doi:10.1038/290457a0. PMID 7219534.
(119)^ Adams MD, Celniker SE, Holt RA, Evans CA, Gocayne JD, Amanatides PG, Scherer SE, Li PW, Hoskins RA, Galle RF, George RA, Lewis SE, Richards S, Ashburner M, Henderson SN, Sutton GG, Wortman JR, Yandell MD, Zhang Q, Chen LX, Brandon RC, Rogers YH, Blazej RG, Champe M, Pfeiffer BD, Wan KH, Doyle C, Baxter EG, Helt G, Nelson CR, Gabor GL, Abril JF, Agbayani A, An HJ, Andrews-Pfannkoch C, Baldwin D, Ballew RM, Basu A, Baxendale J, Bayraktaroglu L, Beasley EM, Beeson KY, Benos PV, Berman BP, Bhandari D, Bolshakov S, Borkova D, Botchan MR, Bouck J, Brokstein P, Brottier P, Burtis KC, Busam DA, Butler H, Cadieu E, Center A, Chandra I, Cherry JM, Cawley S, Dahlke C, Davenport LB, Davies P, de Pablos B, Delcher A, Deng Z, Mays AD, Dew I, Dietz SM, Dodson K, Doup LE, Downes M, Dugan-Rocha S, Dunkov BC, Dunn P, Durbin KJ, Evangelista CC, Ferraz C, Ferriera S, Fleischmann W, Fosler C, Gabrielian AE, Garg NS, Gelbart WM, Glasser K, Glodek A, Gong F, Gorrell JH, Gu Z, Guan P, Harris M, Harris NL, Harvey D, Heiman TJ, Hernandez JR, Houck J, Hostin D, Houston KA, Howland TJ, Wei MH, Ibegwam C, Jalali M, Kalush F, Karpen GH, Ke Z, Kennison JA, Ketchum KA, Kimmel BE, Kodira CD, Kraft C, Kravitz S, Kulp D, Lai Z, Lasko P, Lei Y, Levitsky AA, Li J, Li Z, Liang Y, Lin X, Liu X, Mattei B, McIntosh TC, McLeod MP, McPherson D, Merkulov G, Milshina NV, Mobarry C, Morris J, Moshrefi A, Mount SM, Moy M, Murphy B, Murphy L, Muzny DM, Nelson DL, Nelson DR, Nelson KA, Nixon K, Nusskern DR, Pacleb JM, Palazzolo M, Pittman GS, Pan S, Pollard J, Puri V, Reese MG, Reinert K, Remington K, Saunders RD, Scheeler F, Shen H, Shue BC, Sidén-Kiamos I, Simpson M, Skupski MP, Smith T, Spier E, Spradling AC, Stapleton M, Strong R, Sun E, Svirskas R, Tector C, Turner R, Venter E, Wang AH, Wang X, Wang ZY, Wassarman DA, Weinstock GM, Weissenbach J, Williams SM, Worley KC, Wu D, Yang S, Yao QA, Ye J, Yeh RF, Zaveri JS, Zhan M, Zhang G, Zhao Q, Zheng L, Zheng XH, Zhong FN, Zhong W, Zhou X, Zhu S, Zhu X, Smith HO, Gibbs RA, Myers EW, Rubin GM, Venter JC (March 2000). “The genome sequence of Drosophila melanogaster”. Science 287 (5461): 2185–95. Bibcode: 2000Sci...287.2185.. doi:10.1126/science.287.5461.2185. PMID 10731132.
(120)^ Pertea M, Salzberg SL (2010). “Between a chicken and a grape: estimating the number of human genes”. Genome Biology 11 (5): 206. doi:10.1186/gb-2010-11-5-206. PMC 2898077. PMID 20441615.
(121)^ Belyi VA, Levine AJ, Skalka AM (December 2010). “Sequences from ancestral single-stranded DNA viruses in vertebrate genomes: the parvoviridae and circoviridae are more than 40 to 50 million years old”. Journal of Virology 84 (23): 12458–62. doi:10.1128/JVI.01789-10. PMC 2976387. PMID 20861255.
(122)^ Flores R, Di Serio F, Hernández C (February 1997). “Viroids: The Noncoding Genomes”. Seminars in Virology 8 (1): 65–73. doi:10.1006/smvy.1997.0107.
(123)^ Zonneveld BJ (2010). “New Record Holders for Maximum Genome Size in Eudicots and Monocots”. Journal of Botany 2010: 1–4. doi:10.1155/2010/527357.
(124)^ Perez-Iratxeta C, Palidwor G, Andrade-Navarro MA (December 2007). “Towards completion of the Earth's proteome”. EMBO Reports 8 (12): 1135–41. doi:10.1038/sj.embor.7401117. PMC 2267224. PMID 18059312.
(125)^ Muller HJ (1966). “The gene material as the initiator and the organizing basis of life”. American Naturalist 100 (915): 493–517. doi:10.1086/282445. JSTOR 2459205.
(126)^ Ohno S (1972). “So much "junk" DNA in our genome”. Brookhaven Symposia in Biology 23: 366–370. PMID 5065367.
(127)^ Hatje K, Mühlhausen S, Simm D, Killmar M (2019). “The Protein-Coding Human Genome: Annotating High-Hanging Fruits.”. BioEssays 41 (11): 1900066. doi:10.1002/bies.201900066. PMID 31544971.
(128)^ Schuler GD, Boguski MS, Stewart EA, Stein LD, Gyapay G, Rice K, White RE, Rodriguez-Tomé P, Aggarwal A, Bajorek E, Bentolila S, Birren BB, Butler A, Castle AB, Chiannilkulchai N, Chu A, Clee C, Cowles S, Day PJ, Dibling T, Drouot N, Dunham I, Duprat S, East C, Edwards C, Fan JB, Fang N, Fizames C, Garrett C, Green L, Hadley D, Harris M, Harrison P, Brady S, Hicks A, Holloway E, Hui L, Hussain S, Louis-Dit-Sully C, Ma J, MacGilvery A, Mader C, Maratukulam A, Matise TC, McKusick KB, Morissette J, Mungall A, Muselet D, Nusbaum HC, Page DC, Peck A, Perkins S, Piercy M, Qin F, Quackenbush J, Ranby S, Reif T, Rozen S, Sanders C, She X, Silva J, Slonim DK, Soderlund C, Sun WL, Tabar P, Thangarajah T, Vega-Czarny N, Vollrath D, Voyticky S, Wilmer T, Wu X, Adams MD, Auffray C, Walter NA, Brandon R, Dehejia A, Goodfellow PN, Houlgatte R, Hudson JR, Ide SE, Iorio KR, Lee WY, Seki N, Nagase T, Ishikawa K, Nomura N, Phillips C, Polymeropoulos MH, Sandusky M, Schmitt K, Berry R, Swanson K, Torres R, Venter JC, Sikela JM, Beckmann JS, Weissenbach J, Myers RM, Cox DR, James MR, Bentley D, Deloukas P, Lander ES, Hudson TJ (October 1996). “A gene map of the human genome”. Science 274 (5287): 540–6. Bibcode: 1996Sci...274..540S. doi:10.1126/science.274.5287.540. PMID 8849440.
(129)^ Chi KR (October 2016). “The dark side of the human genome” (英語). Nature 538 (7624): 275–277. Bibcode: 2016Natur.538..275C. doi:10.1038/538275a. PMID 27734873.
(130)^ Ezkurdia, Iakes; Juan, David; Rodriguez, Jose Manuel; Frankish, Adam; Diekhans, Mark; Harrow, Jennifer; Vazquez, Jesus; Valencia, Alfonso et al. (2014-11-15). “Multiple evidence strands suggest that there may be as few as 19 000 human protein-coding genes” (英語). Human Molecular Genetics 23 (22): 5866–5878. doi:10.1093/hmg/ddu309. ISSN 0964-6906. PMC PMC4204768. PMID 24939910.
(131)^ “Human assembly and gene annotation”. Ensembl (2022年). 2023年2月28日閲覧。
(132)^ abHutchison CA, Chuang RY, Noskov VN, Assad-Garcia N, Deerinck TJ, Ellisman MH, Gill J, Kannan K, Karas BJ, Ma L, Pelletier JF, Qi ZQ, Richter RA, Strychalski EA, Sun L, Suzuki Y, Tsvetanova B, Wise KS, Smith HO, Glass JI, Merryman C, Gibson DG, Venter JC (March 2016). “Design and synthesis of a minimal bacterial genome”. Science 351 (6280): aad6253. Bibcode: 2016Sci...351.....H. doi:10.1126/science.aad6253. PMID 27013737.
(133)^ Glass JI, Assad-Garcia N, Alperovich N, Yooseph S, Lewis MR, Maruf M, Hutchison CA, Smith HO, Venter JC (January 2006). “Essential genes of a minimal bacterium”. Proceedings of the National Academy of Sciences of the United States of America 103 (2): 425–30. Bibcode: 2006PNAS..103..425G. doi:10.1073/pnas.0510013103. PMC 1324956. PMID 16407165.
(134)^ Gerdes SY, Scholle MD, Campbell JW, Balázsi G, Ravasz E, Daugherty MD, Somera AL, Kyrpides NC, Anderson I, Gelfand MS, Bhattacharya A, Kapatral V, D'Souza M, Baev MV, Grechkin Y, Mseeh F, Fonstein MY, Overbeek R, Barabási AL, Oltvai ZN, Osterman AL (October 2003). “Experimental determination and system level analysis of essential genes in Escherichia coli MG1655”. Journal of Bacteriology 185 (19): 5673–84. doi:10.1128/jb.185.19.5673-5684.2003. PMC 193955. PMID 13129938.
(135)^ Baba T, Ara T, Hasegawa M, Takai Y, Okumura Y, Baba M, Datsenko KA, Tomita M, Wanner BL, Mori H (2006). “Construction of Escherichia coli K-12 in-frame, single-gene knockout mutants: the Keio collection”. Molecular Systems Biology 2: 2006.0008. doi:10.1038/msb4100050. PMC 1681482. PMID 16738554.
(136)^ abJuhas M, Reuß DR, Zhu B, Commichau FM (November 2014). “Bacillus subtilis and Escherichia coli essential genes and minimal cell factories after one decade of genome engineering”. Microbiology 160 (Pt 11): 2341–2351. doi:10.1099/mic.0.079376-0. PMID 25092907.
(137)^ Tu Z, Wang L, Xu M, Zhou X, Chen T, Sun F (February 2006). “Further understanding human disease genes by comparing with housekeeping genes and other genes”. BMC Genomics 7: 31. doi:10.1186/1471-2164-7-31. PMC 1397819. PMID 16504025.  (138)^ Georgi B, Voight BF, Bućan M (May 2013). “From mouse to human: evolutionary genomics analysis of human orthologs of essential genes”. PLOS Genetics 9 (5): e1003484. doi:10.1371/journal.pgen.1003484. PMC 3649967. PMID 23675308.
(138)^ Georgi B, Voight BF, Bućan M (May 2013). “From mouse to human: evolutionary genomics analysis of human orthologs of essential genes”. PLOS Genetics 9 (5): e1003484. doi:10.1371/journal.pgen.1003484. PMC 3649967. PMID 23675308.  (139)^ Eisenberg E, Levanon EY (October 2013). “Human housekeeping genes, revisited”. Trends in Genetics 29 (10): 569–74. doi:10.1016/j.tig.2013.05.010. PMID 23810203.
(140)^ Amsterdam A, Hopkins N (September 2006). “Mutagenesis strategies in zebrafish for identifying genes involved in development and disease”. Trends in Genetics 22 (9): 473–8. doi:10.1016/j.tig.2006.06.011. PMID 16844256.
(141)^ “About the HGNC”. HGNC Database of Human Gene Names. HUGO Gene Nomenclature Committee. 2015年5月14日閲覧。
(142)^ Cohen SN, Chang AC (May 1973). “Recircularization and autonomous replication of a sheared R-factor DNA segment in Escherichia coli transformants”. Proceedings of the National Academy of Sciences of the United States of America 70 (5): 1293–7. Bibcode: 1973PNAS...70.1293C. doi:10.1073/pnas.70.5.1293. PMC 433482. PMID 4576014.
(143)^ Esvelt KM, Wang HH (2013). “Genome-scale engineering for systems and synthetic biology”. Molecular Systems Biology 9 (1): 641. doi:10.1038/msb.2012.66. PMC 3564264. PMID 23340847.
(144)^ Tan WS, Carlson DF, Walton MW, Fahrenkrug SC, Hackett PB (2012). “Precision editing of large animal genomes”. Advances in Genetics Volume 80. 80. pp. 37–97. doi:10.1016/B978-0-12-404742-6.00002-8. ISBN 9780124047426. PMC 3683964. PMID 23084873
(145)^ Puchta H, Fauser F (2013). “Gene targeting in plants: 25 years later”. The International Journal of Developmental Biology 57 (6–8): 629–37. doi:10.1387/ijdb.130194hp. PMID 24166445.
(146)^ Ran FA, Hsu PD, Wright J, Agarwala V, Scott DA, Zhang F (November 2013). “Genome engineering using the CRISPR-Cas9 system”. Nature Protocols 8 (11): 2281–2308. doi:10.1038/nprot.2013.143. PMC 3969860. PMID 24157548.
(147)^ Kittleson JT, Wu GC, Anderson JC (August 2012). “Successes and failures in modular genetic engineering”. Current Opinion in Chemical Biology 16 (3–4): 329–36. doi:10.1016/j.cbpa.2012.06.009. PMID 22818777.
(148)^ Berg P, Mertz JE (January 2010). “Personal reflections on the origins and emergence of recombinant DNA technology”. Genetics 184 (1): 9–17. doi:10.1534/genetics.109.112144. PMC 2815933. PMID 20061565.
(149)^ Guan C, Ye C, Yang X, Gao J (February 2010). “A review of current large-scale mouse knockout efforts”. Genesis 48 (2): 73–85. doi:10.1002/dvg.20594. PMID 20095055.
(150)^ Austin CP, Battey JF, Bradley A, Bucan M, Capecchi M, Collins FS, Dove WF, Duyk G, Dymecki S, Eppig JT, Grieder FB, Heintz N, Hicks G, Insel TR, Joyner A, Koller BH, Lloyd KC, Magnuson T, Moore MW, Nagy A, Pollock JD, Roses AD, Sands AT, Seed B, Skarnes WC, Snoddy J, Soriano P, Stewart DJ, Stewart F, Stillman B, Varmus H, Varticovski L, Verma IM, Vogt TF, von Melchner H, Witkowski J, Woychik RP, Wurst W, Yancopoulos GD, Young SG, Zambrowicz B (September 2004). “The knockout mouse project”. Nature Genetics 36 (9): 921–4. doi:10.1038/ng0904-921. PMC 2716027. PMID 15340423.
(151)^ Deng C (October 2007). “In celebration of Dr. Mario R. Capecchi's Nobel Prize”. International Journal of Biological Sciences 3 (7): 417–9. doi:10.7150/ijbs.3.417. PMC 2043165. PMID 17998949.
(139)^ Eisenberg E, Levanon EY (October 2013). “Human housekeeping genes, revisited”. Trends in Genetics 29 (10): 569–74. doi:10.1016/j.tig.2013.05.010. PMID 23810203.
(140)^ Amsterdam A, Hopkins N (September 2006). “Mutagenesis strategies in zebrafish for identifying genes involved in development and disease”. Trends in Genetics 22 (9): 473–8. doi:10.1016/j.tig.2006.06.011. PMID 16844256.
(141)^ “About the HGNC”. HGNC Database of Human Gene Names. HUGO Gene Nomenclature Committee. 2015年5月14日閲覧。
(142)^ Cohen SN, Chang AC (May 1973). “Recircularization and autonomous replication of a sheared R-factor DNA segment in Escherichia coli transformants”. Proceedings of the National Academy of Sciences of the United States of America 70 (5): 1293–7. Bibcode: 1973PNAS...70.1293C. doi:10.1073/pnas.70.5.1293. PMC 433482. PMID 4576014.
(143)^ Esvelt KM, Wang HH (2013). “Genome-scale engineering for systems and synthetic biology”. Molecular Systems Biology 9 (1): 641. doi:10.1038/msb.2012.66. PMC 3564264. PMID 23340847.
(144)^ Tan WS, Carlson DF, Walton MW, Fahrenkrug SC, Hackett PB (2012). “Precision editing of large animal genomes”. Advances in Genetics Volume 80. 80. pp. 37–97. doi:10.1016/B978-0-12-404742-6.00002-8. ISBN 9780124047426. PMC 3683964. PMID 23084873
(145)^ Puchta H, Fauser F (2013). “Gene targeting in plants: 25 years later”. The International Journal of Developmental Biology 57 (6–8): 629–37. doi:10.1387/ijdb.130194hp. PMID 24166445.
(146)^ Ran FA, Hsu PD, Wright J, Agarwala V, Scott DA, Zhang F (November 2013). “Genome engineering using the CRISPR-Cas9 system”. Nature Protocols 8 (11): 2281–2308. doi:10.1038/nprot.2013.143. PMC 3969860. PMID 24157548.
(147)^ Kittleson JT, Wu GC, Anderson JC (August 2012). “Successes and failures in modular genetic engineering”. Current Opinion in Chemical Biology 16 (3–4): 329–36. doi:10.1016/j.cbpa.2012.06.009. PMID 22818777.
(148)^ Berg P, Mertz JE (January 2010). “Personal reflections on the origins and emergence of recombinant DNA technology”. Genetics 184 (1): 9–17. doi:10.1534/genetics.109.112144. PMC 2815933. PMID 20061565.
(149)^ Guan C, Ye C, Yang X, Gao J (February 2010). “A review of current large-scale mouse knockout efforts”. Genesis 48 (2): 73–85. doi:10.1002/dvg.20594. PMID 20095055.
(150)^ Austin CP, Battey JF, Bradley A, Bucan M, Capecchi M, Collins FS, Dove WF, Duyk G, Dymecki S, Eppig JT, Grieder FB, Heintz N, Hicks G, Insel TR, Joyner A, Koller BH, Lloyd KC, Magnuson T, Moore MW, Nagy A, Pollock JD, Roses AD, Sands AT, Seed B, Skarnes WC, Snoddy J, Soriano P, Stewart DJ, Stewart F, Stillman B, Varmus H, Varticovski L, Verma IM, Vogt TF, von Melchner H, Witkowski J, Woychik RP, Wurst W, Yancopoulos GD, Young SG, Zambrowicz B (September 2004). “The knockout mouse project”. Nature Genetics 36 (9): 921–4. doi:10.1038/ng0904-921. PMC 2716027. PMID 15340423.
(151)^ Deng C (October 2007). “In celebration of Dr. Mario R. Capecchi's Nobel Prize”. International Journal of Biological Sciences 3 (7): 417–9. doi:10.7150/ijbs.3.417. PMC 2043165. PMID 17998949.
情報源[編集]
- 主要教科書
- Bruce Alberts, Alexander Johnson, Julian Lewis, David Morgan, Martin Raff, Keith Roberts, Peter Walter 著、中村桂子, 松原謙一 訳『細胞の分子生物学 第6版』ニュートンプレス、2017年9月15日。ISBN 978-4315520620。
- Alberts B, Johnson A, Lewis J, Raff M, Roberts K, Walter P (2002). Molecular Biology of the Cell (Fourth ed.). New York: Garland Science. ISBN 978-0-8153-3218-3 – A molecular biology textbook available free online through NCBI Bookshelf.

Molecular Biology of the Cell (4th edition) の参照した章 (英語)
推薦文献[編集]
●ジェームス・D・ワトソン 他 著、中村桂子 監訳, 滋賀陽子 他訳 訳﹃ワトソン遺伝子の分子生物学 第7版﹄東京電機大学出版局、2017年1月20日。ISBN 9784501630300。
●ドーキンス,リチャード 著、日高敏隆/岸由二/羽田節子/垂水雄二 訳﹃利己的な遺伝子40周年記念版﹄紀伊國屋書店、2018年2月15日。ISBN 978-4314011532。
●H. Lodishほか 著、堅田利明, 須藤和夫, 山本啓一 訳﹃分子細胞生物学 第9版﹄東京化学同人、2023年7月31日。ISBN 9784807920518。
●Bruce Alberts, Karen Hopkin, Alexander Johnson, David Morgan, Martin Raff, Keith Roberts, Peter Walter 著、中村桂子, 松原謙一, 榊佳之, 水島昇 訳﹃Essential細胞生物学(原書第5版)﹄南江堂、2021年7月13日。ISBN 978-4-524-22682-5。
●T. A. Brown 著、西郷薫 監訳 訳﹃ブラウン 分子遺伝学 [第3版]﹄東京化学同人、1999年3月25日。ISBN 9784807905010。
●Ridley M (1999). Genome: The Autobiography of a Species in 23 Chapters. Fourth Estate. ISBN 978-0-00-763573-3
●Brown, Terence A. (2023). Genomes 5 (Fifth edition ed.). Boca Raton, FL London New York: CRC Press, Taylor & Francis Group. ISBN 978-0-367-67407-6
外部リンク[編集]
- Comparative Toxicogenomics Database - 米国国立環境健康科学研究所による人間の健康と環境暴露の関係を理解することを目的としたデータベース
- DNA From The Beginning – a primer on genes and DNA コールド・スプリング・ハーバー研究所による現代遺伝学の入門書 (英語)
- Gene - アメリカ国立生物工学情報センターによる遺伝子の検索可能なデータベース
- Genes – オープンアクセス専門の出版社MDPIによるオープンアクセスジャーナル
- IMPC (International Mouse Phenotyping Consortium) – 国際マウス表現型解析コンソーシアムによる哺乳類遺伝子機能の百科事典


