古細菌
表示
| 古細菌 | |||
|---|---|---|---|
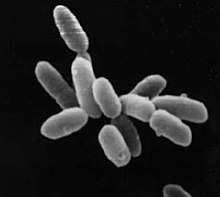 Halobacterium sp. NRC-1 | |||
| 地質時代 | |||
| 太古代(先カンブリア代) - 現代 | |||
| 分類 | |||
| |||
| 学名 | |||
| Archaea Woese et al., 1990[1] | |||
| 下位分類(門)[注 2] | |||
|
(詳細は#系統概観を参照) |
古細菌︵こさいきん︶ または アーキア︵羅: archaea、アルカエア[4]、単数形: 羅: archaeum、archaeon︶は、生物の主要な系統の一つである。細菌︵バクテリア︶、真核生物︵ユーカリオタ︶と共に全生物界を3分している。古細菌は形態や名称こそ細菌と類似するが、細菌とは異なる系統である。高度好塩菌、メタン菌、好熱菌などが良く知られている。
日本語では﹁古細菌﹂または﹁アーキア﹂が呼称されることが多い。﹁始原菌︵しげんきん︶﹂[5][6]も使われる。﹁古細菌﹂という名称は、﹁菌﹂および﹁細菌﹂を名前に含むが、菌類︵真菌︶や細菌︵真正細菌︶とは異なる。

細菌、古細菌、真核生物の関係。
初期の地球上には古細菌と細菌の2ドメインしか存在せず、真核生物は古細菌︵特にアスガルド古細菌︶から遅れて進化したとする説が現在有力である︵古細菌#古細菌の起源と系統的位置を参照︶。古細菌と細菌では、細胞膜や細胞壁の構成、DNA複製機構など様々な点で相違がある︵古細菌#細胞の形態・構造などを参照︶。
古細菌ドメインは、ユーリ古細菌、TACK群︵クレン古細菌、タウム古細菌など︶、アスガルド古細菌、DPANN群に大きく分けられている。タウム古細菌以外はヒトから見れば極端な環境に生息しており、極限環境微生物とも呼ばれる︵古細菌#生息環境を参照︶。ヒトに身近なのは腸内常在微生物叢の一部を占め、嫌気性の沼などにもいるメタン菌や、窒素循環に関連する亜硝酸古細菌程度︵タウム古細菌に含まれる︶である。これ以外にも様々な生物が含まれるとみられるが、多くは未培養であり、古細菌の全容は解明されていない。2018年時点で正式に記載されている古細菌は約550種である。

古細菌内部の系統樹の一例[7]

"Ca. Korarchaeum cryptofilum"︵コル 古細菌︶の電子顕微鏡写真。
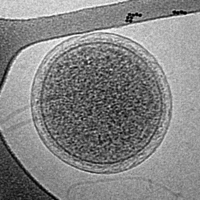
"Ca. Parvarchaeum acidiphilum"︵パ ルウ古細菌︶の電子顕微鏡写真。
分類はNCBIによる。門、綱における太字は記載種を含む系統を表す。各系統の概略もリストの下に記す。
概要[編集]
地球上の全ての生物は、細菌、古細菌、真核生物の3つのドメインのいずれかに分類される。細菌と古細菌は合わせて原核生物とも呼ばれ、真核生物と対比される。ドメインによる分類は、リボソームRNA配列などを用いた分子系統解析を基にしており、ドメイン内では基本的な遺伝の仕組みや生化学的性質に共通性が見られる。例えば、植物と動物は見た目は大きく異なるが、細胞レベルで見るとDNA複製のメカニズムや細胞膜の主成分などは共通性が高い。ドメインが異なる生物同士は、分子レベルでこれら生命を維持する機構が異なっている︵古細菌#他ドメインとの違いを参照︶。
古細菌の分類[編集]
古細菌の種から綱までの命名は国際原核生物命名規約に基づいて行われており、基本的な分類方法は細菌と共通している。原核生物は形態の変化に乏しく無性的に増殖するため、動植物でいう種の基準が適用できず、分類は16S rRNA系統解析やDNA-DNA分子交雑法、ゲノム構造に基づく平均ヌクレオチド一致度(ANI)といった分子技法が主に用いられる。記載種を含む系統にはクレン古細菌、タウム古細菌、ユーリ古細菌があるが、DPANN群など多くの未培養系統が存在している。系統概観[編集]



●ユーリ古細菌界 / Euryarchaeota
●ユーリ古細菌門 / Euryarchaeota
●テルモコックス綱 / Thermococci
●メタノマダ / Methanomada︵クラスIメタン菌︶
●メタノバクテリウム綱 / Methanobacteria
●メタノコックス綱 / Methanococci
●メタノピュルス綱 / Methanopyri
●アルカエオグロブス綱 / Archaeoglobi
●テルモプラズマ綱 / Thermoplasmata
●ステノス古細菌 / Stenosarchaea
●メタノミクロビウム綱 / Methanomicrobia︵クラスIIメタン菌︶
●メタノナトロナルカエウム綱 / Methanonatronarchaeia
●ハロバクテリウム綱 / Halobacteria
●プロテオ古細菌界 / Proteoarchaeota
●TACK群 / TACK superphylum
●クレン古細菌門 / Crenarchaeota
●テルモプロテウス綱 / Thermoplotei
●タウム古細菌門 / Thaumarchaeota
●ニトロソスパエラ綱 / Nitrososphaeria
●コネクシウィスパエラ綱 / Conexivisphaeria
●アイグ古細菌門 / Aigarchaeota
●コル古細菌門 / Korarchaeota
●バテュ古細菌門 / Bathyarchaeota
●ジオ古細菌門 / Geoarchaeota
●ジオサーム古細菌門 / Geothermarchaeota
●マーズ古細菌門 / Marsarchaeota
●ヴェルストレート古細菌門 / Verstraetearchaeota
●アスガルド古細菌 / Asgardarchaeota
●ロキ古細菌門 / Lokiarchaeota
●ヘイムダル古細菌門 / Heimdalarchaeota
●オーディン古細菌門 / Odinarchaeota
●トール古細菌門 / Torarchaeota
●ヘル古細菌門 / Helarchaeota
●ゲルド古細菌門 / Gerdarchaeota
●フレイヤ古細菌門 / Frejaarchaeota
●ゲフィオン古細菌門 / Gefjonarchaeota
●フリッグ古細菌門 / Friggarchaeota
●イズン古細菌門 / Idunarchaeota
●カーリ古細菌門 / Kariarchaeota
●バルドル古細菌門 / Balderarchaeota
●ヘーダア古細菌門 / Hoderarchaeota
●ラグ古細菌門 / Lagarchaeota
●シフ古細菌門 / Sifarchaeota
●ブロック古細菌門 / Brockarchaeota
●ホッド古細菌門 / Hodarchaeota
●ウーコン古細菌門 / Wukongarchaeota
●DPANN群 / DPANN superphylum
●ディアペロトリテス門 / Diapherotrites (pMC2A384)
●パルウ古細菌門 / Parvarchaeota (ARMAN-4, 5)
●ミクル古細菌門 / Micrarchaeota (ARMAN-1, 2)
●アエニグム古細菌門 / Aenigmarchaeota (DSEG)
●ナノ好塩古細菌門 / Nanohaloarchaeota
●ナノ古細菌門 / Nanoarchaeota
●アルティ古細菌門 / Altiarchaeota︵ユーリ古細菌?︶
●ウンディン古細菌門 / Undinarchaeota
●ウーズ古細菌門 / Woesearchaeota
●ペース古細菌門 / Pacearchaeota
●マム古細菌門 / Mamarchaeota
●フーバー古細菌門 / Huberarchaeota

Methanohalophilus mahii︵メタノサルキナ目 ︶の電子顕微鏡写真。

Haloquadratum walsbyi︵ハロバクテリウム目︶ の蛍光顕微鏡写真。

Haloferax volcanii︵ハロフェラクス目︶の電子顕 微鏡写真。
ユーリ古細菌界[編集]
ユーリ古細菌門[編集]



詳細は「ユーリ古細菌」を参照
2018年現在約480種が記載。塩湖や塩田など非常に高い塩濃度を好む高度好塩菌、嫌気環境でメタンを生成するメタン菌、 超好熱菌、好熱好酸菌などを含む。Zリングで分裂する。アクチンを欠く。細胞内にゲノムを複数コピー持つ。
プロテオ古細菌界[編集]
TACK群およびアスガルト古細菌を含む[3]。エオサイト界 (Eocyta)とも呼ばれる[8]。ESCRT複合体で分裂、EF-1︵伸長因子-1︶に11アミノ酸残基の挿入がある。TACK群[編集]
名称はタウム古細菌、アイグ古細菌、クレン古細菌、コル古細菌の頭文字を並べたもの[9]。フィル古細菌 (Filarchaeota)とも呼ばれる[2]。- クレン古細菌門
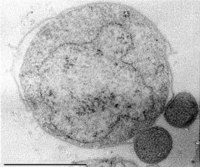
Ignicoccus hospitalis(大きい方がI. hospitalis) 
"Sulfolobus tengchongensis" RT8-4の電子顕微鏡写真 詳細は「クレン古細菌」を参照2018年現在64種が記載。温泉などにいる好熱好酸菌、80℃以上の高温を好み海底熱水噴出孔などにいる超好熱菌などを含む。ESCRT複合体で分裂する。古細菌固有のDNA複製酵素であるDファミリーDNAポリメラーゼを欠く。テルモプロテウス目以外はヒストンを持たないことが多い。 タウム古細菌門詳細は「タウム古細菌」を参照2021年現在6種が記載。2008年にクレン古細菌から分けられた系統。中温性の亜硝酸古細菌などを含む。クレン古細菌同様にESCRT複合体で分裂する。クレン古細菌より低温に適応している。 アイグ古細菌門 地下320mの金鉱より発見された"Ca. Caldiarchaeum subterraneum’"。ゲノム情報からは、好気または硝酸呼吸により、水素や一酸化炭素を酸化していると予想されている。ユビキチン-プロテアソームシステムに必要な遺伝子を持つ[10]。タウム古細菌に近縁で、タウム古細菌に含めることもある。 コル古細菌門 環境DNAサンプルと集積培養系のみ。環境中での存在量・分布は小さいと考えられる。嫌気従属栄養性の超好熱菌。ユーリ古細菌とクレン古細菌の2大系統以外では最も最初に発見され、当初は古細菌の中で原始的な系統に属すと考えられたが[11]、現在ではクレン古細菌に近い系統と考えられている[9]。集積培養が得られている"Ca. Korarchaeum cryptofilum"は、嫌気性・超好熱性の従属栄養生物である[12]。 バテュ古細菌門 海底堆積物などで豊富に見つかる系統。未培養だがメタン生成経路[13]やバクテリオクロロフィルa合成酵素の検出例がある特異な系統[14]。アスガルド古細菌[編集]
詳細は「アスガルド古細菌」を参照古細菌の中で、最も真核生物に近縁とされる系統。
DPANN群[編集]
詳細は「DPANN群」を参照集積培養や環境DNAのみだが、極端に細胞とゲノムサイズが小さい。古細菌の中でもっとも初期に分かれた系統とする系統解析例が多いが、特殊化したユーリ古細菌とする見解もある[3]。 ディアペロトリテス門 明神海丘︵伊豆・小笠原弧︶の水深1330mにあるブラックスモーカーより最初に報告されたもの[15]。ホームステーク金山跡の地下水から検出された集団は、ゲノム解析から従属栄養生物と予想されている[16]。同時に、ナノ古細菌などと同様の寄生生物から進化した可能性に言及されている[16]。提案された古細菌門の中では、唯一-archaeotaを語尾に持たない。 パルウ古細菌門 テルモプラズマ目古細菌に関係︵寄生の可能性もある︶[17]。非常に細胞サイズが小さい特徴がある[17]。 ミクル古細菌 パルウ古細菌同様テルモプラズマ古細菌に関係し[17]、細胞サイズが小さい。細胞サイズの小ささは全生物でもトップクラスで、長さ200 nm×幅60 nm、体積も0.009 μm3から0.04 μm3しかない[18]。ただし系統はやや離れる。 アエニグム古細菌門 深海の熱水噴出孔に存在する系統。 ナノ好塩古細菌門 高塩環境に分布する[19]。ハロバクテリウム綱とは別系統[19]。 ナノ古細菌門 クレン古細菌に寄生する。ゲノムサイズが非常に小さい。最初に発見され、Ignicoccus hospitalisに寄生する"Ca. Nanoarchaeum equitans"は、ゲノムサイズが古細菌最少の49万0885塩基対しかない[20]。生息環境[編集]
極限環境[編集]

高度好塩古細菌によって赤く着色したサンフランシスコ湾の塩田 
東太平洋海嶺のブラックスモーカー 
強い酸性を帯びた坑廃水 古細菌は生物圏の広い範囲に分布し、最大で地球上の総バイオマスの20%を占めるとも言われている[21]。純粋培養が可能な古細菌の多くは、間欠泉やブラックスモーカー、油田、塩田、塩湖、強酸、強アルカリ環境などのヒトから見て極限環境に生息するものがほとんどであり、このため歴史的に極端な環境のみに分布すると考えられてきた︵極限環境微生物︶[22][23]。実際、20世紀末まで通常の土壌・水系から古細菌が分離されることは、一部のメタン菌を除き殆ど無かった。これらの極限的な環境に生息する古細菌は、大まかに高度好塩菌、超好熱菌、好熱好酸菌へと区分することができる。高度好塩菌は、20-25%のNaCl濃度で盛んに増殖し、塩湖など非常に塩濃度の高い環境に生息する。アフリカや中国の塩湖の中にはpHが10を超えるものもあり、このような環境からは、好アルカリ性高度好塩菌が分離されている。有名なものとして、pH12で増殖できる高度好塩菌Natronobacterium gregoryiがある。高度好塩菌は特別な培養装置を必要とせず、基本的には培地に塩を加えるだけで良いので[注 3]、2018年現在記載種は250種近くに達している︵これは古細菌ドメインに記載されている種の半数に近いが、古細菌の半分が好塩菌ということではなく、単に培養の容易さを反映している︶。 好熱菌は温泉など45°C以上の環境でよく活動するものをいう。このうち80°C以上に至適生育温度を持つものを超好熱菌と呼ぶ。Methanopyrus kandleri Strain 116は、全生物中最も高温で生育する生物として知られ、122°Cで増殖が可能と報告された[24]。このほかPyrococcus、Pyrodictiumなどがあり、温泉や陸上硫黄孔、火山、海底熱水噴出孔などの多様な熱水系に生息する。嫌気性のものが多いが、偏性好気性の超好熱菌もAeropyrum pernix、Sulfurisphaera tokodaiiなど幾つかいる︵後者は好酸性も兼ねる︶。 硫黄分を含む熱泉では、硫黄が酸化されてしばしば強い酸性になる。強酸を好む好熱好酸菌は、スルフォロブス目やテルモプラズマ目に代表され、温泉や硫気孔、ボタ山などから発見される。初期に発見されたものとしてはSulfolobusやThermoplasmaなどがある。好酸菌の極端な例としては、pH-0.06︵1.2M硫酸溶液下に相当︶で増殖する好熱好酸菌Picrophilus[25]がいる。Stygiolobus azoricusを除き、大半が偏性好気性か通性嫌気性である。 また、超好熱菌、好熱好酸菌などの菌群は表現型による区分であり、系統による分類と一致するとは限らない。高度好塩菌はハロバクテリウム綱、好熱好酸菌はテルモプラズマ目及びスルフォロブス目にほぼ一致するが、超好熱菌は古細菌ドメインの広い分類範囲に存在し、むしろ超好熱菌のいない目の方が少数派である。また、場合によっては複数条件で極限環境微生物と言えるものもあり、Methanonatronarchaeum thermophilumなどは、好熱性・好アルカリ性かつ強い好塩性のメタン菌である[26]。そのほかにも、超好熱かつ好酸性のSulfurisphaera ohwakuensis︵限界温度92℃、限界pH1︶、超好熱かつ好アルカリ性のThermococcus alkaliphilus︵限界温度90℃、限界pH10.5︶などがある。 各生育・生存パラメータにおける代表種と限界値は以下のとおりである ●高温‥Methanopyrus kandleri 122℃[注 4] ●アルカリ性‥Natronobacterium gregoryi pH12[注 5] ●酸性‥Picrophilus oshimae pH-0.06︵マイナス0.06︶[27][注 6] ●高NaCl濃度‥Halobacterium salinarumなど 飽和濃度[注 7] ●高圧力‥Pyrococcus yayanosii 1200気圧[注 8] ●放射線‥Thermococcus gammatolerans 30000グレイのガンマ線を照射しても一部は生き残る︵Cs137線源︶[28][注 9] なお、極限環境微生物と古細菌はしばしば同義として使われることがあるが、すべての古細菌が極限環境微生物というわけではなく、同様にすべての極限環境微生物が古細菌というわけではない。極限環境で生育する細菌も多数存在しており、少数ではあるが超好熱性の細菌も知られている[注 10]。とはいえ、やはり細菌は医療細菌や常在細菌の存在感が大きく、古細菌ほどは極限環境微生物の割合は多くない。嫌気環境[編集]

タイ王国 チェンマイ県の水田 記載されている嫌気性の古細菌は、偏性嫌気性が約260種、通性嫌気性が約10種となっている。なお、偏性好気性の古細菌は約270種で、記載種に関しては約半分が好気性、半分が嫌気性ということになる。嫌気性の古細菌で代表的なものはメタン菌である。約160種が記載されている。これは代謝の結果メタンを生成する微生物の総称であるが、このような代謝を行う生物は古細菌以外に知られていない。強い嫌気度を要求し、水素や酢酸などを代謝する為、それらが豊富な環境に分布する。例えば海底熱水噴出孔などでは、付近に生息する微生物によって水素が発生しており、それらを餌にメタン菌が大量に存在している。これらは同時に超好熱性も備えている。これら超好熱菌は、偏性好気性のAeropyrum、Sulfolobus、Sulfurococcus、通性嫌気性のAcidianus、Pyrolobus、Pyrobaculum aerophilumを除いて大半が偏性嫌気性であり、Methanopyrus kandleri、Methanocaldococcus jannaschii、Methanothermus fervidusなどがある。 水田や湖沼、海洋堆積物の中も微生物の働きによって酸素が消費され、水素や有機酸・アルコールなどが発生しており、それらをメタン菌が消費している。海洋ではメタノコックス綱[29]、淡水系ではメタノバクテリウム綱やメタノミクロビウム綱が主にみられる[29]。動物の消化器官や発酵槽などでもメタノバクテリウム綱やメタノミクロビウム綱が生息している[30]。メタン発酵槽には好熱性のものやMethanosaetaが多い[30]。 メタン菌はかなり広い範囲に分布しており、メタン菌そのものは極限環境微生物に含めないことが多い。ただし、増殖には酸化還元電位にして-0.33Vの非常に強い嫌気環境が必要である。メタン菌以外では、冷湧水帯堆積物や海洋堆積物に、未培養系統であるが、ANME I-IIIと呼ばれる嫌気的メタン酸化菌が存在する[31]。この他にも、膨大な数の古細菌が海底の堆積物の中から見つかっている。2008年には、海底1m以深の沈澱物中に存在する生物の大部分を古細菌が占めるという報告がなされた[32][33]。これらは殆ど培養されておらず、不明な点が多い。より温和な環境[編集]

海綿の1種︵アキシネラ︶内部に多数のCandidatus "Ce narchaeum symbiosum"︵タウム古細菌︶を共生させている 現在では、極限環境や嫌気環境だけでなく、より温和な環境にも︵メタン菌以外の︶古細菌が存在することが知られている。例えば極地の海、湖などの冷たい環境において古細菌の遺伝子が高頻度で検出されている[34]。一般的な海洋においても、細胞数当たりで微生物の約20%を古細菌が占めるという[35]。湿原や下水、海洋、土壌などにも古細菌は存在する[36]。これらの古細菌の多くは、メタゲノム、脂質解析といった手法によりその生態が明らかにされつつある。 特にタウム古細菌[37]は、2000年以降急速に研究が進展したグループである。2005年に初めてNitrosopumilus martimus純粋培養に成功し[38]、2014年にはNitrososphaera viennensisが記載[39]された︵2018年度の記載種の数は6種︶。分離源は水族館のフィルターや海水、畑の土といった"通常の"環境で、生育温度も25~42℃と低く、pH、塩濃度といった他の生育パラメータも極端な数字ではない[39][40][41]。タウム古細菌以外の系統は培養に成功していないが、環境DNAサンプルとして多数存在し、代表的なものとして海洋の有光層に多いMarine group IIと呼ばれるグループが知られている[42]。細胞の形態・構造[編集]
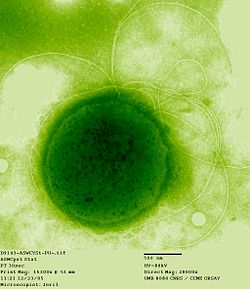
典型的な超好熱菌Thermococcus gammatolera nsの電子顕微鏡写真
古細菌の基本的な構造︵モデルは上のThermococcus ga mmatolerans︶。細胞膜の外側には細胞壁がある。膜につつまれた細胞小器官は存在せず、内容物は混ざっている。色は区別のための着色で、実際のものとは異なる。 古細菌の外観は細菌と似ている。0.5から数マイクロメートル程度の大きさを有し、球菌、桿菌またはディスク状など様々な形が見られる。大きさは最大の球菌で直径10数μm程度である[43]。珍しい形として、Haloquadratum walsbyiは、極薄の四角形の紙片状[44]、高度好塩菌には他に三角菌︵Haloarcula japonica ︶もいる。 Thermofilum pendensは極細の針状︵最大長~100μm︶、ThermoplasmaやFerroplasmaは、強固な細胞壁を持たないために、一部の種は定まった形を持たず、アメーバのような形になることもできる[45]。また複数の細胞が集合して大規模な融合細胞を形成するものも存在する。この例としてはThermococcus coalescensが知られている[46]。 古細菌は原核生物であるため、通常細胞内の膜系を発達させず、細胞内の目立つ構造物と言えばDNAとリボソーム、ガス泡、PHBの顆粒くらいである。これらを含む細胞質を細胞膜がつつみ、その外側を細胞壁が覆う。一般に細胞壁は細菌よりも薄く、機械的強度も弱い。細胞表面には、鞭毛や線毛、繊維状の付属構造を持つ場合がある。なお、細胞内の膜系に関しては、ThermoplasmaやIgnicoccusといった例外も存在する[47][48]。Ignicoccusは、外細胞膜と内細胞膜、その間の巨大な疑似ペリプラズムに特徴づけられる。外側の膜にATP合成酵素があり、疑似ペリプラズムにおいてもATPが利用可能な点で、グラム陰性細菌と異なる[49]。内部はフィラメントや網構造が非常に入り組んで観察される[50]。 細胞よりも高次の構造も乏しく、殆どの種は単独か原始的な群体を持つに過ぎない。Methanosarcinaは接着物質を使用し、小荷物様の群体を形成する。他のメタン菌の中には、シースと呼ばれる鞘の中に複数の細胞が鎖のようにつながった形態をとるものがある。シート形成や網目状のネットワークを形成するものもある[51][52]。何れにせよその形態の多様性は限定されており、形態により古細菌を特徴づけるのは困難である。古細菌を特徴付けているのは、ほとんどが分子生物学的知見による。細胞壁[編集]
古細菌の細胞壁は一般的にタンパク質性のS層である[53]。S層は多くの細菌にも認められるが、細菌と異なりペプチドグリカンを持たず、S層そのものが細胞壁になっているという点で異なる。古細菌のS層は熱に対して極めて安定だが、細菌の細胞壁と異なり浸透圧変化に脆弱で機械的強度も弱いものが多い[54]。メタノバクテリウム綱は、シュードムレインと呼ばれる糖ペプチドを持つ[53]。これはペプチドグリカンの一種ではあるが、ムラミン酸やdアミノ酸を欠くという点で細菌の細胞壁と区別できる。Methanopyrus kandleri、Methanothermusは、シュードムレインの外側に更にS層がある[53]。S層もシュードムレインも、その合成系の違いから、細菌の細胞壁合成を阻害するβ-ラクタム系抗生物質、グリコペプチド系抗生物質は効果が無い。一般的な傾向として、グラム染色ではS層が陰性に、シュードムレインが陽性に染色される。[55] その他の細胞表層構造としては、シース︵Methanospirillus、Methanosaeta︶、メタノコンドロイチン[56]︵Methanosarcina︶、多糖類︵Halococcus︶、グルタミニルグリカン︵Natronococcus︶などがある[53]。また、テルモプラズマ綱は細胞壁が無い。多くの古細菌はグラム陽性細菌同様外膜を持たないが、Ignicoccus及びMethanomassiliicoccus luminyensis、未培養系統であるARMAN[53]、アルティアルカエウム目は外膜︵または外細胞膜︶を持つ[57]。これらは系統的に離れていて、進化的な意義は不明である。鞭毛[編集]
基部のモーターにより鞭を回転させ、細胞の移動を可能とする器官である。直径10-15nm、全長10-15μm。細菌の鞭毛に似るが、よく見るとやや細く、また、構成するタンパク質にも相同性はない。むしろ古細菌自身やグラム陰性細菌が持つIV型線毛との共通点が多い。一方、細菌の鞭毛はIII型分泌装置との共通点が多く、両者は異なる起源を持つと考えられている。鞭毛の繊維部分は根元にユニットが追加される形で伸長する[注 11]。また、細菌は鞭毛の駆動力として水素イオン濃度差を利用するが、こちらはATPの加水分解により駆動する。エネルギー変換効率はほぼ100%の高効率を達成している細菌に比べて著しく低く、6~10%程度と見積もられている[58]。[59][60][61]
古細菌とその他の生物の極性脂質の違い。1-4, 10は古細菌細胞 膜脂質の代表例2,3-ジフィタニル-sn-グリセロール1-リン酸、5-9はそのほかの生物の細胞膜脂質細胞膜[編集]
細胞膜を構成する脂質は、古細菌と他の2ドメインを区別する最大の特徴である。真核生物および細菌はグリセロールのsn-3位[注 12]にリン酸基が結合したsn-グリセロール3-リン酸を基盤として、sn-1位、2位に脂肪酸残基がエステル結合している。対して古細菌がもつリン脂質はこれと鏡像体の関係にあり、sn-1位にリン酸基が結合したsn-グリセロール1-リン酸を基盤として、sn-2位、3位に脂肪酸ではなくイソプレノイド基がエーテル結合している。ただし、両者の区別は以前考えられていたほど厳密ではない。エーテル結合を含む脂質や環状脂質は、超好熱菌アクイフェクスやサーモデスルホバクテリア︵Thermodesulfobacterium︶などからも見つかっている。また2020年、一部の細菌︵FCB群︶が細菌タイプと古細菌タイプの脂質が混在した細胞膜をもっている可能性が報告された[62]。一方で、一部の古細菌は、細菌タイプの脂質を合成するための遺伝子の一部をもつことが知られている[63]。ただし、必要な遺伝子をすべてもつ古細菌は知られておらず、実際に細菌タイプの細胞膜を合成する例は未だに報告されていない。 古細菌および細菌どちらの場合も、脂質分子は互いに向かい合って脂質2重層を形成している。古細菌がもつ脂質分子の炭化水素鎖は多くの場合、炭素数20︵稀に25︶のイソプレノイドからなる。不飽和型は稀である[要出典]。一部の古細菌の細胞膜には、C20イソプレノイド鎖︵フィタン鎖︶が向かい合って結合したC40ジフィタン鎖からなるテトラエーテル型脂質や、炭化水素鎖の途中で架橋、あるいは環状構造を有す物も存在する。これら脂質分子の組成は古細菌の種類および生息環境によって変化する。 細胞膜上にはATP合成酵素や電子伝達体︵その他メタン生成経路やバクテリオロドプシンなども︶などの酵素類が偏在しており、古細菌の代謝の主要な場である。膜上にはこの他に各種輸送体や各種センサーなどが存在する。[64]細胞質[編集]
細胞質に目立つ構造は少ない。DNA、エネルギー貯蔵用のポリヒドロキシ酪酸の顆粒、リボソーム、高度好塩菌などが持つ浮力調整用のガス胞などが比較的目立つ程度である。細胞骨格については、Thermoplasmaが細胞壁がないにもかかわらず、様々な形をとることから、発見時より何らかの形で細胞骨格が存在することが推測されていた[65]。これは細菌のMreBに類似した蛋白質が使われている。 一方、クレン古細菌からはアクチンに類似するタンパク質が報告されている。ロキ古細菌から発見されたアクチンは、ヒトアクチンと58-60%の同一性を持ち、プロフィリンはウサギアクチンと相互作用を起こすことができる[66][67]。この系統やタウム古細菌からはチューブリンに近い遺伝子も報告されているが、実態は良く分かっていない[68]。DNAと遺伝子発現[編集]
DNA[編集]
1996年にMethanocaldococcus jannaschiiの全ゲノムが解読されて以来、2018年までに250株以上の古細菌についてゲノムの解析が行われた。ゲノムサイズは1.2 - 6 Mbp︵Mbp=100万塩基対︶と細菌と比較してもやや小さく、Methanothermus fervidusのゲノムは124万3342bpしかない[69]。完全独立生物を送るものとしては最小である。さらにIgnicoccus hospitalisという古細菌に共生している“Ca. Nanoarchaeum equitans”に至っては宿主に完全に依存しているとはいえ、49万885bpというきわめて小さなゲノムを持つ[70]。これまでに解析された古細菌のうち、最大のゲノムを持つのはMethanosarcina acetivorans︵575万1492bp︶である[71][注 13]。ゲノムは好熱菌では1分子のことが多いが、高度好塩菌や一部のメタン生成菌は副ゲノムやプラスミドを所持する例も多い。ゲノムサイズは小さいものの、古細菌のゲノムは細菌や真核生物よりも複雑性が高いという[72]。 DNAの構造は細菌に類似しており、環状のDNAを持ち、それが凝集して核様態を形成している。一方で、DNA結合タンパクは細菌とは異なり、一般的に古細菌型ヒストンである[73][74]。Methanothermus fervidusのヒストンは詳細に観察されていて、真核生物のH3-H4四量体に対応する構造をとる。この四量体におおよそ60bpのDNAが巻き付き、真核生物のヌクレオソームに類似する構造を形成することが報告されている[75][76][注 14]。ただし古細菌型ヒストンは、三次構造レベルでは真核生物ヒストンによく似ているものの、翻訳後修飾を受けるという報告は無く、N末側テールに相当する領域も欠く[注 15]。その他各種DNA結合タンパクが存在する。テルモプラズマ綱とデスルフロコックス目、スルフォロブス目はヒストンを持っておらず、それぞれ細菌のHU様タンパクや[77]、独自のAlbaタンパクを使用する[78]。 古細菌のDNA複製は真核生物に近い。対して細菌のもつ複製機構とは類似性がなく、古細菌・真核生物と細菌とで起源が異なると推定される[79][80]。複製を担うDNAポリメラーゼについては、真核生物がBファミリーDNAポリメラーゼ︵以下PolB︶を使用するのに対して、古細菌は独自のDファミリーDNAポリメラーゼ︵以下PolD︶を併せもち[81]、このどちらか、または両方が複製に使用されている[注 16]。岡崎フラグメントの長さは真核生物と同様短いが[82]、複製速度は細菌同様速い。DNAそのものは細菌と同じく環状2本鎖にもかかわらず、複製開始地点が複数存在する場合もある[83]。一般に、古細菌のDNA複製機構は、真核生物のそれの祖先型とみられている[84]。[85] また、古細菌に感染するウイルスも多数発見されている。多くは二本鎖DNAウイルスで、形態はかなり多様性に富んでいる。海洋環境でのウイルスによる感染の影響は細菌よりも古細菌の方が大きく、ウイルスによって死滅させられた古細菌から、炭素換算で年間3~5億トンものバイオマスが供給されていると見積もられている[86]。ウイルスに対しての獲得免疫システムとしてCRISPR/Casシステムがあり、古細菌の84%にCRISPRが存在すると推定される[87][注 17]。少数だが制限酵素を持つ古細菌もいる。タンパク質の合成[編集]
DNA情報をもとにタンパク質が合成される過程︵セントラル・ドグマ︶は転写および複製からなっており、これは現存する全生物に共通するが、各ドメイン間で合成機構は微妙に異なる。古細菌のタンパク質合成過程は細菌と類似する点もあるが、使用される酵素の分子構造は真核生物︵真核生物の翻訳参照︶と類似している[88]。まずRNAポリメラーゼがDNA配列に従いmRNAを合成︵転写︶し、さらにリボソームにおいてmRNA配列に従ってタンパク質に翻訳される。転写機構は真核生物のRNAポリメラーゼIIによる転写機構とよく似ていて、立体構造も酷似する[注 18]。リボソームは3つのRNAと70種弱のタンパク質より成り、RNAはやや細菌に、タンパク質は真核生物に近い[注 19]。翻訳開始アミノ酸はメチオニンで、リボソームがストレプトマイシンやキロマイシンによって阻害を受けず、ジフテリア毒素によって阻害を受けることなどの点で真核生物に似ている[注 20]。中央代謝[編集]
古細菌のTCA回路は他の生物とほぼ同じである。好気性の古細菌や一部の嫌気性クレン古細菌は完全なTCA回路を備えており[92]、反応は通常の好気性細菌や真核生物と同様に進行する。残りの嫌気性菌はTCA回路を部分的にしか備えておらず、炭素固定などに利用している。[93] 解糖系は各古細菌種によってED経路︵エントナー-ドウドロフ経路︶、EM経路︵エムデン-マイヤーホフ経路︶何れかが存在する。こちらは他の生物といくつか相違が見られる。いくつかのメタン菌やテルモコックス綱からはEM経路に関係する酵素が見つかっているが、ADP依存性グルコキナーゼやADP依存性ホスホフルクトキナーゼ、ホスホエノールピルビン酸シンターゼなど特異な酵素が関与するため、変形EM経路と呼ばれている[94][92] [注 21]。一方、好気性の古細菌の多くは、好気性の細菌の一部に見られるエントナー-ドウドロフ経路︵ED経路︶に似る経路を使用する[92]。高度好塩菌では、一部の経路がリン酸化せずに進行するため、部分リン酸化ED経路と言う。テルモプラズマ目の非リン酸化経路では、反応の末端である2-ホスホグリセリン酸に至るまでリン酸化を伴わず、更にグリセルアルデヒドからグリセリン酸までの反応が、グリセルアルデヒドデヒドロゲナーゼによってバイパスされるため、系全体の収支としてATPは生成しない。[96][97] ペントースリン酸経路はあまり見られず、リブロースモノリン酸経路を用いる種が多い[98]。独立栄養性の種では、炭素固定回路は種によって異なる。古細菌特有の経路として、Ignicoccusなどがジカルボン酸/4-ヒドロキシ酪酸回路を用いている[99]。繁殖・細胞分裂[編集]
古細菌は基本的に細菌と同様、単純な二分裂によって増殖︵繁殖︶する。出芽により増殖するテルモプロテウス目など一部を除くと、分裂後も殆ど同じクローンが2体できるだけである。胞子や芽胞の形成も確認されていない。最適条件での増殖速度はMethanocaldococcusやPyrococcusで約30分、Methanosaetaなど遅いものだと数日を要する。分裂に伴う細胞膜の切断やDNAの分配は細菌に似ていると言われている。Methanocaldococcus jannaschiiを始めとしたユーリ古細菌のゲノム上にはFtsZ、MinDなどが存在し、細菌と同様、Zリングの収縮で細胞を分裂させると考えられている。一方でクレン古細菌からはFtsZが見つからず、分裂機構は長い間全く不明であった。2008年に真核生物のエンドソーム選別輸送複合体︵ESCRT複合体︶に相当するタンパク質が細胞分裂に関与するという報告がなされている[100][101][102][103]。タウム古細菌も同様の機構を持ち、アスガルド古細菌ではさらに多くのESCRT複合体関連遺伝子が見つかっている[68]。一方で、テルモプロテウス目のゲノムからは、FtsZもESCRT複合体も見つからず、アクチンに類似するタンパク質を用いた細胞分裂機構を持つと予想されている[104]。 有性生殖は存在しないが、特殊な例として、Haloferax volcaniiにおける細胞間架橋構造の形成がある[105]。細菌の接合はプラスミドを移行させる現象であるが、この例ではプラスミドや細胞質は移行せず、ゲノムDNAのみが移行する点で異なる[105]。スルフォロブス目の例では、UV照射や薬剤暴露によるDNA損傷によって細胞凝集が誘導され、染色体の組み換えが起こる[106][107]。それ以外のストレスによっては誘導されず、プラスミドも関与しないなどといった点で細菌の接合とは異なる。また、繊毛を失った株は凝集できず[注 22]、UV照射に対する生存性が低下する[108]。細胞融合性を持つFerroplasma acidarmanusでも激しいゲノム組み換えが見られる[109]。いずれも組み換えはプラスミドでは無くゲノムDNAに制御されている。Sulfolobusではゲノムの交換は種特異的であり、Ferroplasma acidarmanusも進化距離の違いにより組み換え率が急速に低下する[注 23][108][109]。これは古細菌における種の概念を示している可能性がある[108][109]。物質・エネルギー循環における役割[編集]
古細菌は、かつてメタン生成を除き、地球上の物質循環への影響は限定的と考えられてきた。しかし、難培養性の古細菌の研究が進むにつれ、地球規模の物質循環への寄与が無視できないものであることが明らかとなってきている。全体として見た場合、環境中の古細菌は、炭素や窒素、硫黄における物質循環の一部を構成している。 近年注目されているのは窒素循環への関与である。以前からメタン生成菌や好熱菌など一部の古細菌が窒素固定や硝酸塩呼吸を行うことは知られていたが、これらに加え、2005年にタウム古細菌がアンモニア酸化を行うことが発見された[40]。メタゲノム解析は、アンモニアモノオキシゲナーゼを有すタウム古細菌︵亜硝酸古細菌︶が、海洋、土壌何れにおいてもアンモニア酸化細菌を遥かに上回ることまで示している[110][111]。これにより、アンモニア酸化は細菌が行うというこれまでの常識が崩された。農業用土壌では、アンモニア酸化細菌と古細菌は、アンモニア濃度やpH、土壌深度等に応じて住み分けを行っているようである[112]。亜硝酸はその後別の細菌によって硝酸に酸化され、植物など他の生物によって利用される。この過程に古細菌が関与するという報告はない。亜硝酸古細菌はまた、温室効果ガスである一酸化二窒素を放出する[113]。一方で、亜硝酸古細菌はメタンの酸化分解を行うという報告もある。 また、硫黄循環においては、鉱物から硫黄を遊離する過程で古細菌が働く。例えばSulfolobusは単体硫黄を酸化することによって増殖する。この活動によって生成する硫酸が環境汚染を引き起こすことがあるが[114]、硫黄循環においては、硫黄を植物に利用できる形に変化させるという点において重要である。ただし、この反応は細菌の一部も同様に起こすことができる。 メタン生成菌は炭素循環において独特の地位を占める。これらの古細菌が持つ水素や有機酸をメタンとして除去する能力は、嫌気条件での有機物代謝の最終段階を担っている。この過程は﹁メタン菌﹂において詳しい。天然ガスやメタンハイドレートも、その生成にはメタン菌が関与している。しかしながら、メタンの温室効果は二酸化炭素の21倍強く、地球温暖化寄与率は18%に達する[115]。メタン菌は地球上におけるメタン放出量の少なくとも2/3以上を占めると考えられている。水田や反芻動物から放出されるメタンも、元を辿ればほぼ全てがメタン生成菌由来である。なお、古細菌の中には、硫酸還元細菌と共生し、嫌気条件下でメタンを硫化水素と二酸化炭素に分解する系統も存在する[116]。 2015年には、植物プランクトンにとって重要な補因子である、海洋のビタミンB12生産の大部分をタウム古細菌が担うと報告された[117]。 一部の古細菌は光エネルギーの利用も行うようである。バクテリオクロロフィルを使った光合成は知られていないものの[注 24]、高度好塩菌やMarine group IIが保有する、バクテリオロドプシンやプロテオロドプシンは、光駆動プロトンポンプの機能を持つ[118][119]。地球上における光エネルギーの利用はバクテリオクロロフィルを含むクロロフィル型が主だと考えられてきたが、細菌を含めたプロテオロドプシンによるエネルギー生産量はその1割にも達すると見積もられており[120]、古細菌Marine group IIもその一部を占める。ただしこれらは炭素固定を行わない光従属栄養生物と考えられる。他生物との関係[編集]

牛などの反芻動物の消化器官には大量のメタン菌が存在しており、メタ ンを生成している。 他の生物との関係は、相利共生か片利共生のどちらかである。病原性の古細菌は確実なものは知られていない[121][122][注 25]。寄生の例としては、"Ca. Nanoarchaeum equitans"が、別の古細菌Ignicoccus hospitalisとの共存下のみで増殖する例がある[124]。 メタン菌と原虫の相互作用は相利共生として理解されている。これは、反芻動物や白アリの消化器官でセルロースを分解するために働く[125]。原虫は嫌気条件でエネルギーを得るためにセルロースを代謝し、その過程で廃棄物として水素を放出する。水素が蓄積すると原虫は増殖が阻害される。メタン菌はこの水素の除去を行い、原虫は効率的なエネルギー生産を可能とする[126]。有機酸や水素を放出する嫌気性細菌との間にも同様の共生関係が成り立つ。この関係は古細菌同士でも可能で、メタン菌であるMethanopyrus kandleriが存在すると、水素を放出するPyrococcus furiosusはM. kandleriに付着してバイオフィルムを形成する[127]。こういった関係はいくつかの原虫、菌類でより進展しており、例えばPlagiopyla frontata、Nyctotherus ovalisなどは細胞内に共生メタン菌を保有する[128][129]。 ヒトの体内で最も一般的なのはMethanobrevibacter smithiiというメタン菌である[130]。このメタン菌を保有するマウスは体重増加が報告されており[131]、栄養吸収や肥満に関係している可能性がある。高齢者に多い[132]Methanomassiliicoccus luminyensisは、有害なメチルアミンを無害なメタンに分解する[133]。一方、口腔内に存在するMethanobrevibacter oralisについては、免疫応答に関与することで歯周病を悪化させる危険因子であるとされている[134][135][136]。メタン菌はヒトにとって、有益でも有害でもありうる[137]。 メタン菌以外では、海綿Axinella mexicanaとタウム古細菌"Ca. Cenarchaeum symbiosum"の関係が報告されている[138]。ヒトによる利用[編集]

ドイツのバイオガス製造プラント 汚水処理施設やバイオガスの製造において、メタン菌によるメタン発酵が行われている。この他菌体を直接利用するものはあまりないが、キムチ[139][注 26]や魚醤[140][141]からHalococcusやHalobacteriumに代表される高度好塩菌が検出されることがあり、腐敗や発酵に関与する。好熱好酸菌は硫化水素や金属の処理目的に研究されている[142][143]。 一方、新しい遺伝子資源としても注目を集めてきた[144]。Pyrococcus furiosusやThermococcus kodakaraensisなどに由来するDNAポリメラーゼ︵Pfuポリメラーゼ、KODポリメラーゼ︶は、Taqポリメラーゼ︵細菌Thermus aquaticus由来︶に比べ複製正確性が高く、PCRになくてはならない酵素の一つである[145]。タンパク質が結晶化しやすく、真核生物のホモログあるいは新規酵素を多数持つことから、タンパク質の構造研究にもしばしば使用される[146]。これまでのところあまり実用化されていないが、CRISPR/Cas[注 27]や抗生物質[147]など未利用の遺伝子資源も存在する。古細菌の起源と系統的位置[編集]

岩部らの論文[148]の概略。全生物を対象にした系統樹では外群が 設定できないため、無根系統樹しか得られない。EF-Tu/1α、EF-G/2を使用しても、それぞれ単独では無根系統樹になる。しかし、この2つの遺伝子は元々は1つの遺伝子が重複し、その後別々に進化したことが分かっており、互いに相手を外群として使用することで系統解析が可能である。それにより、共通祖先は最初細菌と古細菌︵+真核生物︶に分かれたことが分かった。 (1):EF-Tu/1α、EF-G/2それぞれの無根系統樹 (2):EF-Tu/1α、EF-G/2を合わせた系統樹 (3):互いに相手を外群として折り曲げた系統樹 共通祖先、原始生命体、生命の起源なども参照 地球の歴史は45億4000万年前までさかのぼる[149]。また、海洋も44億年前までには形成された可能性がある[150]。生命の起源は不明であるが、西オーストラリア州ジャックヒルズから見つかった41億年前に形成されたジルコンから、異常に同位体比率の偏った炭素が発見されており、生命活動の痕跡の可能性が指摘されている[注 28][152]。起源が38億年より遡る場合、後期重爆撃期との関係も重要になる。少なくとも35億年前まで[153]、そしておそらくそれよりももっと以前から生命は存在していたと考えられている。 地球上に誕生した生命が細菌と古細菌に分かれた理由についても不明なところが多い。共通祖先が分かれて古細菌が出現した時期についてはいくつか説が出されていて、例えば35億年前の地層から異常にC13の比率が低いメタンが発見されており、メタン菌が当時存在していた、つまりこの時期までには古細菌が出現していたと考えることもできる[154]。系統解析からの推定では、38億年前[155]、42億年以前[156]、44億年以前[157]、などという数字も出ているが、あまりにも古い時代のため、不確実性が大きい。細菌との関係[編集]
細菌と古細菌は共通の祖先を有する可能性が高い[158]。DNAやたんぱく質が脂質二重膜よりなる細胞膜につつまれる構造は全生物に共通する。コドンなど基本的な遺伝の仕組みも共通している。一方で、細胞膜の成分やDNA複製系は全く異なっており、両者が分かれたのは非常に古いと推定される。 ウーズが最初に描き出した系統樹は無根系統樹であり、共通祖先がどの位置にあるかは確定できないものであった。例えば、真核生物と原核生物が最初に分かれた後、原核生物が細菌と古細菌に分かれた、あるいは細菌と古細菌が最初に分かれた後、細菌から真核生物が進化した、もしくは細菌の中に共通祖先があり、細菌の一系統から古細菌や真核生物が進化した、など様々に解釈できる。この問題は、16S rRNAなどの遺伝子の単純な解析では導き出せないが、共通祖先以前に重複、その後独立して進化した遺伝子を比較することで可能となる。1989年に、H+-ATPアーゼ、伸長因子、リンゴ酸デヒドロゲナーゼ、乳酸デヒドロゲナーゼなど共通祖先以前に分かれた遺伝子を用い、共通祖先がまず細菌と古細菌類に分岐したことが明らかになった[148][159]。時期的には前述のとおり、30億年よりも遥かに遡る古い時代に起こったと考えられる。 これ以外の説としては、共通祖先は現代的な細胞膜やDNA複製の仕組みを持っていなかったとする説もある。アルカリ熱水泉の細孔で生命が誕生したとすれば、透過度の低い細胞膜は必要ではない[160]。古細菌と細菌はそれぞれ独立に細胞膜やDNA複製の仕組みを獲得し、別々に細孔から脱出したとする[160]。また、トーマス・キャバリエ=スミスらは、最初に細菌が多様化した後、細菌の一グループである放線菌の中から真核生物と古細菌の祖先が出現したネオムラ説を提唱している[161]。他の細菌との違いは、高温への適応や、細菌間での化学戦の産物であるとする[161]。放線菌は外膜の無い単膜細菌︵MD細菌︶であり、膜が一重であることが古細菌と共通する。Hsp70やグルタミン合成酵素Iなどから得られる系統樹がネオムラ説を支持するという[161]。真核生物との関係[編集]

左:3ドメイン説、右:エオサイト説。EF-1のインデルの比較では エオサイト説が支持される[162][注 29]。
29遺伝子と化石証拠に基づく系統樹︵Holly C. Betts et al., 2018[157]︶の概観。この系統樹では2分岐説が採用されている。主要系統以外は省略。古細菌の中では最初に分岐したDPANN系統や、コル古細菌などは省略した。 真核生物と古細菌は基本的な遺伝の仕組みを共有しており、この2つの生物は細菌よりも密接に関係している。16S rRNAによる分析では、古細菌が多様化するよりもはるか以前に古細菌と真核生物は分岐したため、特に真核生物に近い古細菌はいないという系統樹を描き出したが、EF-1/EF-2︵伸長因子︶を使った解析では、クレン古細菌と真核生物が単系統となる系統樹を描き出した[163]。 ウーズによる3ドメイン説では、真核生物と古細菌はそれぞれが単系統であり、互いに姉妹群であるとする[164][1]。3つの各ドメインが分かれる以前の共通祖先は遺伝の仕組みが成立していない生物であったとしており︵プロゲノート説︶、古細菌︵または古細菌に近い生物︶から真核生物が進化したわけでは無いとしていた[165]。 もう一つの有力な説は、クレン古細菌に近い生物から真核生物が進化したとする2分岐説︵2ドメイン説︶である[166][167][168][169][170]。これは1984年にレイクが提唱したエオサイト説[171][172]を原型とし、3ドメイン説よりも古いものである[注 30]。山岸らも系統樹がどちらであるにせよ、地球上の生物を細菌とアーキア︵Crenarchaeota、Euryarchaeota及びUrkaryotes[注 31]︶の2つに分けるべきと主張した[174]。2010年以降、クレン古細菌や近縁な古細菌からアクチンやESCRT、ユビキチン[175]、チューブリン[176]など真核生物様の遺伝子が発見され、更に2015年以降、クレン古細菌以上に真核生物に近縁なアスガルド古細菌が発見されたことで[68][177]、2ドメイン説が有力になりつつある。ただし、真核生物のゲノム全体を見た場合、真核生物は細菌および古細菌由来のどちらの遺伝子も併せ持っており[178]、それが真核生物の起源の解明を複雑なものにしている︵真核生物の起源の項を参照︶。 この他にも、前述︵#細菌との関係︶の﹁ネオムラ説﹂[161]、RNAを基盤とするクロノサイトという生物に古細菌と細菌が合体した﹁ABC仮説﹂[179]、古細菌にウイルスが感染して真核生物になった﹁細胞核ウイルス起源説﹂[180]などが提案されている。他ドメインとの違い[編集]
細菌 古細菌 真核生物[2 1] 細
胞
構
造大きさ 1-10 μm (細菌と同様) 5-100 μm 細胞の移動 細菌型鞭毛、滑走[2 2] 古細菌型鞭毛[2 3] 鞭毛(チューブリン)、形状変化 組織化 単細胞、稀に群体[2 4] (細菌と同様)[2 5] 単細胞、群体、多核体、多細胞 細胞分裂 Zリング ESCRT複合体[2 6]、Zリング[2 7]、出芽[2 8] アクチンミオシン収縮環 細胞壁 ペプチドグリカンなど タンパク質など 糖鎖など 細胞膜 エステル型脂質(sn-1,2位)[2 9] エーテル型脂質(sn-2,3位) (細菌と同様) 細胞小器官 無し[2 10] (細菌と同様) 細胞核、ミトコンドリアなど多数 細胞質 細胞骨格は限定的[2 11] (細菌と同様)[2 12] 細胞骨格を持ち原形質流動有り エンドサイトーシス 起こさない (細菌と同様) 起こす 核
酸
/
蛋
白
質
関
係DNA 環状[2 13] (細菌と同様) 直線状 DNA結合タンパク HUタンパク 古細菌型ヒストン[2 14] ヒストン ゲノムサイズ 小さい (細菌と同様) 大きい DNA複製酵素 ファミリーC ファミリーB及びD[2 15] ファミリーB プロモーター プリブノーボックス (真核生物と同様) TATAボックス 転写開始機構 シグマ因子 (真核生物と同様) 転写開始前複合体 RNAポリメラーゼ 単純[2 16] (真核生物と同様)[2 17] 複雑[2 18] mRNA 修飾を受けない[2 19] (細菌と同様)[2 20] キャップ構造付加、スプライソソーム型イントロン リボソーム 50S+30S
ストレプトマイシン感受性(細菌と同様)
(真核生物と同様)[2 21]60S+40S
ジフテリア毒素感受性翻訳開始tRNA フォルミルメチオニル-tRNA (真核生物と同様) メチオニル-tRNA t-RNA イントロン無し[2 22] (真核生物と同様) イントロン有 ATP依存性プロテアーゼ FtsHなど[2 23] (真核生物と同様) プロテアソーム (一)^ ここでは、真核生物の細胞小器官のうち、細菌に由来することがほぼ確実であるミトコンドリアや葉緑体、及びそこで働く機構やタンパク質、tRNA、リボソームなどは除く。これらは細菌の特徴を一部残している。 (二)^ 線毛や匍匐、アクチンロケットなどによって移動することを言う。 (三)^ 鞭毛を回転させ推進するという点では細菌と同じである。ただし細菌の鞭毛とは構造が全く異なる。#鞭毛参照 (四)^ 粘液細菌や一部の藍藻、放線菌の中には、相当高度な群体を形成するものがある。 (五)^ 稀に融合体︵多核巨大細胞︶を形成する種が存在する。群体についても細菌と同様広く見られる。 (六)^ プロテオ古細菌︵クレン古細菌、タウム古細菌、アスガルド古細菌など︶ (七)^ ユーリ古細菌など (八)^ テルモプロテウス目のみ (九)^ 一部の好熱細菌はエーテル結合を含む脂質を持つが、その場合でも炭化水素鎖が結合しているのは1,2位であり、古細菌の脂質とは区別できる。 (十)^ 例外はチラコイドなどがある。稀にだがDNAを囲む膜を持つもの (Planctomyces) もいる。 (11)^ MreBなど今日では多数の細胞骨格が見つかっている。ただし真核生物ほど多様な機能は持たない。原核生物の細胞骨格参照。 (12)^ クレン古細菌からはクレンアクチンが見つかっている。ユーリ古細菌から発見された例は少ないが、テルモプラズマからMreBの報告がある。チューブリンホモログも存在︵オーディン古細菌、タウム古細菌︶ (13)^ 放線菌、スピロヘータの一部は直鎖状のDNAを持つ。 (14)^ クレン古細菌はテルモプロテウス目のみヒストンを持つ (15)^ クレン古細菌はファミリーDを持たない。 (16)^ サブユニット数は大腸菌で5 (17)^ サブユニット数は11〜13 (18)^ 基本的なサブユニット数は12︵RNAポリメラーゼII︶。古細菌と全てが共通する (19)^ 稀に存在するイントロンは自己スプライシング型であり、真核生物特有のスプライソソーム型は存在しない[181]。 (20)^ rRNAやtRNAには広くイントロンが知られているが、mRNAにイントロンが含まれている例は少ない。スプライソソーム型は存在せず、細菌に見られる自己スプライシング型も1例︵Methanosarcina acetivorans︶しかない。代わりに古細菌のみに存在する酵素が触媒するイントロンがある[181]。 (21)^ RNAは細菌、タンパク質は真核生物にやや類似。rRNAは細菌と同様5S、16S、26S。タンパク質︵68個︶は殆ど全てのサブユニットが真核生物と一致︵67個︶する一方、細菌とは34個しか一致しない。[90]。抗生物質感受性はどちらかと言えば真核生物寄りだが、個々の種、抗生物質の種類によって差が大きい。 (22)^ tRNAのイントロンは少数見つかっているが、全て自己スプライシング型。古細菌と真核生物にある酵素触媒型は殆どない。 (23)^ そのほかLon、Clp、HslVUなど。放線菌目のみプロテアソームも持つ︵他の放線菌綱は持たない︶。呼称[編集]
古細菌という呼称は、6界説を提唱したカール・ウーズらが名づけたArchaebacteriaの翻訳である。Archaebacteria自体は、メタン菌が太古の地球大気の主要構成成分と考えられていた二酸化炭素と水素の混合気体を基質として生育するため、Archae︵ギリシア語:αρχαία/太古・始原︶ + Bacteria︵小さな棒︶と名づけられたことに由来する[182]。1980年代に入ると、古細菌 (Archaebacteria)が真正細菌よりもむしろ真核生物に近いことが明らかになり、それ程広くは使われなかったが後生細菌 (Metabacteria; メタバクテリア) という用語が提唱された[183]。1990年になるとウーズは3ドメイン説を発表した。この際、これまでArchaebacteriaと呼ばれてきた生物群に対して、Archaeaという名称が与えられ、細菌と区別するためにbacteriaが外された[1]。以後英語圏ではArchaeaが定着した。日本でもこれに対応して細菌が外され、始原菌という和名が提唱された。しかしながら始原菌という用語はそれ程定着しなかった。現在、最も一般に使用されるのは古細菌、または英語読みのアーキア︵まれにアーケア︶で、一部の研究者の間では始原菌、ラテン語に由来するアルカエア︵アルケア︶[184][注 32]といった呼び方もされる。 このほかの呼称としては、Mendosicutes︵メンドシクテス︶[注 33]や古バクテリア類、後生細菌といった表現もみられる。いずれも古い用語であり、使用頻度は下がっている。英語圏でもまだArchaeabacteriaは使用されており、特に著名な研究者であるトーマス・キャバリエ=スミスらがこの語を用いている[2][注 34]。2分岐説︵エオサイト説︶において、アーキア︵Archaea︶を真核生物を含む範囲に拡張する場合、原核生物のみを指して古細菌と便宜上呼称する場合もある[185][186]。なお、中国語でも当初は古細菌と呼ばれていたが、﹁Archaea﹂に対しては、﹁古菌﹂[187]や﹁古生菌﹂[188]という漢字名が広く使用されている。 ﹁古細菌﹂という名称は、﹁菌﹂や﹁細菌﹂を含むが、狭義の菌類︵真菌︶や狭義の細菌︵真正細菌︶とは異なる。細菌︵Bacteria︶が菌︵Fungi︶で無いのと同様に、古細菌︵Archaea︶は菌類でも細菌でも無く、系統も全く異なっている。分類学上は菌類が真核生物ドメインに含まれ、細菌が細菌ドメイン、古細菌が古細菌ドメインを構成している。なお、ラテン語名である﹁Archaea﹂という名称も、1854年にアゴダチグモ科の化石種に与えられているが、古細菌と直接の関係はない[注 35]。発見史[編集]
古細菌︵archaebacteria︶発見の歴史は細菌︵eubacteria︶発見の歴史に並行している。今日知られているような枠組みが完成する以前は、高度好塩菌、メタン菌、好熱菌それぞれ別々の枠組みで研究が進められていた。古細菌という枠組みができたのは1977年以降である。発見[編集]
1674年、アントニ・ファン・レーウェンフックが微生物を発見して以来、徐々に研究が進んでいた。1868年には微生物の働きによりメタンが発生することを初めて確認し、1880年代には高度好塩菌の研究が始まった。これ以前にも沼などからメタンが発生すること、塩蔵の食品や塩田が赤く染まることは知られていたが、微生物によるものとは考えられていなかった[189]。明朝の本草綱目にも、天日塩の製造過程で塩水が赤く染まることが記述されている[190]。 20世紀に入ると、1922年に高度好塩菌の分離が始まり、Pseudomonas salinaria︵後のHalobacterium salinarum︶と名づけられた。翌年Serattiaに移され、その後もPseudomonasに戻されるなど分類は混乱した。1974年にようやくハロバクテリウム科にまとめられた。一方、メタン菌は存在することは分かっていたものの、酸素を極端に嫌う生物であり、1936年にやっと培養に成功し、1947年にはMethanobacterium formicicumとMethanosarcina barkeriが分離された。[191] 既に1930年頃には原核生物と真核生物の違いが認識されており、原核生物帝︵1937年︶次いで五界説モネラ界︵1969年︶が提唱された[192]。高度好塩菌とメタン菌には明らかに核がなく、以後モネラ界の枠組みに含まれることとなった。一方、好熱性の古細菌[注 36]は少し遅れ、1970年に炭鉱のボタ山から好熱好酸菌Thermoplasma acidophilumが発見された[194]。この生物は細胞壁を欠くことからマイコプラズマの仲間とされた[194]。1972年にはイエローストーン国立公園より好熱好酸菌Sulfolobus acidocaldariusが発見された[195]が、これらは別々に少し変わった生物だとして知られているに過ぎなかった。当時、メタン菌、高度好塩、Thermoplasma、Sulfolobusはそれぞれ別々の門や群に分類されていた[注 37]。 しかし、1960年頃から他の生物とは性質が異なるという報告もされ始めている。今日知られている古細菌の特徴の一つであるエーテル型脂質は、1962年に高度好塩菌Halobacterium salinarum (Halobacter cutirubrum[注 38])より発見され[196]、1972年には好熱菌Thermoplasma acidophilumも、やはり同じ脂質を持つことが判明した[197]。[198] ペプチドグリカン細胞壁を持たないという報告も1970年代にはいくつか出されている[199][182]。定義[編集]

ウーズによる16S rRNAを基にした系統樹 
左がカール・ウーズ、真ん中がラルフ・ウォルフ、右がオットー・カン ドラー。1981年 これらの生物を他の原核生物と区別した最初の人物は、イリノイ大学のカール・ウーズである。1960年代、互いに近縁な生物はタンパク質のアミノ酸配列や塩基配列が似ているという理論を背景にした分子時計や中立進化説[200]が提唱され、生物の系統解析が開始されようとしていた。原核生物では、DNA-23S rRNA分子交雑法[201][202]、5S rRNA塩基配列などといった方法が取られ始めていた[注 39]。この流れの中で、ウーズらも、ライナス・ポーリングらの研究[204]に影響を受け、1960年代後半から16S rRNAを用いて生物の分類を始めていた[205]。彼が考案・使用した方法は、16S rRNAをいくつかの小断片に切断し、対応する配列と一致する塩基の割合を比較するポリヌクレオチドカタログ法[注 40]というものだった[206]。様々な生物のrRNAを比較する中で、1976年、ウーズは同僚のウォルフ[注 41]からメタン菌のコロニーの提供を受け、そのrRNAが他の原核生物と大きく異なるという結果を得た[207]。ウーズらはさらに研究を続け、翌1977年、この結果を元に原核生物を古細菌界︵Archaebacteria。メタン菌︶と真正細菌界︵Eubacteria。その他の細菌︶に分けるべきと主張した[182]。 この時点で古細菌界はメタン菌のみを含むものであったが、1978年にメタン菌からエーテル型脂質が発見[208][209]され、古細菌の特徴の一つとして、エーテル脂質を持つ可能性が出てきた。これは、既にエーテル脂質を持つ事が知られていた高度好塩菌及び好熱菌の一部も古細菌界に含まれることを示唆した。同年、rRNA系統解析が行われ、高度好塩菌と好熱菌の一部も古細菌界に属すことが支持された[210]。しかしながら、通常の細菌と形態の殆ど変わらない生物を塩基配列データのみで分類することに抵抗は大きく、古細菌界という分類群が受け入れられるには時間がかかった。分割に反対する研究者もいた[211][212]。 1980年代以降、古細菌の研究が活発になり、この時期、真正細菌と古細菌の差異を示す研究が蓄積された。それと共に古細菌という概念も受け入れられ始めた。1982年、それまでの常識を打ち破る110℃で増殖する古細菌が発見され[213]、古細菌研究をさらに活発化させた。1989年には共通祖先以前に重複した遺伝子を用いることによって古細菌が真正細菌よりも真核生物に近いことが報告された[注 42]。ウーズはこの説を採用し、1990年には全生物を真核生物ドメイン、古細菌ドメイン、細菌ドメインの3つのグループに大別する3ドメイン説を提唱した[1]。 1996年には、超好熱性のメタン菌Methanocaldcoccus jannaschiiの全ゲノムが解読された[214][注 43]。これは古細菌として初めて、全生物の中でも4番目の解析例である[216][注 44]。先行して解読されていたインフルエンザ菌、Mycoplasma genitalium、出芽酵母との比較により、代謝系の遺伝子は細菌にやや類似、転写・複製・翻訳に関連する遺伝子は真核生物に類似するが、細菌と類似の遺伝子はわずか11〜17%しか見つからず、半分以上の遺伝子はどちらにも見つからない新規の遺伝子であった[214]。これは古細菌が、他の生物とは大きく異なることを裏付けるものであった[218]。これらの結果を受け、今日大方の微生物学者に古細菌ドメインという分類群は受け入れられている。脚注[編集]
注釈[編集]
(一)^ ラテン語名としては、トーマス・キャバリエ=スミスによる、フィル古細菌︵Filarchaeota。ESCRT-IIIフィラメントを持つことから[2]︶があるが、既にトーマス・キャバリエ=スミス本人も使用していない。 (二)^ ドメインの本来の下位分類である界は明確ではない。一応Woeseによってユーリ古細菌界とクレン古細菌界の2界に大別されている[1]ほか、クレン古細菌界を代替するものとしてプロテオ古細菌界が提案されている[3]。一方、門は国際原核生物命名規約中では定められていない︵正式な学名ではない︶ものの、Bergey's Manual 2ndは、Domain Archaeaの下にPhylum Euryarchaeota︵ユーリ古細菌門︶とPhylum Crenarchaeota︵クレン古細菌門︶を置いており、この2門については広く認められているといって良い。また、タウム古細菌門も記載種を含んでいる。本来は全て正式な学名では無いが、前述の記載種を含む3門のみ引用符で囲んでいない。その他の門は記載種を含まず、多くの場合はゲノム情報をもとに提案された分類である。ここでは門以上の分類群やクレードについても記載した。 (三)^ あくまでも相対的な話である。嫌気培養装置や高温培養装置を必要としない点では有利だが、培地の管理は"通常の"細菌に比べれば難しく︵水分が少し飛ぶだけで塩が析出したりする︶、また寒天培地上でコロニーを形成する高度好塩菌は少数派と言われている。Haloquadratum walsbyiは、発見してから20年以上純粋培養できなかった。 (四)^ 全生物1位。130℃でも耐える。タイプ株であるAV19株の限界生育温度は110℃で、この記録はStrain116株による。また、常圧に近い0.4MPaでは116℃での増殖が限界で、122℃で増殖させるには200-400気圧に加圧する必要がある[24]。細菌の最高増殖記録はAquifex aeolicusの95℃なので、高温適応の点では古細菌が圧倒的である。 (五)^ 全生物3位。2001年までは最も強アルカリで増殖できる生物であったが、現在はAlkaliphilus transvaalensis︵pH12.5。フィルミクテス門︶に更新されている。飽和塩濃度でも増殖できる高度好塩菌を兼ねる。 (六)^ 全生物1位。同属のP. torridusや近縁のFerroplasma acidiphilumもほぼ同じpHで生育可能 (七)^ 全生物1位。近縁な古細菌にも同濃度で増殖できるものがいる。 (八)^ 全生物1位。なお増殖温度も全生物6位。 (九)^ D37︵37%が生き残る限界線量︶の比較では、細菌であるRubrobacter radiotoleransの方が上回っている。 (十)^ 超好熱性の細菌は、Aquifex aeolicus︵至適増殖温度85℃︶、Thermotoga martima︵至適増殖温度80℃︶などが知られる (11)^ 細菌の鞭毛は、鞭毛の中を通って先端から構築されるが、古細菌の鞭毛は細いためそのようなことができない (12)^ ﹁sn-﹂とは立体特異的番号付けを用いた時につける接頭辞である。DL表記法で表記すると、細菌や真核生物のsn-グリセロール3-リン酸はL-グリセロール3-リン酸またはD-グリセロール1-リン酸、古細菌のsn-グリセロール1-リン酸はL-グリセロール1-リン酸またはD-グリセロール3-リン酸となる。 (13)^ これを超えるものとして、未培養系統ではあるが、LC_2︵ヘイムダル古細菌︶の推定ゲノムサイズが6.35Mbp[68]。 (14)^ 真核生物ではヒストンH2A、H2Bが追加され、八量体になっている。1周期のDNAはおおよそ147bp[76]。 (15)^ ヘイムダル古細菌からN末テイルを持つヒストン相同配列が発見されているが、まだ研究されておらず、翻訳後修飾を受ける証拠はない[76]。 (16)^ 高度好塩菌Halobacterium salinarum NRC-1ではアフィジコリンによって増殖阻害を受けること︵アフィジコリンはBファミリーDNAポリメラーゼの活性を阻害する︶、PolB、PolDのどちらの破壊株も得られないことからともに複製に必須と考えられた[81]。PyrococcusやMethanococcusは、PolBの破壊株は得られるが、PolDは破壊出来ない[81]。一方、クレン古細菌はPolDを欠き、複数のPolBを保有する[81]。 (17)^ 2014年8月現在。全ゲノムが解読されている古細菌150種中。一方、細菌の45%にも存在する。[87] (18)^ RNAポリメラーゼは11〜13のサブユニットより構成され、真核生物のRNAポリメラーゼII︵古細菌と共通する12のサブユニットより構成︶とよく似ている。細菌ではサブユニット数は5で、いくつかのサブユニットが省かれている。[89]。転写機構もRNAポリメラーゼIIと良く似ており[84]、転写開始前複合体︵真核生物よりもサブユニット数が少ない︶により転写が開始されると考えられている。 (19)^ リボソームの大きさは細菌と同様70S︵50S+30S︶。rRNAは16S、5S、23Sの3つで、真核生物の5.8Sに相当する配列は23Sに組み込まれている。また、一般に16S rRNAにはシャイン・ダルガノ配列が認められる。タンパク質はユーリ古細菌で58〜63、クレン古細菌で66〜68の構成。その内67種については真核生物に対応するrタンパクが存在する︵細菌と共通するのは33~34しかない︶[90]。 (20)^ ジフテリア毒素︵真核生物︶やアニソマイシン︵真核生物︶はほぼ感受性、キロマイシン︵細菌︶やストレプトマイシン︵細菌︶にはほぼ非感受性であるが、例外としてクロラムフェニコール︵細菌︶は効果がある。クロラムフェニコールはMethanosphaera stadtmanaeには良く効くが、MethanobrevibacterやMethanomassiliicoccusは中程度の耐性を持ち、SulfolobusやHalobacteriumにはあまり効かない[91]。リボソームをターゲットとする抗生物質に対する感受性は各古細菌種によって多様性が大きい。なお、ジフテリア毒素は、ほぼ全ての古細菌が感受性を示すため、かつて古細菌と細菌を区別する手っ取り早い手段の一つとして使われていた。 (21)^ テルモコックス綱古細菌では、細菌や真核生物が糖のリン酸化にATPを消費するところ、ADPを消費する形態になっている。また、グリセルアルデヒド3-リン酸から3-ホスホグリセリン酸への経路が、フェレドキシンオキシドレダクターゼ、または非リン酸化グリセルアルデヒド3-リン酸デヒドロゲナーゼでバイパスされている。この反応では還元型フェレドキシンないし、NADPHが得られるが、ホスホグリセリン酸キナーゼによるATP合成が起こらないため、後者では通常型EM経路よりもATPの生成が1分子少ない。最終段階のホスホエノールピルビン酸からピルビン酸への変換の際、通常型EM経路と異なりホスホエノールピルビン酸シンターゼが働き、AMPとピロリン酸からATPが生成される。これらの形態は、熱に弱い反応中間体を経ないことで高温に適応していると考えられる。[95] (22)^ 片方が繊毛を持っていれば組み換えは起こるようである。両方が繊毛を失った場合は組み換えが起こらない (23)^ Ferroplasma acidarmanusとFerroplasma type II︵16S rRNA配列99.2%︶の間にも組み換えは起こるが、Ferroplasma acidarmanus同士よりもはるかに少ない組み換えしか見られない (24)^ 1例のみだが、未培養系統の古細菌MCG-A︵Bathyarchaeota class 6︶からバクテリオクロロフィルa合成酵素を含む配列が報告されている[14]。このar-bchG︵古細菌型バクテリオクロロフィルa合成酵素︶は、細菌の持つバクテリオクロロフィルa合成酵素とは系統的に離れており、最も同一性の高いRhodospirillum rubrumとの比較でも27%しか一致しない。大腸菌で発現させた実験によると、実際にこの遺伝子は機能するようである。 (25)^ 古細菌を病気の直接の原因とする報告は殆どない。感染症と関係が深い細菌とは対照的である。僅かに、特殊な脳脊髄炎の患者から、未知の古細菌DNA配列が多数検出された例がある[123]が、この1例のみしか報告が無く、培養もできなかったことから確定していない。 (26)^ これらの食品は塩濃度が低すぎるため、使用した食塩由来のコンタミの可能性もある。 (27)^ なお、現在ゲノム編集に利用されているCRISPR/Cas9は細菌由来である (28)^ 生物は、物質を代謝する際に特定の同位体を好むことがよくあるため、炭素や硫黄の同位体比率が大きくずれた分子は生命活動の証拠となりうる︵ただし、非生物的に同様の物質が形成されることもあるため慎重に検討する必要がある︶。37億7000万年前︵42億8000万年前︶の熱水噴出孔で形成されたと考えられる岩石からも同様の痕跡が見つかっている[151] (29)^ 真核生物とクレン古細菌︵及びタウム古細菌やアスガルド古細菌などにも︶には、EF-1︵伸長因子-1︶に11アミノ酸残基の挿入がある。3ドメイン説が正しい場合、クレン古細菌と真核生物にそれぞれ独立して同じ位置に同じ長さの配列が挿入されたと考える必要がある。一方、エオサイト説が正しい場合、クレン古細菌と真核生物の祖先に11アミノ酸残基が挿入されたと考えれば、挿入が1回で済みはるかに合理的である。 (30)^ 元々のエオサイト説では、生物全体をEubacteria、Archaeabacteria、Eocyte、Eukaryotesの4界に分けた。Archaeabacteriaは後のユーリ古細菌、Eocyteは後のクレン古細菌に相当する。なお、EocyteはのちにTACKなど近縁種を含むように拡張されている[8]。 (31)^ Urkaryotesは真核生物の遺伝子、または真核生物の本体を指している。また、Crenarchaeota、Euryarchaeotaを総称してArchaeabacteriaと呼ぶとした。[173] (32)^ ラテン語をそのまま仮名転写するとアルカエア。アルケアは中世ラテン語に近い (33)^ "Mendosicutes Gibbons & Murray 1978"。1984年発行のBergey's Manualなどでこの表現が見られる。ここでは原核生物界を構成する4つの門の一つとして位置づけられていた。 (34)^ トーマス・キャバリエ=スミスは近年Archaeabacteriaを使用しているが、Metabacteria︵後生細菌︶の方が適切であるとも主張している[161] (35)^ 界が異なるため同名でも命名規約上の問題はない。なお、同様にBacteriaという学名を持つナナフシも存在する (36)^ 好熱性の細菌は20世紀前半に好熱性の“Bacillus”︵後のGeobacillus︶などが発見されていた。また、Thermoplasmaよりも少し前の1969年には、イエローストーン国立公園より、高度好熱性の細菌Thermus aquaticus︵至適生育温度72°C︶が報告されている[193] (37)^ Archaeabacteria提唱後に出版された文献であるが、﹃五つの王国﹄︵リン・マーギュリス著︶や、古いBergey's Manual中では、いわゆる古細菌類が様々な細菌グループの中に散らばって分類されている。︵Thermoplasmaがマイコプラズマ類、高度好塩菌がグラム陰性好気性細菌群︵シュードモナス門︶、Sulfolobusが化学合成細菌門など。︶ (38)^ "Halobacter cutirubrum"はHalobacterium salinarumに統合されており、現在は無効名 (39)^ 例えば、日本では1968年の東京大学の高橋による、DNA-23S rRNAハイブリッド法を用いたBacillusに関する研究がある[203]。 (40)^ 16S rRNAは細胞中に大量に存在するため、PCRが開発されていない当時でも配列の比較が可能だった。ただし、当時はRNA配列の全長を決定するのが困難だったため、16S rRNAをいくつかの小断片に切断し、対応する配列と一致する塩基の割合を比較することによって系統解析を行っていた[206] (41)^ ラルフ・ウォルフ。当時、ウーズと同じイリノイ大学の同じ階でメタン菌の研究をしていた。Methanothermobacter wolfeii、Methanobacterium wolfeiといったメタン菌に献名されている。なお、提供を受けたメタン菌は、Methanobacterium thermoautorophicum︵現Methanothermobacter thermautotrophicus︶、Methanobacterium ruminantium M-1︵現Methanobrevibacter ruminantium︶、Methanobacterium sp. JR-1︵後にMethanogenium cariaciとして記載︶、Methanosarcina barkeriの4つ。 (42)^ この当時描かれていた系統樹は、全生物を対象にしたため外群が設定できず、それぞれの生物から系統樹が一点に収束する無根系統樹しか描けなかった。これでは、古細菌が真正細菌、真核生物何れに近縁なのか、何れとも近縁ではないのかといった情報を得ることができない。Lakeや堀は、それぞれ独自に5S rRNAから得られる無根系統樹を折り曲げて共通祖先を作ったが、根本的な解決とはならなかった︵両者が描く系統樹は違っていた︶。これを解決したのが、岩部らの研究で、両者はそれぞれ独自に共通祖先以前に重複した遺伝子を選び出し、その一方を外群として用いることにより、系統樹に根をつけることに成功した。これにより得られた系統樹は、共通祖先がまず真正細菌と古細菌類に分岐したこと、その後古細菌と真核生物の分岐が起きたことを支持するものであった。[148][159] (43)^ 旧名Methanococcus jannaschii。2002年にMethanocaldcoccus属に変更となった[215]。 (44)^ 同年にSynechocystis sp. PCC6803の解析が行われており[217]、こちらの方が先行している可能性がある。その場合5番目。出典[編集]
(一)^ abcdeWoese, C.R., et al. (1990). “Towards a natural system of organisms: proposal for the domains Archaea, Bacteria, and Eucarya”. Proc. Natl. Acad. Sci. U S A 87 (12): 4576–9. PMID 2112744. (二)^ abcCavalier-Smith, T. (2014). “The neomuran revolution and phagotrophic origin of eukaryotes and cilia in the light of intracellular coevolution and a revised tree of life”. Cold Spring Harb. Perspect. Biol. 6 (9). doi:10.1101/cshperspect.a016006. PMID 25183828. (三)^ abcPetitjean, C., et al. (2014). “Rooting the Domain archaea by phylogenomic analysis supports the foundation of the new kingdom proteoarchaeota”. Genome. Biol. Evol. 7 (1): 191-204. doi:10.1093/gbe/evu274. PMID 25527841. (四)^ 英語発音: [ɑrˈkiːə] ( 音声ファイル)
(五)^ 山岸明彦﹁生命の進化と古細菌﹂﹃蛋白質核酸酵素﹄第54巻第2号、2009年、108-113頁。
(六)^ 石野, 跡見2017、195頁。
(七)^ Castelle, C.J., Banfield, J.F. (2018). “Major New Microbial Groups Expand Diversity and Alter our Understanding of the Tree of Life”. Cell 172 (6): 1181-1197. doi:10.1016/j.cell.2018.02.016. PMID 29522741.
(八)^ abJames A. Lake (2015). “Eukaryotic origins”. Philos. Trans. R. Soc. Lond. B. Biol. Sci. 370 (1678). doi:10.1098/rstb.2014.0321. PMID 26323753.
(九)^ abLionel Guy, Thijs J.G. Ettema (2011). “The archaeal ‘TACK’ superphylum and the origin of eukaryotes”. Trends in Microbiology 19 (12): 580-587.
(十)^ Nunoura, T., et al. (2010). “Insights into the evolution of Archaea and eukaryotic protein modifier systems revealed by the genome of a novel archaeal group”. Nucleic Acids Research 39 (8): 3204-23. doi:10.1093/nar/gkq1228. PMID 21169198.
(11)^ Barns, S. M., et al. (1996). “Perspectives on archaeal diversity, thermophily and monophyly from environmental rRNA sequences”. Proc Natl Acad Sci U S A 93 (17): 9188–9193. PMID 8799176.
(12)^ Elkins, J. G., et al. (2008). “A korarchaeal genome reveals insights into the evolution of the Archaea”. Proc Natl Acad Sci U S A 105 (23): 8102–7. PMID 18535141.
(13)^ Evans, P. N., et al. (2015). “Methane metabolism in the archaeal phylum Bathyarchaeota revealed by genome-centric metagenomics”. Nucleic Acids Research 350 (6259): 434-8. doi:10.1126/science.aac7745. PMID 26494757.
(14)^ abMeng, J., et al. (2009). “An uncultivated crenarchaeota contains functional bacteriochlorophyll a synthase”. ISME J. 3 (1): 106-16. doi:10.1038/ismej.2008.85. PMID 18830277.
(15)^ Takai, K., Horikoshi, K. (2016). “Genetic diversity of archaea in deep-sea hydrothermal vent environments”. Genetics 152 (4): 1285-97. PMID 10430559.
(16)^ abYoussef, N. H., et al. (2015). “Insights into the metabolism, lifestyle and putative evolutionary history of the novel archaeal phylum 'Diapherotrites'”. ISME J. 9 (2): 447-60. doi:10.1038/ismej.2014.141. PMID 25083931.
(17)^ abcBaker, B. J., et al. (2010). “Enigmatic, ultrasmall, uncultivated Archaea”. Proc. Natl. Acad. Sci. U S A. 107 (19): 8806-11. doi:10.1073/pnas.0914470107. PMID 20421484.
(18)^ Comolli, L. R., et al. (2009). “Three-dimensional analysis of the structure and ecology of a novel, ultra-small archaeon”. ISME J. 3 (2): 159-67. doi:10.1038/ismej.2008.99. PMID 18946497.
(19)^ abNarasingarao, P., et al. (2012). “De novo metagenomic assembly reveals abundant novel major lineage of Archaea in hypersaline microbial communities”. ISME J. 6 (1): 81-93. doi:10.1038/ismej.2011.78. PMID 21716304.
(20)^ Waters, E., et al. (2003). “The genome of Nanoarchaeum equitans: insights into early archaeal evolution and derived parasitism”. Proc Natl Acad Sci U S A 100 (22): 12984–8. PMID 14566062.
(21)^ Todd Lowe (2009年). “Why Sequence Archaeal transcriptomes?” (英語). 米国エネルギー省共同ゲノム研究所. 2009年11月2日閲覧。
(22)^ Valentine, D. L. (2007). “Adaptations to energy stress dictate the ecology and evolution of the Archaea”. Nat. Rev. Microbiol. 5 (4): 316–23. doi:10.1038/nrmicro1619. PMID 17334387.
(23)^ Pikuta, E. V., et al. (2007). “Microbial extremophiles at the limits of life”. Crit. Rev. Microbiol. 33 (3): 183–209. doi:10.1080/10408410701451948. PMID 17653987.
(24)^ abTakai, K., et al. (2008). “Cell proliferation at 122°C and isotopically heavy CH4 production by a hyperthermophilic methanogen under high-pressure cultivation”. Proc. Natl. Acad. Sci. U S A 105: 10949-54. doi:10.1073/pnas.0712334105.
(25)^ Schleper, C., et al. (1995). “Picrophilus gen. nov., fam. nov.: a novel aerobic, heterotrophic, thermoacidophilic genus and family comprising archaea capable of growth around pH 0”. J. Bacteriol. 177: 7050–7059. PMID 8522509.
(26)^ Sorokin, D.Y., et al. (2018). “Methanonatronarchaeum thermophilum gen. nov., sp. nov. and Candidatus Methanohalarchaeum thermophilum', extremely halo(natrono)philic methyl-reducing methanogens from hypersaline lakes comprising a new euryarchaeal class Methanonatronarchaeia classis nov.”. Int. J. Syst. Evol. Microbiol. 68 (7): 2199-2208. doi:10.1099/ijsem.0.002810. PMID 29781801.
(27)^ Schleper, C., et al. (1995). “Picrophilus gen. nov., fam. nov.: a novel aerobic, heterotrophic, thermoacidophilic genus and family comprising archaea capable of growth around pH 0”. J. Bacteriol. 177: 7050–7059. PMID 8522509.
(28)^ Jolivet, E., et al. (2003). “Thermococcus gammatolerans sp. nov., a hyperthermophilic archaeon from a deep-sea hydrothermal vent that resists ionizing radiation”. Int. J. Syst. Evol. Microbiol. 53: 847-51. doi:10.1099/ijs.0.02503-0. PMID 12807211.
(29)^ ab石野, 跡見2017、26頁。
(30)^ ab石野, 跡見2017、27頁。
(31)^ Christa Schleper (2007). “Diversity of Uncultivated Archaea: Perspectives From Microbial Ecology and Metagenomics”. Archaea: Evolution, Physiology, and Molecular Biology: 39-50. doi:10.1002/9780470750865.ch4.
(32)^ Teske, A., Sørensen, K. B. (2008). “Uncultured archaea in deep marine subsurface sediments: have we caught them all?”. ISME J. 2 (1): 3–18. doi:10.1038/ismej.2007.90. PMID 18180743.
(33)^ Lipp, J. S., et al. (2008). “Significant contribution of Archaea to extant biomass in marine subsurface sediments”. Nature 454 (7207): 991. doi:10.1038/nature07174. PMID 18641632.
(34)^ López-García, P., et al. (2001). “Diversity of free-living prokaryotes from a deep-sea site at the Antarctic Polar Front”. FEMS Microbiol. Ecol. 36 (2–3): 193–202. PMID 11451524.
(35)^ DeLong, E. F., Pace, N. R. (2001). “Environmental diversity of bacteria and archaea”. Syst. Biol. 50 (4): 470–8. doi:10.1080/106351501750435040. PMID 12116647.
(36)^ DeLong, E. F. (1998). “Everything in moderation: archaea as 'non-extremophiles'”. Curr. Opin. Genet. Dev. 8 (6): 649–54. doi:10.1016/S0959-437X(98)80032-4. PMID 9914204.
(37)^ Brochier-Armanet, C., et al. (2008). “Mesophilic crenarchaeota: proposal for a third archaeal phylum, the Thaumarchaeota”. Nature Reviews Microbiology 6 (3): 245–52. doi:10.1038/nrmicro1852. PMID 18274537.
(38)^ Konneke, M., et al. (2005). “Isolation of an autotrophic ammonia-oxidizing marine archaeon”. Nature 437: 543–546. PMID 16177789.
(39)^ abStieglmeier, M., et al. (2014). “Nitrososphaera viennensis gen. nov., sp. nov., an aerobic and mesophilic, ammonia-oxidizing archaeon from soil and a member of the archaeal phylum Thaumarchaeota”. Int. J. Syst. Evol. Microbiol. 64: 2738-52. doi:10.1099/ijs.0.063172-0. PMID 24907263.
(40)^ abQin, W., et al. (2017). “Nitrosopumilus maritimus gen. nov., sp. nov., Nitrosopumilus cobalaminigenes sp. nov., Nitrosopumilus oxyclinae sp. nov., and Nitrosopumilus ureiphilus sp. nov., four marine ammonia-oxidizing archaea of the phylum Thaumarchaeota”. Int. J. Syst. Evol. Microbiol. 67 (12): 5067-5079. doi:10.1099/ijsem.0.002416. PMID 29034851.
(41)^ Jung, M.Y., et al. (2018). “Nitrosarchaeum koreense gen. nov., sp. nov., an aerobic and mesophilic, ammonia-oxidizing archaeon member of the phylum Thaumarchaeota isolated from agricultural soil”. Int. J. Syst. Evol. Microbiol.: 3084-3095. doi:10.1099/ijsem.0.002926. PMID 30124400.
(42)^ Mincer, T.J. et al. (2007). “Quantitative distribution of presumptive archaeal and bacterial nitrifiers in Monterey Bay and the North Pacific Subtropical Gyre”. Environ. Microbiol. 9 (5): 1162-75. doi:10.1111/j.1462-2920.2007.01239.x. PMID 17472632.
(43)^ 古賀1998、79頁。
(44)^ Burns, D. G., et al. (2007). “Haloquadratum walsbyi gen. nov., sp. nov., the square haloarchaeon of Walsby, isolated from saltern crystallizers in Australia and Spain”. Int J Syst Evol Microbiol 57: 387-92. doi:10.1099/ijs.0.64690-0.
(45)^ Golyshina, O. V., et al. (2000). “Ferroplasma acidiphilum gen. nov., sp. nov., an acidophilic, autotrophic, ferrous-iron-oxidizing, cell-wall-lacking, mesophilic member of the Ferroplasmaceae fam. nov., comprising a distinct lineage of the Archaea”. Int. J. Syst. Evol. Microbiol. 50 Pt 3 (3): 997–1006. PMID 10843038.
(46)^ Kuwabara, T., et al. (2005). “Thermococcus coalescens sp. nov., a cell-fusing hyperthermophilic archaeon from Suiyo Seamount”. Int. J. Syst. Evol. Microbiol. 55 (Pt 6): 2507–14. doi:10.1099/ijs.0.63432-0. PMID 16280518.
(47)^ 古賀1998、35-36頁。
(48)^ Huber H, et al. (2000). “Ignicoccus gen. nov., a novel genus of hyperthermophilic, chemolithoautotrophic Archaea, represented by two new species, Ignicoccus islandicus sp. nov. and Ignicoccus pacificus sp. nov.”. Int. J. Syst. Evol. Microbiol. 50: 2093–100. PMID 11155984.
(49)^ Küper, U., et al. (2010). “Energized outer membrane and spatial separation of metabolic processes in the hyperthermophilic Archaeon Ignicoccus hospitalis”. Proc. Natl. Acad. Sci. U S A 107 (7): 3152-6. doi:10.1073/pnas.0911711107.
(50)^ Heimerl, T., et al. (2017). “A Complex Endomembrane System in the Archaeon Ignicoccus hospitalis Tapped by Nanoarchaeum equitans”. Front. Microbiol. 8. doi:10.3389/fmicb.2017.01072.
(51)^ Nickell, S., et al. (2003). “Pyrodictium cannulae enter the periplasmic space but do not enter the cytoplasm, as revealed by cryo-electron tomography”. J. Struct. Biol. 141 (1): 34–42. doi:10.1016/S1047-8477(02)00581-6. PMID 12576018.
(52)^ Horn, C., et al. (1999). “In vivo observation of cell division of anaerobic hyperthermophiles by using a high-intensity dark-field microscope”. J. Bacteriol. 181 (16): 5114–8. PMC 94007. PMID 10438790.
(53)^ abcdeKlingl, A. (2014). “S-layer and cytoplasmic membrane – exceptions from the typical archaeal cell wall with a focus on double membranes”. Front. Microbiol. 5. doi:10.3389/fmicb.2014.00624. PMID 25505452.
(54)^ 古賀1998、262-264頁。
(55)^ 古賀1998、258-261頁。
(56)^ Kreisl, P., Kandler, O. (1986). “Chemical structure of the cell wall polymer of Methanosarcina”. System Appl. Microbiol. 7 (2-3): 293–9.
(57)^ 古賀1998、261-262, 264-265頁。
(58)^ Kinosita, Y., et al. (2016). “Direct observation of rotation and steps of the archaellum in the swimming halophilic archaeon Halobacterium salinarum”. Nat. Microbiol. 1. doi:10.1038/nmicrobiol.2016.148. PMID 27564999.
(59)^ Matzke, N. J. (2006年9月5日︵アップデータ︶). “Evolution in (brownian) space: a model for the origin of the bacterial flagellum” (英語). 2008年4月5日閲覧。
(60)^ Ng, S. Y., et al. (2006). “Archaeal flagella, bacterial flagella and type IV pili: a comparison of genes and posttranslational modifications”. J. Mol. Microbiol. Biotechnol. 11 (3-5): 167–91. PMID 16983194.
(61)^ Metlina, A. L. (2004). “Bacterial and archaeal flagella as prokaryotic motility organelles”. Biochemistry (Mosc) 69 (11): 1203–12. PMID 15627373.
(62)^ Villanueva, Laura; von Meijenfeldt, F. A. Bastiaan; Westbye, Alexander B.; Yadav, Subhash; Hopmans, Ellen C.; Dutilh, Bas E.; Damsté, Jaap S. Sinninghe (2021-01). “Bridging the membrane lipid divide: bacteria of the FCB group superphylum have the potential to synthesize archaeal ether lipids” (英語). The ISME Journal 15 (1): 168-182. doi:10.1038/s41396-020-00772-2. ISSN 1751-7370. PMC 7852524. PMID 32929208.
(63)^ Coleman, Gareth A; Pancost, Richard D; Williams, Tom A (2019-02-08). “Investigating the Origins of Membrane Phospholipid Biosynthesis Genes Using Outgroup-Free Rooting”. Genome Biology and Evolution 11 (3): 883-898. doi:10.1093/gbe/evz034. ISSN 1759-6653.
(64)^ 古賀1998、214, 265-272頁。
(65)^ Hixon, W. G., Searcy, D. G. (1993). “Cytoskeleton in the archaebacterium Thermoplasma acidophilum? Viscosity increase in soluble extracts”. Biosystems 29 (2-3): 151–60. PMID 8374067.
(66)^ “古細菌、アルケアから真核生物進化の機能的進化研究”. 論文ウォッチ. AASJ (2018年10月9日). 2018年10月9日閲覧。
(67)^ Akıl, C., et al. (2018). “Genomes of Asgard archaea encode profilins that regulate actin”. Nature. doi:10.1038/s41586-018-0548-6. PMID 30283132.
(68)^ abcdZaremba-Niedzwiedzka, K., et al. (2017). “Asgard archaea illuminate the origin of eukaryotic cellular complexity”. Nature 541 (7637): 353–358. doi:10.1038/nature21031.
(69)^ KEGG: 生命システム情報統合データベース (20). “Methanothermus fervidus” (英語/一部日本語). 2013年11月16日閲覧。
(70)^ Waters, E., et al. (2003). “The genome of Nanoarchaeum equitans: insights into early archaeal evolution and derived parasitism”. Proc. Natl. Acad. Sci. U S A 100 (22): 12984–8. PMID 14566062.
(71)^ Galagan JE, et al. (2002). “The genome of M. acetivorans reveals extensive metabolic and physiological diversity”. Genome Res 12 (4): 532–42. PMID 11932238.
(72)^ Pratas, D., Pinho, A. (2017). “On the Approximation of the Kolmogorov Complexity for DNA Sequences”. Pattern Recognition and Image Analysis 10255: 259–266. doi:10.1007/978-3-319-58838-4_29.
(73)^ Musgrave, D. R., et al. (1991). “DNA binding by the archaeal histone HMf results in positive supercoiling”. Proc Natl Acad Sci U S A 88 (23): 10397–401. PMID 1660135.
(74)^ Fahrner, R. L., et al. (2001). “An ancestral nuclear protein assembly: crystal structure of the Methanopyrus kandleri histone”. Protein Sci. 10 (10): 2002–7. PMID 11567091.
(75)^ Pereira, S.L., et al. (1997). “Archaeal nucleosomes”. Proc. Natl. Acad. Sci. USA. 94: 12633-12637. PMID 9356501.
(76)^ abcMattiroli, F., et al. (2017). “Structure of histone-based chromatin in Archaea”. Science 357: 609-612. doi:10.1126/science.aaj1849.. PMID 28798133.
(77)^ DeLange, R. J., et al. (1981). “A histone-like protein (HTa) from Thermoplasma acidophilum. II. Complete amino acid sequence”. J. Biol. Chem. 256 (2): 905–11. PMID 7005226.
(78)^ Luo, X., et al. (2007). “CC1, a novel crenarchaeal DNA binding protein”. J. Bacteriol. 189 (2): 403–9. PMID 17085561.
(79)^ Barry, E. R., Bell, S. D. (2006). “DNA replication in the archaea”. Microbiol Mol Biol Rev 70 (4): 876–87. PMID 17158702.
(80)^ Robinson, N. P., Bell, S. D. (2005). “Origins of DNA replication in the three domains of life”. FEBS J 272 (15): 3757–66. PMID 16045748.
(81)^ abcd石野, 跡見2017、79-81頁。
(82)^ Matsunaga, F., et al. (2003). “Identification of short 'eukaryotic' Okazaki fragments synthesized from a prokaryotic replication origin”. EMBO Rep 4 (2): 154–8. PMID 12612604.
(83)^ Robinson, N.P., et al. (2004). “Identification of two origins of replication in the single chromosome of the archaeon Sulfolobus solfataricus”. CELL 161 (1): 25–38. PMID 14718164.
(84)^ abBernander, R. (1998). “Archaea and the cell cycle”. Mol. Microbiol. 29 (4): 955–61. doi:10.1046/j.1365-2958.1998.00956.x. PMID 9767564.
(85)^ 石野園子, 石野良純﹁アーキアのDNAトランスアクション--その共通性と多様性﹂﹃蛋白質核酸酵素﹄第54巻第2号、2009年、141-147頁。
(86)^ Danovaro, R., et al. (2016). “Virus-mediated archaeal hecatomb in the deep seafloor”. Sci. Adv. 2. doi:10.1126/sciadv.1600492.
(87)^ ab石野, 跡見2017、170頁。
(88)^ 古賀1998、140-141, 153-154頁。
(89)^ 石野, 跡見2017、100-101頁。
(90)^ abLecompte, O., et al. (2002). “Comparative analysis of ribosomal proteins in complete genomes: An example of reductive evolution at the domain scale”. Nucleic Acids Research 30: 5382–5390. PMID 12490706.
(91)^ Dridi, B., et al. (2011). “The antimicrobial resistance pattern of cultured human methanogens reflects the unique phylogenetic position of archaea”. J. Antimicrob. Chemother. 66 (9): 2038-44. doi:10.1093/jac/dkr251. PMID 21680581.
(92)^ abc跡見晴幸, 福居俊昭, 金井保, 今中忠行﹁アーキアのゲノムからみえるもの﹂﹃蛋白質核酸酵素﹄第54巻第2号、2009年、120–126。
(93)^ 古賀1998、176-180頁。
(94)^ 大島敏久, 櫻庭春彦﹁超好熱性アーキアのユニークな糖代謝系﹂﹃蛋白質核酸酵素﹄第54巻第2号、2009年、134–140。
(95)^ 石野, 跡見2017、132-133頁。
(96)^ 石野, 跡見2017、134-135頁。
(97)^ 古賀1998、178頁。
(98)^ 石野, 跡見2017、137頁。
(99)^ Huber, H., et al. (2008). “A dicarboxylate/4-hydroxybutyrate autotrophic carbon assimilation cycle in the hyperthermophilic Archaeum Ignicoccus hospitalis”. Proc Natl Acad Sci U S A 105 (22): 7851-6. doi:10.1073/pnas.0801043105.
(100)^ Samson, R. Y., et al. (2008). “A role for the ESCRT system in cell division in archaea”. Science 322 (5908): 1710-3. doi:10.1126/science.1165322. PMID 19008417.
(101)^ Lindås, A.C., et al. (2008). “A unique cell division machinery in the Archaea”. Proc. Natl. Acad. Sci. U S A 105 (45): 18942-6. doi:10.1073/pnas.0809467105. PMID 18987308.
(102)^ Cann, I. K. (2008). “Cell sorting protein homologs reveal an unusual diversity in archaeal cell division”. Proc Natl Acad Sci U S A 105 (45): 18653-4. doi:10.1073/pnas.0810505106. PMID 19033202.
(103)^ 帯田孝之﹁古細菌にも存在するエンドソーム選別輸送複合体(ESCRT)﹂﹃化学と生物﹄第47巻第11号、2009年、742-743頁、doi:10.1271/kagakutoseibutsu.47.742。
(104)^ 石野, 跡見2017、87頁。
(105)^ abRosenshine, I., et al. (1989). “The mechanism of DNA transfer in the mating system of an archaebacterium”. Science 245 (4924): 1387-9. PMID 2818746.
(106)^ Fröls, S., et al. (2008). “UV-inducible cellular aggregation of the hyperthermophilic archaeon Sulfolobus solfataricus is mediated by pili formation”. Mol. Microbiol. 70 (4): 938-52. doi:10.1111/j.1365-2958.2008.06459.x. PMID 18990182.
(107)^ Fröls, S., et al. (2011). “Reactions to UV damage in the model archaeon Sulfolobus solfataricus”. Biochem. Soc. Trans. 37: 36-41. doi:10.1042/BST0370036. PMID 19143598.
(108)^ abcAjon, M., et al. (2011). “UV-inducible DNA exchange in hyperthermophilic archaea mediated by type IV pili”. Mol. Microbiol. 82 (4): 807-17. doi:10.1111/j.1365-2958.2011.07861.x. PMID 21999488.
(109)^ abcEppley, J.M., et al. (2007). “Genetic exchange across a species boundary in the archaeal genus ferroplasma”. Genetics 177 (1): 407-16. doi:10.1534/genetics.107.072892. PMID 17603112.
(110)^ Francis CA, et al. (2007). “New processes and players in the nitrogen cycle: the microbial ecology of anaerobic and archaeal ammonia oxidation”. ISME J. 1 (1): 19–27. doi:10.1038/ismej.2007.8. PMID 18043610.
(111)^ Leininger, S., et al. (2006). “Archaea predominate among ammonia-oxidizing prokaryotes in soils”. Nature 442 (7104): 806–9. doi:10.1038/nature04983. PMID 16915287.
(112)^ 森本晶 他﹁硝化を担う微生物の多様性と機能﹂﹃化学と生物﹄第49巻第9号、2011年、639-644頁、doi:10.1271/kagakutoseibutsu.49.639。
(113)^ 石野, 跡見2017、36頁。
(114)^ Baker, B. J., Banfield, J. F. (2003). “Microbial communities in acid mine drainage”. FEMS Microbiology Ecology 44 (2): 139–152. doi:10.1016/S0168-6496(03)00028-X. PMID 19719632.
(115)^ “EDGAR 3.2 Fast Track 2000”. 2008年6月26日閲覧。
(116)^ Boetius, A., et al. (2000). “A marine microbial consortium apparently mediating anaerobic oxidation of methane”. Nature 407 (6804): 623-6. doi:10.1038/35036572. PMID 11034209.
(117)^ Doxey, A. C., et al. (2015). “Aquatic metagenomes implicate Thaumarchaeota in global cobalamin production”. ISME J. 9 (2): 461-71. doi:10.1038/ismej.2014.142. PMID 25126756.
(118)^ Frigaard, N.U., et al. (2006). “Proteorhodopsin lateral gene transfer between marine planktonic Bacteria and Archaea”. Nature 439 (7078): 847-50. doi:10.1038/nature04435. PMID 16482157.
(119)^ 石野, 跡見2017、38頁。
(120)^ ﹁プロテオロドプシン型のエネルギー生産量は予想外に大きい - 東大海洋研﹂マイナビニュース2012年2月17日
(121)^ Eckburg P, et al. (2003). “Archaea and their potential role in human disease”. Infect. Immun. 71 (2): 591–6. doi:10.1128/IAI.71.2.591-596.2003. PMC 145348. PMID 12540534.
(122)^ Cavicchioli, R., et al. (2003). “Pathogenic archaea: do they exist?”. Bioessays 25 (11): 1119–28. doi:10.1002/bies.10354. PMID 14579252.
(123)^ ﹁古細菌﹂感染で脳脊髄炎 認知症患者、ウイルス・細菌とは別 日本経済新聞 2018年10月7日
(124)^ Huber, H., et al. (2002). “A new phylum of Archaea represented by a nanosized hyperthermophilic symbiont”. Nature 417 (6884): 63–7. PMID 11986665.
(125)^ Chaban B, et al. (2006). “Archaeal habitats—from the extreme to the ordinary”. Can. J. Microbiol. 52 (2): 73–116. doi:10.1139/w05-147. PMID 16541146.
(126)^ Schink, B. (1997). “Energetics of syntrophic cooperation in methanogenic degradation”. Microbiol. Mol. Biol. Rev. 61 (2): 262–80. PMC 232610. PMID 9184013.
(127)^ Schopf, S., et al. (2008). “An archaeal bi-species biofilm formed by Pyrococcus furiosus and Methanopyrus kandleri”. Arch. Microbiol. 190 (3): 371-7. doi:10.1007/s00203-008-0371-9. PMID 18438643.
(128)^ Lange, M., et al. (2005). “Archaea in protozoa and metazoa”. Applied Microbiology and Biotechnology 66 (5): 465–474. doi:10.1007/s00253-004-1790-4. PMID 15630514.
(129)^ van Hoek, A. H., et al. (2000). “Multiple acquisition of methanogenic archaeal symbionts by anaerobic ciliates”. Mol. Biol. Evol. 17 (2): 251–8. PMID 10677847.
(130)^ Eckburg, P. B., et al. (2005). “Diversity of the human intestinal microbial flora”. Science 308 (5728): 1635–8. doi:10.1126/science.1110591. PMC 1395357. PMID 15831718.
(131)^ Samuel, B.S., et al. (2007). “Genomic and metabolic adaptations of Methanobrevibacter smithii to the human gut”. Proc. Natl. Acad. Sci. U S A 104 (25): 10643–8. PMID 17563350.
(132)^ Dridi, B., et al. (2012). “Age‐related prevalence of Methanomassiliicoccus luminyensis in the human gut microbiome”. APMIS 120: 10. doi:10.1111/j.1600-0463.2012.02899.x. PMID 22958284.
(133)^ Brugère, J. F., et al. (2014). “Archaebiotics: proposed therapeutic use of archaea to prevent trimethylaminuria and cardiovascular disease.”. Gut Microbes. 5 (1): 5-10. doi:10.4161/gmic.26749.
(134)^ Vianna, M. E., et al. (2006). “Identification and quantification of archaea involved in primary endodontic infections”. J. Clin. Microbiol. 44 (4): 1274–82. PMID 16597851.
(135)^ 山部こころ 他﹁メタン生成古細菌は歯周病の病原因子?﹂﹃日本歯周病学会会誌﹄第48巻第7号、2010年、463-470頁、doi:10.1271/kagakutoseibutsu.48.463。
(136)^ 前田博史﹁プラークを再考する : メタン生成古細菌と歯周病﹂﹃日本歯周病学会会誌﹄第54巻第3号、2012年、238-244頁、doi:10.2329/perio.54.238。
(137)^ Chaudhary, P.P., et al. (2018). “Methanogens in humans: potentially beneficial or harmful for health”. Appl. Microbiol. Biotechnol. 102 (7): 3095-3104. doi:10.1007/s00253-018-8871-2. PMID 29497795.
(138)^ Preston, C. M., et al. (1996). “A psychrophilic crenarchaeon inhabits a marine sponge: Cenarchaeum symbiosum gen. nov., sp. nov”. Proc. Natl. Acad. Sci. USA 93 (13): 6241–6. doi:10.1073/pnas.93.13.6241. PMC 39006. PMID 8692799.
(139)^ 李宗勲 (2009). “韓国キムチにおける乳酸菌研究の進展”. ミルクサイエンス 58 (3): 153-159. doi:10.11465/milk.58.153.
(140)^ 芝崎 勲 (2004). “食品加工と微生物 その24食塩による微生物制御”. パートナーニュース 101.
(141)^ Namwong, S., et al. (2007). “Halococcus thailandensis sp. nov., from fish sauce in Thailand.”. Int. J. Syst. Evol. Microbiol. 57: 2199-203. doi:10.1099/ijs.0.65218-0. PMID 17911282.
(142)^ 独立行政法人製品評価技術基盤機構ゲノム解析部門(NGAC) - 好熱好酸性古細菌 (Sulfolobus tokodaii strain7T (= NBRC 100140T))
(143)^ Norris, P. R., et al. (2000). “Acidophiles in bioreactor mineral processing”. Extremophiles 4 (2): 71–6. doi:10.1007/s007920050139. PMID 10805560.
(144)^ Schiraldi, C., et al. (2002). “Perspectives on biotechnological applications of archaea”. Archaea 1 (2): 75–86. PMID 15803645.
(145)^ KOD DNA polymease
(146)^ Jenney, F. E., Adams, M. W. (January 2008). “The impact of extremophiles on structural genomics (and vice versa)”. Extremophiles 12 (1): 39–50. doi:10.1007/s00792-007-0087-9. PMID 17563834.
(147)^ Schiraldi, C., et al. (2002). “Halocins and sulfolobicins: the emerging story of archaeal protein and peptide antibiotics”. J. Ind. Microbiol. Biotechnol. 28 (1): 23-31. doi:10.1038/sj/jim/7000190. PMID 11938468.
(148)^ abcIwabe, N., et al. (1989). “Evolutionary relationship of archaebacteria, eubacteria, and eukaryotes inferred from phylogenetic trees of duplicated genes”. Proc. Natl. Acad. Sci. U S A 86 (23): 9355–9. PMID 2531898.
(149)^ Wilde, S. A., et al. (1980). “Evidence from detrital zircons for the existence of continental crust and oceans on the Earth 4.4 Gyr ago”. Earth and Planetary Science Letters 47 (3): 370–382. doi:10.1016/0012-821X(80)90024-2.
(150)^ Manhesa, G., et al. (2001). “Lead isotope study of basic-ultrabasic layered complexes: Speculations about the age of the earth and primitive mantle characteristics”. Earth and Planetary Science Letters 409 (6817): 175-8. doi:10.1038/35051550. PMID 11196637.
(151)^ Dodd, M. S., et al. (2017). “Evidence for early life in Earth's oldest hydrothermal vent precipitates”. Nature 543 (7643). doi:10.1038/nature21377. PMID 28252057.
(152)^ Bell, E. A., et al. (2015). “Potentially biogenic carbon preserved in a 4.1 billion-year-old zircon”. Proc. Natl. Acad. Sci. U S A 112 (47): 14518-21. doi:10.1073/pnas.1517557112. PMID 26483481.
(153)^ Timmer, John (2012年9月4日). “3.5 billion year old organic deposits show signs of life”. Ars Technica. 2018年10月10日閲覧。
(154)^ Ueno, Y., et al. (2006). “Evidence from fluid inclusions for microbial methanogenesis in the early Archaean era”. Nature 440 (7083): 516–519. PMID 16554816.
(155)^ Feng, D. F., et al. (1997). “Determining divergence times with a protein clock: update and reevaluation”. Proc. Natl. Acad. Sci. U S A 94 (24): 13028–33. PMID 9371794.
(156)^ Wolfe, J. M., et al. (2018). “Horizontal gene transfer constrains the timing of methanogen evolution”. Nat. Ecol. Evol. 2 (5): 897-903. doi:10.1038/s41559-018-0513-7. PMID 29610466.
(157)^ abBetts, H. C., et al. (2018). “Integrated genomic and fossil evidence illuminates life's early evolution and eukaryote origin”. Nat. Ecol. Evol. 2: 1556–1562. doi:10.1038/s41559-018-0644-x. PMID 30127539.
(158)^ Theobald, D. L. (2010). “A formal test of the theory of universal common ancestry”. Nature 465 (7295): 219-22. doi:10.1038/nature09014. PMID 20463738.
(159)^ abGogarten, J.P., et al. (1989). “Evolution of the vacuolar H+-ATPase: implications for the origin of eukaryotes”. Proc. Natl. Acad. Sci. U S A 86 (27): 6661–5. PMID 2528146.
(160)^ ab斉藤2016、148-149頁。
(161)^ abcdeCavalier-Smith, T. (2002). “The neomuran origin of archaebacteria, the negibacterial root of the universal tree and bacterial megaclassification”. Cold Spring Harb Perspect Biol. 52: 7-76. doi:10.1099/00207713-52-1-7.
(162)^ Lake, J. A., et al. (1992). “Origin of the eukaryotic nucleus determined by rate-invariant analysis of rRNA sequences”. Science 257 (5066): 74-6. PMID 1621096.
(163)^ Baldauf, S. L., et al. (1996). “The root of the universal tree and the origin of eukaryotes based on elongation factor phylogeny”. Proc. Natl. Acad. Sci. U S A 93 (15): 7749-54. PMID 8755547.
(164)^ Yutin, N., et al. (2008). “The Deep Archaeal Roots of Eukaryotes”. Mol. Biol. Evol. 25 (8): 1619-1630. doi:10.1093/molbev/msn108. PMID 18463089.
(165)^ Woese, C. (1998). “The universal ancestor”. Proc. Natl. Acad. Sci. U S A 95 (12): 6854-9. PMID 9618502.
(166)^ Cox, C. J., et al. (2008). “The Deep Archaeal Roots of Eukaryotes”. Proc. Natl. Acad. Sci. U S A 105 (51): 20356–20361. doi:10.1073/pnas.0810647105. PMID 19073919.
(167)^ Williams, T. A., et al. (2013). “An archaeal origin of eukaryotes supports only two primary domains of life”. Nature 504 (7479): 231-6. doi:10.1038/nature12779. PMID 24336283.
(168)^ Katoh, K., et al. (2001). “Genetic algorithm-based maximum-likelihood analysis for molecular phylogeny”. J. Mol. Evol. 53 (4-5): 477–84. PMID 11675608.
(169)^ Battistuzzi, F. U., et al. (2004). “A genomic timescale of prokaryote evolution: insights into the origin of methanogenesis, phototrophy, and the colonization of land”. BMC. Evol. Biol. 4 (44). PMID 15535883.
(170)^ Baldauf, S. L., et al. (1996). “The root of the universal tree and the origin of eukaryotes based on elongation factor phylogeny”. Proc Natl Acad Sci U S A 93 (15): 7749-54. PMID 8755547.
(171)^ Lake, J. A., et al. (1984). “Eocytes: a new ribosome structure indicates a kingdom with a close relationship to eukaryotes”. Proc. Natl. Acad. Sci. U S A 81 (12): 3786-90. PMID 6587394.
(172)^ Lake, J. A., et al. (1988). “Origin of the eukaryotic nucleus determined by rate-invariant analysis of rRNA sequences”. Nature 331 (6152): 184-6. doi:10.1038/331184a0. PMID 3340165.
(173)^ Yamagishi, A., T. Oshima (1995). “Retern to dichotomy: Bacteria and Archaea”. Chemical evolution: Self-organization of the macromolecules of life: 155-158. ISBN 978-0937194324. PMID.
(174)^ Yamagishi, A., T. Oshima (1995). “Retern to dichotomy: Bacteria and Archaea”. Chemical evolution: Self-organization of the macromolecules of life: 156. ISBN 978-0937194324. PMID.
(175)^ Nunoura, T., et al. (2011). “Insights into the evolution of Archaea and eukaryotic protein modifier systems revealed by the genome of a novel archaeal group”. Nucleic Acids Res 39 (8): 3204-23. doi:10.1093/nar/gkq1228. PMID 21169198.
(176)^ Ettema, T.J., et al. (2011). “An actin-based cytoskeleton in archaea”. Molecular Microbiology 80 (4): 1052–61. doi:10.1111/j.1365-2958.2011.07635.x. PMID 21414041.
(177)^ Liu, Yang; Makarova, Kira S.; Huang, Wen-Cong; Wolf, Yuri I.; Nikolskaya, Anastasia N.; Zhang, Xinxu; Cai, Mingwei; Zhang, Cui-Jing et al. (2021-05-27). “Expanded diversity of Asgard archaea and their relationships with eukaryotes” (英語). Nature 593 (7860): 553–557. doi:10.1038/s41586-021-03494-3. ISSN 0028-0836.
(178)^ Brueckner, Julia; Martin, William F (2020-04-01). Pisani, Davide. ed. “Bacterial Genes Outnumber Archaeal Genes in Eukaryotic Genomes” (英語). Genome Biology and Evolution 12 (4): 282–292. doi:10.1093/gbe/evaa047. ISSN 1759-6653. PMC 7151554. PMID 32142116.
(179)^ Poole, A. M., Penny, D. (2007). “Evaluating hypotheses for the origin of eukaryotes”. Bioessays 29 (1): 74-84. PMID 17187354.
(180)^ 武村政春、﹁巨大ウイルスの細胞侵入・増殖機構﹂ ﹃ウイルス﹄ 2016年66巻2号 p.135-146, doi:10.2222/jsv.66.135
(181)^ ab渡邊洋一, 横堀伸一, 河原林裕﹁原核生物遺伝子のイントロン - 古細菌蛋白質遺伝子のイントロンの発見﹂﹃蛋白質核酸酵素﹄第47巻第7号、2002年、833–836。
(182)^ abcWoese, C. R., Fox, G. E. (1977). “Phylogenetic structure of the prokaryotic domain: the primary kingdoms”. Proc. Natl. Acad. Sci. U S A 74 (11): 5088–90. PMID 270744.
(183)^ Hori, H., Osawa, S. (1987). “Origin and evolution of organisms as deduced from 5S ribosomal RNA sequences”. Mol. Biol. Evol. 4 (5): 445–72. PMID 2452957.
(184)^ 長野2003、91頁。
(185)^ 古賀1998、29頁。
(186)^ 進化の歴史 - 細胞核の起源︵長谷川政美︶
(187)^ 张丽梅, 贺纪正.一个新的古菌类群—奇古菌门 (Thaumarchaeota). 微生物学报, 2012,54(4): 411-421.
(188)^ “Science子刊‥在深海中,病毒对古生菌进行大屠杀” (中国語). 壹讀/READ01. 2018年10月4日閲覧。
(189)^ 古賀1998、1-4頁。
(190)^ 亀倉正博﹁﹁塩と好塩菌﹂-其の1-﹂﹃極限環境微生物学会誌﹄第6巻第1号、2007年、4-10頁、doi:10.3118/jjse.6.4。
(191)^ 古賀1998、4-5頁。
(192)^ 長野2003、88-89頁。
(193)^ Brock, T.D., et al. (1969). “Thermus aquaticus gen. n. and sp. n., a nonsporulating extreme thermophile”. J. Bacteriol. 98 (1): 289-97. PMID 5781580.
(194)^ abDarland, G., et al. (1970). “A thermophilic, acidophilic mycoplasma isolated from a coal refuse pile”. Science 170 (3965): 1416-8. PMID 5481857.
(195)^ Brock, T.D., et al. (1972). “Sulfolobus: a new genus of sulfur-oxidizing bacteria living at low pH and high temperature”. Arch. Mikrobiol. 84 (1): 54-68. PMID 4559703.
(196)^ Sehgal, S.N., et al. (1962). “Lipids of Halobacterium cutirubrum”. Can J Biochem Physiol 40: 69-81. PMID 13910279.
(197)^ Langworthy, T.A., et al. (1972). “Lipids of Thermoplasma acidophilum”. J Bacteriol 112 (3): 1193-200. PMID 4344918.
(198)^ 古賀1998、6頁。
(199)^ Brock, T. D., et al. (1972). “Sulfolobus: a new genus of sulfur-oxidizing bacteria living at low pH and high temperature”. Arch. Mikrobiol. 84: 54–68. doi:10.1007/BF00408082. PMID 4559703.
(200)^ Kimura, M. (1968). “Evolutionary rate at the molecular level”. Nature 217 (5129): 624-6. PMID 5637732.
(201)^ DOI, R.H., IGARASHI, R.T. (March 1965). “Conservation of Ribosomal and Messenger Ribonucleic Acid Cistrons in Bacillus Species”. J. Bacteriol. 90: 384-90. doi:10.1016/0022-5193(65)90083-4. PMID 14329452.
(202)^ De Smedt, J., De Ley, J., (1977). “Intra- and Intergeneric Similarities of Agrobacterium Ribosomal Ribonucleic Acid Cistrons”. Int. J. Syst. Bacteria 27: 222-240.
(203)^ Takahashi, H., et al. (1969). “Conserved portion in bacterial ribosomal RNA”. J. Gen. Appl. Microbiol. 15 (2): 209-216. doi:10.2323/jgam.15.209.
(204)^ Zuckerkandl, E., Pauling, L. (March 1965). “Molecules as documents of evolutionary history”. Journal of Theoretical Biology 8 (2): 357–66. doi:10.1016/0022-5193(65)90083-4. PMID 5876245.
(205)^ 長野2003、83-84頁。
(206)^ ab長野2003、85-86頁。
(207)^ 長野2003、91-92頁。
(208)^ Makula, R.A., Singer, M. E. (1978). “Ether-containing lipids of methanogenic bacteria”. Biochem. Biophys. Res. Commun. 82 (2): 716–22. PMID 666868.
(209)^ Tornabene, T.G., et al. (1978). “Phytanyl-glycerol ethers and squalenes in the archaebacterium Methanobacterium thermoautotrophicum”. J. Mol. Evol. 11 (3): 259–66. PMID 691077.
(210)^ Woese, C. R., et al. (1978). “Archaebacteria”. J Mol Evol 11 (3): 245–51. PMID 691075.
(211)^ Mayr, E. (1998). “Two empires or three?”. Proc. Natl. Acad. Sci. U S A 95 (17): 9720-3. PMID 9707542.
(212)^ Sapp, J. (2007). “The structure of microbial evolutionary theory.”. Stud. Hist. Philos. Biol. Biomed. Sci. 38 (4): 780-95. doi:10.1016/j.shpsc.2007.09.011. PMID 18053933.
(213)^ Stetter, K. O. (1982). “Ultrathin mycelia-forming organisms from submarine volcanic areas having an optimum growth temperature of 105 °C”. Nature 300: 258 - 260. doi:10.1038/300258a0.
(214)^ abBult, C.J., et al. (1996). “Complete genome sequence of the methanogenic archaeon, Methanococcus jannaschii”. Science 273 (5278): 1058–1073. doi:10.1126/science.273.5278.1058. PMID 868808.
(215)^ No authors (2002). “Validation of publication of new names and new combinations previously effectively published outside the IJSEM. International Journal of Systematic and Evolutionary Microbiology”. Int. J. Syst. Evol. Microbiol. 52: 685-90. doi:10.1099/00207713-52-3-685. PMID 12054225.
(216)^ 石野, 跡見2017、5頁。
(217)^ Kaneko, T., et al. (1996). “Sequence analysis of the genome of the unicellular cyanobacterium Synechocystis sp. strain PCC6803. II. Sequence determination of the entire genome and assignment of potential protein-coding regions”. DNA Res. 3 (3): 109-36. PMID 8905231.
(218)^ 斉藤2007、44頁。
音声ファイル)
(五)^ 山岸明彦﹁生命の進化と古細菌﹂﹃蛋白質核酸酵素﹄第54巻第2号、2009年、108-113頁。
(六)^ 石野, 跡見2017、195頁。
(七)^ Castelle, C.J., Banfield, J.F. (2018). “Major New Microbial Groups Expand Diversity and Alter our Understanding of the Tree of Life”. Cell 172 (6): 1181-1197. doi:10.1016/j.cell.2018.02.016. PMID 29522741.
(八)^ abJames A. Lake (2015). “Eukaryotic origins”. Philos. Trans. R. Soc. Lond. B. Biol. Sci. 370 (1678). doi:10.1098/rstb.2014.0321. PMID 26323753.
(九)^ abLionel Guy, Thijs J.G. Ettema (2011). “The archaeal ‘TACK’ superphylum and the origin of eukaryotes”. Trends in Microbiology 19 (12): 580-587.
(十)^ Nunoura, T., et al. (2010). “Insights into the evolution of Archaea and eukaryotic protein modifier systems revealed by the genome of a novel archaeal group”. Nucleic Acids Research 39 (8): 3204-23. doi:10.1093/nar/gkq1228. PMID 21169198.
(11)^ Barns, S. M., et al. (1996). “Perspectives on archaeal diversity, thermophily and monophyly from environmental rRNA sequences”. Proc Natl Acad Sci U S A 93 (17): 9188–9193. PMID 8799176.
(12)^ Elkins, J. G., et al. (2008). “A korarchaeal genome reveals insights into the evolution of the Archaea”. Proc Natl Acad Sci U S A 105 (23): 8102–7. PMID 18535141.
(13)^ Evans, P. N., et al. (2015). “Methane metabolism in the archaeal phylum Bathyarchaeota revealed by genome-centric metagenomics”. Nucleic Acids Research 350 (6259): 434-8. doi:10.1126/science.aac7745. PMID 26494757.
(14)^ abMeng, J., et al. (2009). “An uncultivated crenarchaeota contains functional bacteriochlorophyll a synthase”. ISME J. 3 (1): 106-16. doi:10.1038/ismej.2008.85. PMID 18830277.
(15)^ Takai, K., Horikoshi, K. (2016). “Genetic diversity of archaea in deep-sea hydrothermal vent environments”. Genetics 152 (4): 1285-97. PMID 10430559.
(16)^ abYoussef, N. H., et al. (2015). “Insights into the metabolism, lifestyle and putative evolutionary history of the novel archaeal phylum 'Diapherotrites'”. ISME J. 9 (2): 447-60. doi:10.1038/ismej.2014.141. PMID 25083931.
(17)^ abcBaker, B. J., et al. (2010). “Enigmatic, ultrasmall, uncultivated Archaea”. Proc. Natl. Acad. Sci. U S A. 107 (19): 8806-11. doi:10.1073/pnas.0914470107. PMID 20421484.
(18)^ Comolli, L. R., et al. (2009). “Three-dimensional analysis of the structure and ecology of a novel, ultra-small archaeon”. ISME J. 3 (2): 159-67. doi:10.1038/ismej.2008.99. PMID 18946497.
(19)^ abNarasingarao, P., et al. (2012). “De novo metagenomic assembly reveals abundant novel major lineage of Archaea in hypersaline microbial communities”. ISME J. 6 (1): 81-93. doi:10.1038/ismej.2011.78. PMID 21716304.
(20)^ Waters, E., et al. (2003). “The genome of Nanoarchaeum equitans: insights into early archaeal evolution and derived parasitism”. Proc Natl Acad Sci U S A 100 (22): 12984–8. PMID 14566062.
(21)^ Todd Lowe (2009年). “Why Sequence Archaeal transcriptomes?” (英語). 米国エネルギー省共同ゲノム研究所. 2009年11月2日閲覧。
(22)^ Valentine, D. L. (2007). “Adaptations to energy stress dictate the ecology and evolution of the Archaea”. Nat. Rev. Microbiol. 5 (4): 316–23. doi:10.1038/nrmicro1619. PMID 17334387.
(23)^ Pikuta, E. V., et al. (2007). “Microbial extremophiles at the limits of life”. Crit. Rev. Microbiol. 33 (3): 183–209. doi:10.1080/10408410701451948. PMID 17653987.
(24)^ abTakai, K., et al. (2008). “Cell proliferation at 122°C and isotopically heavy CH4 production by a hyperthermophilic methanogen under high-pressure cultivation”. Proc. Natl. Acad. Sci. U S A 105: 10949-54. doi:10.1073/pnas.0712334105.
(25)^ Schleper, C., et al. (1995). “Picrophilus gen. nov., fam. nov.: a novel aerobic, heterotrophic, thermoacidophilic genus and family comprising archaea capable of growth around pH 0”. J. Bacteriol. 177: 7050–7059. PMID 8522509.
(26)^ Sorokin, D.Y., et al. (2018). “Methanonatronarchaeum thermophilum gen. nov., sp. nov. and Candidatus Methanohalarchaeum thermophilum', extremely halo(natrono)philic methyl-reducing methanogens from hypersaline lakes comprising a new euryarchaeal class Methanonatronarchaeia classis nov.”. Int. J. Syst. Evol. Microbiol. 68 (7): 2199-2208. doi:10.1099/ijsem.0.002810. PMID 29781801.
(27)^ Schleper, C., et al. (1995). “Picrophilus gen. nov., fam. nov.: a novel aerobic, heterotrophic, thermoacidophilic genus and family comprising archaea capable of growth around pH 0”. J. Bacteriol. 177: 7050–7059. PMID 8522509.
(28)^ Jolivet, E., et al. (2003). “Thermococcus gammatolerans sp. nov., a hyperthermophilic archaeon from a deep-sea hydrothermal vent that resists ionizing radiation”. Int. J. Syst. Evol. Microbiol. 53: 847-51. doi:10.1099/ijs.0.02503-0. PMID 12807211.
(29)^ ab石野, 跡見2017、26頁。
(30)^ ab石野, 跡見2017、27頁。
(31)^ Christa Schleper (2007). “Diversity of Uncultivated Archaea: Perspectives From Microbial Ecology and Metagenomics”. Archaea: Evolution, Physiology, and Molecular Biology: 39-50. doi:10.1002/9780470750865.ch4.
(32)^ Teske, A., Sørensen, K. B. (2008). “Uncultured archaea in deep marine subsurface sediments: have we caught them all?”. ISME J. 2 (1): 3–18. doi:10.1038/ismej.2007.90. PMID 18180743.
(33)^ Lipp, J. S., et al. (2008). “Significant contribution of Archaea to extant biomass in marine subsurface sediments”. Nature 454 (7207): 991. doi:10.1038/nature07174. PMID 18641632.
(34)^ López-García, P., et al. (2001). “Diversity of free-living prokaryotes from a deep-sea site at the Antarctic Polar Front”. FEMS Microbiol. Ecol. 36 (2–3): 193–202. PMID 11451524.
(35)^ DeLong, E. F., Pace, N. R. (2001). “Environmental diversity of bacteria and archaea”. Syst. Biol. 50 (4): 470–8. doi:10.1080/106351501750435040. PMID 12116647.
(36)^ DeLong, E. F. (1998). “Everything in moderation: archaea as 'non-extremophiles'”. Curr. Opin. Genet. Dev. 8 (6): 649–54. doi:10.1016/S0959-437X(98)80032-4. PMID 9914204.
(37)^ Brochier-Armanet, C., et al. (2008). “Mesophilic crenarchaeota: proposal for a third archaeal phylum, the Thaumarchaeota”. Nature Reviews Microbiology 6 (3): 245–52. doi:10.1038/nrmicro1852. PMID 18274537.
(38)^ Konneke, M., et al. (2005). “Isolation of an autotrophic ammonia-oxidizing marine archaeon”. Nature 437: 543–546. PMID 16177789.
(39)^ abStieglmeier, M., et al. (2014). “Nitrososphaera viennensis gen. nov., sp. nov., an aerobic and mesophilic, ammonia-oxidizing archaeon from soil and a member of the archaeal phylum Thaumarchaeota”. Int. J. Syst. Evol. Microbiol. 64: 2738-52. doi:10.1099/ijs.0.063172-0. PMID 24907263.
(40)^ abQin, W., et al. (2017). “Nitrosopumilus maritimus gen. nov., sp. nov., Nitrosopumilus cobalaminigenes sp. nov., Nitrosopumilus oxyclinae sp. nov., and Nitrosopumilus ureiphilus sp. nov., four marine ammonia-oxidizing archaea of the phylum Thaumarchaeota”. Int. J. Syst. Evol. Microbiol. 67 (12): 5067-5079. doi:10.1099/ijsem.0.002416. PMID 29034851.
(41)^ Jung, M.Y., et al. (2018). “Nitrosarchaeum koreense gen. nov., sp. nov., an aerobic and mesophilic, ammonia-oxidizing archaeon member of the phylum Thaumarchaeota isolated from agricultural soil”. Int. J. Syst. Evol. Microbiol.: 3084-3095. doi:10.1099/ijsem.0.002926. PMID 30124400.
(42)^ Mincer, T.J. et al. (2007). “Quantitative distribution of presumptive archaeal and bacterial nitrifiers in Monterey Bay and the North Pacific Subtropical Gyre”. Environ. Microbiol. 9 (5): 1162-75. doi:10.1111/j.1462-2920.2007.01239.x. PMID 17472632.
(43)^ 古賀1998、79頁。
(44)^ Burns, D. G., et al. (2007). “Haloquadratum walsbyi gen. nov., sp. nov., the square haloarchaeon of Walsby, isolated from saltern crystallizers in Australia and Spain”. Int J Syst Evol Microbiol 57: 387-92. doi:10.1099/ijs.0.64690-0.
(45)^ Golyshina, O. V., et al. (2000). “Ferroplasma acidiphilum gen. nov., sp. nov., an acidophilic, autotrophic, ferrous-iron-oxidizing, cell-wall-lacking, mesophilic member of the Ferroplasmaceae fam. nov., comprising a distinct lineage of the Archaea”. Int. J. Syst. Evol. Microbiol. 50 Pt 3 (3): 997–1006. PMID 10843038.
(46)^ Kuwabara, T., et al. (2005). “Thermococcus coalescens sp. nov., a cell-fusing hyperthermophilic archaeon from Suiyo Seamount”. Int. J. Syst. Evol. Microbiol. 55 (Pt 6): 2507–14. doi:10.1099/ijs.0.63432-0. PMID 16280518.
(47)^ 古賀1998、35-36頁。
(48)^ Huber H, et al. (2000). “Ignicoccus gen. nov., a novel genus of hyperthermophilic, chemolithoautotrophic Archaea, represented by two new species, Ignicoccus islandicus sp. nov. and Ignicoccus pacificus sp. nov.”. Int. J. Syst. Evol. Microbiol. 50: 2093–100. PMID 11155984.
(49)^ Küper, U., et al. (2010). “Energized outer membrane and spatial separation of metabolic processes in the hyperthermophilic Archaeon Ignicoccus hospitalis”. Proc. Natl. Acad. Sci. U S A 107 (7): 3152-6. doi:10.1073/pnas.0911711107.
(50)^ Heimerl, T., et al. (2017). “A Complex Endomembrane System in the Archaeon Ignicoccus hospitalis Tapped by Nanoarchaeum equitans”. Front. Microbiol. 8. doi:10.3389/fmicb.2017.01072.
(51)^ Nickell, S., et al. (2003). “Pyrodictium cannulae enter the periplasmic space but do not enter the cytoplasm, as revealed by cryo-electron tomography”. J. Struct. Biol. 141 (1): 34–42. doi:10.1016/S1047-8477(02)00581-6. PMID 12576018.
(52)^ Horn, C., et al. (1999). “In vivo observation of cell division of anaerobic hyperthermophiles by using a high-intensity dark-field microscope”. J. Bacteriol. 181 (16): 5114–8. PMC 94007. PMID 10438790.
(53)^ abcdeKlingl, A. (2014). “S-layer and cytoplasmic membrane – exceptions from the typical archaeal cell wall with a focus on double membranes”. Front. Microbiol. 5. doi:10.3389/fmicb.2014.00624. PMID 25505452.
(54)^ 古賀1998、262-264頁。
(55)^ 古賀1998、258-261頁。
(56)^ Kreisl, P., Kandler, O. (1986). “Chemical structure of the cell wall polymer of Methanosarcina”. System Appl. Microbiol. 7 (2-3): 293–9.
(57)^ 古賀1998、261-262, 264-265頁。
(58)^ Kinosita, Y., et al. (2016). “Direct observation of rotation and steps of the archaellum in the swimming halophilic archaeon Halobacterium salinarum”. Nat. Microbiol. 1. doi:10.1038/nmicrobiol.2016.148. PMID 27564999.
(59)^ Matzke, N. J. (2006年9月5日︵アップデータ︶). “Evolution in (brownian) space: a model for the origin of the bacterial flagellum” (英語). 2008年4月5日閲覧。
(60)^ Ng, S. Y., et al. (2006). “Archaeal flagella, bacterial flagella and type IV pili: a comparison of genes and posttranslational modifications”. J. Mol. Microbiol. Biotechnol. 11 (3-5): 167–91. PMID 16983194.
(61)^ Metlina, A. L. (2004). “Bacterial and archaeal flagella as prokaryotic motility organelles”. Biochemistry (Mosc) 69 (11): 1203–12. PMID 15627373.
(62)^ Villanueva, Laura; von Meijenfeldt, F. A. Bastiaan; Westbye, Alexander B.; Yadav, Subhash; Hopmans, Ellen C.; Dutilh, Bas E.; Damsté, Jaap S. Sinninghe (2021-01). “Bridging the membrane lipid divide: bacteria of the FCB group superphylum have the potential to synthesize archaeal ether lipids” (英語). The ISME Journal 15 (1): 168-182. doi:10.1038/s41396-020-00772-2. ISSN 1751-7370. PMC 7852524. PMID 32929208.
(63)^ Coleman, Gareth A; Pancost, Richard D; Williams, Tom A (2019-02-08). “Investigating the Origins of Membrane Phospholipid Biosynthesis Genes Using Outgroup-Free Rooting”. Genome Biology and Evolution 11 (3): 883-898. doi:10.1093/gbe/evz034. ISSN 1759-6653.
(64)^ 古賀1998、214, 265-272頁。
(65)^ Hixon, W. G., Searcy, D. G. (1993). “Cytoskeleton in the archaebacterium Thermoplasma acidophilum? Viscosity increase in soluble extracts”. Biosystems 29 (2-3): 151–60. PMID 8374067.
(66)^ “古細菌、アルケアから真核生物進化の機能的進化研究”. 論文ウォッチ. AASJ (2018年10月9日). 2018年10月9日閲覧。
(67)^ Akıl, C., et al. (2018). “Genomes of Asgard archaea encode profilins that regulate actin”. Nature. doi:10.1038/s41586-018-0548-6. PMID 30283132.
(68)^ abcdZaremba-Niedzwiedzka, K., et al. (2017). “Asgard archaea illuminate the origin of eukaryotic cellular complexity”. Nature 541 (7637): 353–358. doi:10.1038/nature21031.
(69)^ KEGG: 生命システム情報統合データベース (20). “Methanothermus fervidus” (英語/一部日本語). 2013年11月16日閲覧。
(70)^ Waters, E., et al. (2003). “The genome of Nanoarchaeum equitans: insights into early archaeal evolution and derived parasitism”. Proc. Natl. Acad. Sci. U S A 100 (22): 12984–8. PMID 14566062.
(71)^ Galagan JE, et al. (2002). “The genome of M. acetivorans reveals extensive metabolic and physiological diversity”. Genome Res 12 (4): 532–42. PMID 11932238.
(72)^ Pratas, D., Pinho, A. (2017). “On the Approximation of the Kolmogorov Complexity for DNA Sequences”. Pattern Recognition and Image Analysis 10255: 259–266. doi:10.1007/978-3-319-58838-4_29.
(73)^ Musgrave, D. R., et al. (1991). “DNA binding by the archaeal histone HMf results in positive supercoiling”. Proc Natl Acad Sci U S A 88 (23): 10397–401. PMID 1660135.
(74)^ Fahrner, R. L., et al. (2001). “An ancestral nuclear protein assembly: crystal structure of the Methanopyrus kandleri histone”. Protein Sci. 10 (10): 2002–7. PMID 11567091.
(75)^ Pereira, S.L., et al. (1997). “Archaeal nucleosomes”. Proc. Natl. Acad. Sci. USA. 94: 12633-12637. PMID 9356501.
(76)^ abcMattiroli, F., et al. (2017). “Structure of histone-based chromatin in Archaea”. Science 357: 609-612. doi:10.1126/science.aaj1849.. PMID 28798133.
(77)^ DeLange, R. J., et al. (1981). “A histone-like protein (HTa) from Thermoplasma acidophilum. II. Complete amino acid sequence”. J. Biol. Chem. 256 (2): 905–11. PMID 7005226.
(78)^ Luo, X., et al. (2007). “CC1, a novel crenarchaeal DNA binding protein”. J. Bacteriol. 189 (2): 403–9. PMID 17085561.
(79)^ Barry, E. R., Bell, S. D. (2006). “DNA replication in the archaea”. Microbiol Mol Biol Rev 70 (4): 876–87. PMID 17158702.
(80)^ Robinson, N. P., Bell, S. D. (2005). “Origins of DNA replication in the three domains of life”. FEBS J 272 (15): 3757–66. PMID 16045748.
(81)^ abcd石野, 跡見2017、79-81頁。
(82)^ Matsunaga, F., et al. (2003). “Identification of short 'eukaryotic' Okazaki fragments synthesized from a prokaryotic replication origin”. EMBO Rep 4 (2): 154–8. PMID 12612604.
(83)^ Robinson, N.P., et al. (2004). “Identification of two origins of replication in the single chromosome of the archaeon Sulfolobus solfataricus”. CELL 161 (1): 25–38. PMID 14718164.
(84)^ abBernander, R. (1998). “Archaea and the cell cycle”. Mol. Microbiol. 29 (4): 955–61. doi:10.1046/j.1365-2958.1998.00956.x. PMID 9767564.
(85)^ 石野園子, 石野良純﹁アーキアのDNAトランスアクション--その共通性と多様性﹂﹃蛋白質核酸酵素﹄第54巻第2号、2009年、141-147頁。
(86)^ Danovaro, R., et al. (2016). “Virus-mediated archaeal hecatomb in the deep seafloor”. Sci. Adv. 2. doi:10.1126/sciadv.1600492.
(87)^ ab石野, 跡見2017、170頁。
(88)^ 古賀1998、140-141, 153-154頁。
(89)^ 石野, 跡見2017、100-101頁。
(90)^ abLecompte, O., et al. (2002). “Comparative analysis of ribosomal proteins in complete genomes: An example of reductive evolution at the domain scale”. Nucleic Acids Research 30: 5382–5390. PMID 12490706.
(91)^ Dridi, B., et al. (2011). “The antimicrobial resistance pattern of cultured human methanogens reflects the unique phylogenetic position of archaea”. J. Antimicrob. Chemother. 66 (9): 2038-44. doi:10.1093/jac/dkr251. PMID 21680581.
(92)^ abc跡見晴幸, 福居俊昭, 金井保, 今中忠行﹁アーキアのゲノムからみえるもの﹂﹃蛋白質核酸酵素﹄第54巻第2号、2009年、120–126。
(93)^ 古賀1998、176-180頁。
(94)^ 大島敏久, 櫻庭春彦﹁超好熱性アーキアのユニークな糖代謝系﹂﹃蛋白質核酸酵素﹄第54巻第2号、2009年、134–140。
(95)^ 石野, 跡見2017、132-133頁。
(96)^ 石野, 跡見2017、134-135頁。
(97)^ 古賀1998、178頁。
(98)^ 石野, 跡見2017、137頁。
(99)^ Huber, H., et al. (2008). “A dicarboxylate/4-hydroxybutyrate autotrophic carbon assimilation cycle in the hyperthermophilic Archaeum Ignicoccus hospitalis”. Proc Natl Acad Sci U S A 105 (22): 7851-6. doi:10.1073/pnas.0801043105.
(100)^ Samson, R. Y., et al. (2008). “A role for the ESCRT system in cell division in archaea”. Science 322 (5908): 1710-3. doi:10.1126/science.1165322. PMID 19008417.
(101)^ Lindås, A.C., et al. (2008). “A unique cell division machinery in the Archaea”. Proc. Natl. Acad. Sci. U S A 105 (45): 18942-6. doi:10.1073/pnas.0809467105. PMID 18987308.
(102)^ Cann, I. K. (2008). “Cell sorting protein homologs reveal an unusual diversity in archaeal cell division”. Proc Natl Acad Sci U S A 105 (45): 18653-4. doi:10.1073/pnas.0810505106. PMID 19033202.
(103)^ 帯田孝之﹁古細菌にも存在するエンドソーム選別輸送複合体(ESCRT)﹂﹃化学と生物﹄第47巻第11号、2009年、742-743頁、doi:10.1271/kagakutoseibutsu.47.742。
(104)^ 石野, 跡見2017、87頁。
(105)^ abRosenshine, I., et al. (1989). “The mechanism of DNA transfer in the mating system of an archaebacterium”. Science 245 (4924): 1387-9. PMID 2818746.
(106)^ Fröls, S., et al. (2008). “UV-inducible cellular aggregation of the hyperthermophilic archaeon Sulfolobus solfataricus is mediated by pili formation”. Mol. Microbiol. 70 (4): 938-52. doi:10.1111/j.1365-2958.2008.06459.x. PMID 18990182.
(107)^ Fröls, S., et al. (2011). “Reactions to UV damage in the model archaeon Sulfolobus solfataricus”. Biochem. Soc. Trans. 37: 36-41. doi:10.1042/BST0370036. PMID 19143598.
(108)^ abcAjon, M., et al. (2011). “UV-inducible DNA exchange in hyperthermophilic archaea mediated by type IV pili”. Mol. Microbiol. 82 (4): 807-17. doi:10.1111/j.1365-2958.2011.07861.x. PMID 21999488.
(109)^ abcEppley, J.M., et al. (2007). “Genetic exchange across a species boundary in the archaeal genus ferroplasma”. Genetics 177 (1): 407-16. doi:10.1534/genetics.107.072892. PMID 17603112.
(110)^ Francis CA, et al. (2007). “New processes and players in the nitrogen cycle: the microbial ecology of anaerobic and archaeal ammonia oxidation”. ISME J. 1 (1): 19–27. doi:10.1038/ismej.2007.8. PMID 18043610.
(111)^ Leininger, S., et al. (2006). “Archaea predominate among ammonia-oxidizing prokaryotes in soils”. Nature 442 (7104): 806–9. doi:10.1038/nature04983. PMID 16915287.
(112)^ 森本晶 他﹁硝化を担う微生物の多様性と機能﹂﹃化学と生物﹄第49巻第9号、2011年、639-644頁、doi:10.1271/kagakutoseibutsu.49.639。
(113)^ 石野, 跡見2017、36頁。
(114)^ Baker, B. J., Banfield, J. F. (2003). “Microbial communities in acid mine drainage”. FEMS Microbiology Ecology 44 (2): 139–152. doi:10.1016/S0168-6496(03)00028-X. PMID 19719632.
(115)^ “EDGAR 3.2 Fast Track 2000”. 2008年6月26日閲覧。
(116)^ Boetius, A., et al. (2000). “A marine microbial consortium apparently mediating anaerobic oxidation of methane”. Nature 407 (6804): 623-6. doi:10.1038/35036572. PMID 11034209.
(117)^ Doxey, A. C., et al. (2015). “Aquatic metagenomes implicate Thaumarchaeota in global cobalamin production”. ISME J. 9 (2): 461-71. doi:10.1038/ismej.2014.142. PMID 25126756.
(118)^ Frigaard, N.U., et al. (2006). “Proteorhodopsin lateral gene transfer between marine planktonic Bacteria and Archaea”. Nature 439 (7078): 847-50. doi:10.1038/nature04435. PMID 16482157.
(119)^ 石野, 跡見2017、38頁。
(120)^ ﹁プロテオロドプシン型のエネルギー生産量は予想外に大きい - 東大海洋研﹂マイナビニュース2012年2月17日
(121)^ Eckburg P, et al. (2003). “Archaea and their potential role in human disease”. Infect. Immun. 71 (2): 591–6. doi:10.1128/IAI.71.2.591-596.2003. PMC 145348. PMID 12540534.
(122)^ Cavicchioli, R., et al. (2003). “Pathogenic archaea: do they exist?”. Bioessays 25 (11): 1119–28. doi:10.1002/bies.10354. PMID 14579252.
(123)^ ﹁古細菌﹂感染で脳脊髄炎 認知症患者、ウイルス・細菌とは別 日本経済新聞 2018年10月7日
(124)^ Huber, H., et al. (2002). “A new phylum of Archaea represented by a nanosized hyperthermophilic symbiont”. Nature 417 (6884): 63–7. PMID 11986665.
(125)^ Chaban B, et al. (2006). “Archaeal habitats—from the extreme to the ordinary”. Can. J. Microbiol. 52 (2): 73–116. doi:10.1139/w05-147. PMID 16541146.
(126)^ Schink, B. (1997). “Energetics of syntrophic cooperation in methanogenic degradation”. Microbiol. Mol. Biol. Rev. 61 (2): 262–80. PMC 232610. PMID 9184013.
(127)^ Schopf, S., et al. (2008). “An archaeal bi-species biofilm formed by Pyrococcus furiosus and Methanopyrus kandleri”. Arch. Microbiol. 190 (3): 371-7. doi:10.1007/s00203-008-0371-9. PMID 18438643.
(128)^ Lange, M., et al. (2005). “Archaea in protozoa and metazoa”. Applied Microbiology and Biotechnology 66 (5): 465–474. doi:10.1007/s00253-004-1790-4. PMID 15630514.
(129)^ van Hoek, A. H., et al. (2000). “Multiple acquisition of methanogenic archaeal symbionts by anaerobic ciliates”. Mol. Biol. Evol. 17 (2): 251–8. PMID 10677847.
(130)^ Eckburg, P. B., et al. (2005). “Diversity of the human intestinal microbial flora”. Science 308 (5728): 1635–8. doi:10.1126/science.1110591. PMC 1395357. PMID 15831718.
(131)^ Samuel, B.S., et al. (2007). “Genomic and metabolic adaptations of Methanobrevibacter smithii to the human gut”. Proc. Natl. Acad. Sci. U S A 104 (25): 10643–8. PMID 17563350.
(132)^ Dridi, B., et al. (2012). “Age‐related prevalence of Methanomassiliicoccus luminyensis in the human gut microbiome”. APMIS 120: 10. doi:10.1111/j.1600-0463.2012.02899.x. PMID 22958284.
(133)^ Brugère, J. F., et al. (2014). “Archaebiotics: proposed therapeutic use of archaea to prevent trimethylaminuria and cardiovascular disease.”. Gut Microbes. 5 (1): 5-10. doi:10.4161/gmic.26749.
(134)^ Vianna, M. E., et al. (2006). “Identification and quantification of archaea involved in primary endodontic infections”. J. Clin. Microbiol. 44 (4): 1274–82. PMID 16597851.
(135)^ 山部こころ 他﹁メタン生成古細菌は歯周病の病原因子?﹂﹃日本歯周病学会会誌﹄第48巻第7号、2010年、463-470頁、doi:10.1271/kagakutoseibutsu.48.463。
(136)^ 前田博史﹁プラークを再考する : メタン生成古細菌と歯周病﹂﹃日本歯周病学会会誌﹄第54巻第3号、2012年、238-244頁、doi:10.2329/perio.54.238。
(137)^ Chaudhary, P.P., et al. (2018). “Methanogens in humans: potentially beneficial or harmful for health”. Appl. Microbiol. Biotechnol. 102 (7): 3095-3104. doi:10.1007/s00253-018-8871-2. PMID 29497795.
(138)^ Preston, C. M., et al. (1996). “A psychrophilic crenarchaeon inhabits a marine sponge: Cenarchaeum symbiosum gen. nov., sp. nov”. Proc. Natl. Acad. Sci. USA 93 (13): 6241–6. doi:10.1073/pnas.93.13.6241. PMC 39006. PMID 8692799.
(139)^ 李宗勲 (2009). “韓国キムチにおける乳酸菌研究の進展”. ミルクサイエンス 58 (3): 153-159. doi:10.11465/milk.58.153.
(140)^ 芝崎 勲 (2004). “食品加工と微生物 その24食塩による微生物制御”. パートナーニュース 101.
(141)^ Namwong, S., et al. (2007). “Halococcus thailandensis sp. nov., from fish sauce in Thailand.”. Int. J. Syst. Evol. Microbiol. 57: 2199-203. doi:10.1099/ijs.0.65218-0. PMID 17911282.
(142)^ 独立行政法人製品評価技術基盤機構ゲノム解析部門(NGAC) - 好熱好酸性古細菌 (Sulfolobus tokodaii strain7T (= NBRC 100140T))
(143)^ Norris, P. R., et al. (2000). “Acidophiles in bioreactor mineral processing”. Extremophiles 4 (2): 71–6. doi:10.1007/s007920050139. PMID 10805560.
(144)^ Schiraldi, C., et al. (2002). “Perspectives on biotechnological applications of archaea”. Archaea 1 (2): 75–86. PMID 15803645.
(145)^ KOD DNA polymease
(146)^ Jenney, F. E., Adams, M. W. (January 2008). “The impact of extremophiles on structural genomics (and vice versa)”. Extremophiles 12 (1): 39–50. doi:10.1007/s00792-007-0087-9. PMID 17563834.
(147)^ Schiraldi, C., et al. (2002). “Halocins and sulfolobicins: the emerging story of archaeal protein and peptide antibiotics”. J. Ind. Microbiol. Biotechnol. 28 (1): 23-31. doi:10.1038/sj/jim/7000190. PMID 11938468.
(148)^ abcIwabe, N., et al. (1989). “Evolutionary relationship of archaebacteria, eubacteria, and eukaryotes inferred from phylogenetic trees of duplicated genes”. Proc. Natl. Acad. Sci. U S A 86 (23): 9355–9. PMID 2531898.
(149)^ Wilde, S. A., et al. (1980). “Evidence from detrital zircons for the existence of continental crust and oceans on the Earth 4.4 Gyr ago”. Earth and Planetary Science Letters 47 (3): 370–382. doi:10.1016/0012-821X(80)90024-2.
(150)^ Manhesa, G., et al. (2001). “Lead isotope study of basic-ultrabasic layered complexes: Speculations about the age of the earth and primitive mantle characteristics”. Earth and Planetary Science Letters 409 (6817): 175-8. doi:10.1038/35051550. PMID 11196637.
(151)^ Dodd, M. S., et al. (2017). “Evidence for early life in Earth's oldest hydrothermal vent precipitates”. Nature 543 (7643). doi:10.1038/nature21377. PMID 28252057.
(152)^ Bell, E. A., et al. (2015). “Potentially biogenic carbon preserved in a 4.1 billion-year-old zircon”. Proc. Natl. Acad. Sci. U S A 112 (47): 14518-21. doi:10.1073/pnas.1517557112. PMID 26483481.
(153)^ Timmer, John (2012年9月4日). “3.5 billion year old organic deposits show signs of life”. Ars Technica. 2018年10月10日閲覧。
(154)^ Ueno, Y., et al. (2006). “Evidence from fluid inclusions for microbial methanogenesis in the early Archaean era”. Nature 440 (7083): 516–519. PMID 16554816.
(155)^ Feng, D. F., et al. (1997). “Determining divergence times with a protein clock: update and reevaluation”. Proc. Natl. Acad. Sci. U S A 94 (24): 13028–33. PMID 9371794.
(156)^ Wolfe, J. M., et al. (2018). “Horizontal gene transfer constrains the timing of methanogen evolution”. Nat. Ecol. Evol. 2 (5): 897-903. doi:10.1038/s41559-018-0513-7. PMID 29610466.
(157)^ abBetts, H. C., et al. (2018). “Integrated genomic and fossil evidence illuminates life's early evolution and eukaryote origin”. Nat. Ecol. Evol. 2: 1556–1562. doi:10.1038/s41559-018-0644-x. PMID 30127539.
(158)^ Theobald, D. L. (2010). “A formal test of the theory of universal common ancestry”. Nature 465 (7295): 219-22. doi:10.1038/nature09014. PMID 20463738.
(159)^ abGogarten, J.P., et al. (1989). “Evolution of the vacuolar H+-ATPase: implications for the origin of eukaryotes”. Proc. Natl. Acad. Sci. U S A 86 (27): 6661–5. PMID 2528146.
(160)^ ab斉藤2016、148-149頁。
(161)^ abcdeCavalier-Smith, T. (2002). “The neomuran origin of archaebacteria, the negibacterial root of the universal tree and bacterial megaclassification”. Cold Spring Harb Perspect Biol. 52: 7-76. doi:10.1099/00207713-52-1-7.
(162)^ Lake, J. A., et al. (1992). “Origin of the eukaryotic nucleus determined by rate-invariant analysis of rRNA sequences”. Science 257 (5066): 74-6. PMID 1621096.
(163)^ Baldauf, S. L., et al. (1996). “The root of the universal tree and the origin of eukaryotes based on elongation factor phylogeny”. Proc. Natl. Acad. Sci. U S A 93 (15): 7749-54. PMID 8755547.
(164)^ Yutin, N., et al. (2008). “The Deep Archaeal Roots of Eukaryotes”. Mol. Biol. Evol. 25 (8): 1619-1630. doi:10.1093/molbev/msn108. PMID 18463089.
(165)^ Woese, C. (1998). “The universal ancestor”. Proc. Natl. Acad. Sci. U S A 95 (12): 6854-9. PMID 9618502.
(166)^ Cox, C. J., et al. (2008). “The Deep Archaeal Roots of Eukaryotes”. Proc. Natl. Acad. Sci. U S A 105 (51): 20356–20361. doi:10.1073/pnas.0810647105. PMID 19073919.
(167)^ Williams, T. A., et al. (2013). “An archaeal origin of eukaryotes supports only two primary domains of life”. Nature 504 (7479): 231-6. doi:10.1038/nature12779. PMID 24336283.
(168)^ Katoh, K., et al. (2001). “Genetic algorithm-based maximum-likelihood analysis for molecular phylogeny”. J. Mol. Evol. 53 (4-5): 477–84. PMID 11675608.
(169)^ Battistuzzi, F. U., et al. (2004). “A genomic timescale of prokaryote evolution: insights into the origin of methanogenesis, phototrophy, and the colonization of land”. BMC. Evol. Biol. 4 (44). PMID 15535883.
(170)^ Baldauf, S. L., et al. (1996). “The root of the universal tree and the origin of eukaryotes based on elongation factor phylogeny”. Proc Natl Acad Sci U S A 93 (15): 7749-54. PMID 8755547.
(171)^ Lake, J. A., et al. (1984). “Eocytes: a new ribosome structure indicates a kingdom with a close relationship to eukaryotes”. Proc. Natl. Acad. Sci. U S A 81 (12): 3786-90. PMID 6587394.
(172)^ Lake, J. A., et al. (1988). “Origin of the eukaryotic nucleus determined by rate-invariant analysis of rRNA sequences”. Nature 331 (6152): 184-6. doi:10.1038/331184a0. PMID 3340165.
(173)^ Yamagishi, A., T. Oshima (1995). “Retern to dichotomy: Bacteria and Archaea”. Chemical evolution: Self-organization of the macromolecules of life: 155-158. ISBN 978-0937194324. PMID.
(174)^ Yamagishi, A., T. Oshima (1995). “Retern to dichotomy: Bacteria and Archaea”. Chemical evolution: Self-organization of the macromolecules of life: 156. ISBN 978-0937194324. PMID.
(175)^ Nunoura, T., et al. (2011). “Insights into the evolution of Archaea and eukaryotic protein modifier systems revealed by the genome of a novel archaeal group”. Nucleic Acids Res 39 (8): 3204-23. doi:10.1093/nar/gkq1228. PMID 21169198.
(176)^ Ettema, T.J., et al. (2011). “An actin-based cytoskeleton in archaea”. Molecular Microbiology 80 (4): 1052–61. doi:10.1111/j.1365-2958.2011.07635.x. PMID 21414041.
(177)^ Liu, Yang; Makarova, Kira S.; Huang, Wen-Cong; Wolf, Yuri I.; Nikolskaya, Anastasia N.; Zhang, Xinxu; Cai, Mingwei; Zhang, Cui-Jing et al. (2021-05-27). “Expanded diversity of Asgard archaea and their relationships with eukaryotes” (英語). Nature 593 (7860): 553–557. doi:10.1038/s41586-021-03494-3. ISSN 0028-0836.
(178)^ Brueckner, Julia; Martin, William F (2020-04-01). Pisani, Davide. ed. “Bacterial Genes Outnumber Archaeal Genes in Eukaryotic Genomes” (英語). Genome Biology and Evolution 12 (4): 282–292. doi:10.1093/gbe/evaa047. ISSN 1759-6653. PMC 7151554. PMID 32142116.
(179)^ Poole, A. M., Penny, D. (2007). “Evaluating hypotheses for the origin of eukaryotes”. Bioessays 29 (1): 74-84. PMID 17187354.
(180)^ 武村政春、﹁巨大ウイルスの細胞侵入・増殖機構﹂ ﹃ウイルス﹄ 2016年66巻2号 p.135-146, doi:10.2222/jsv.66.135
(181)^ ab渡邊洋一, 横堀伸一, 河原林裕﹁原核生物遺伝子のイントロン - 古細菌蛋白質遺伝子のイントロンの発見﹂﹃蛋白質核酸酵素﹄第47巻第7号、2002年、833–836。
(182)^ abcWoese, C. R., Fox, G. E. (1977). “Phylogenetic structure of the prokaryotic domain: the primary kingdoms”. Proc. Natl. Acad. Sci. U S A 74 (11): 5088–90. PMID 270744.
(183)^ Hori, H., Osawa, S. (1987). “Origin and evolution of organisms as deduced from 5S ribosomal RNA sequences”. Mol. Biol. Evol. 4 (5): 445–72. PMID 2452957.
(184)^ 長野2003、91頁。
(185)^ 古賀1998、29頁。
(186)^ 進化の歴史 - 細胞核の起源︵長谷川政美︶
(187)^ 张丽梅, 贺纪正.一个新的古菌类群—奇古菌门 (Thaumarchaeota). 微生物学报, 2012,54(4): 411-421.
(188)^ “Science子刊‥在深海中,病毒对古生菌进行大屠杀” (中国語). 壹讀/READ01. 2018年10月4日閲覧。
(189)^ 古賀1998、1-4頁。
(190)^ 亀倉正博﹁﹁塩と好塩菌﹂-其の1-﹂﹃極限環境微生物学会誌﹄第6巻第1号、2007年、4-10頁、doi:10.3118/jjse.6.4。
(191)^ 古賀1998、4-5頁。
(192)^ 長野2003、88-89頁。
(193)^ Brock, T.D., et al. (1969). “Thermus aquaticus gen. n. and sp. n., a nonsporulating extreme thermophile”. J. Bacteriol. 98 (1): 289-97. PMID 5781580.
(194)^ abDarland, G., et al. (1970). “A thermophilic, acidophilic mycoplasma isolated from a coal refuse pile”. Science 170 (3965): 1416-8. PMID 5481857.
(195)^ Brock, T.D., et al. (1972). “Sulfolobus: a new genus of sulfur-oxidizing bacteria living at low pH and high temperature”. Arch. Mikrobiol. 84 (1): 54-68. PMID 4559703.
(196)^ Sehgal, S.N., et al. (1962). “Lipids of Halobacterium cutirubrum”. Can J Biochem Physiol 40: 69-81. PMID 13910279.
(197)^ Langworthy, T.A., et al. (1972). “Lipids of Thermoplasma acidophilum”. J Bacteriol 112 (3): 1193-200. PMID 4344918.
(198)^ 古賀1998、6頁。
(199)^ Brock, T. D., et al. (1972). “Sulfolobus: a new genus of sulfur-oxidizing bacteria living at low pH and high temperature”. Arch. Mikrobiol. 84: 54–68. doi:10.1007/BF00408082. PMID 4559703.
(200)^ Kimura, M. (1968). “Evolutionary rate at the molecular level”. Nature 217 (5129): 624-6. PMID 5637732.
(201)^ DOI, R.H., IGARASHI, R.T. (March 1965). “Conservation of Ribosomal and Messenger Ribonucleic Acid Cistrons in Bacillus Species”. J. Bacteriol. 90: 384-90. doi:10.1016/0022-5193(65)90083-4. PMID 14329452.
(202)^ De Smedt, J., De Ley, J., (1977). “Intra- and Intergeneric Similarities of Agrobacterium Ribosomal Ribonucleic Acid Cistrons”. Int. J. Syst. Bacteria 27: 222-240.
(203)^ Takahashi, H., et al. (1969). “Conserved portion in bacterial ribosomal RNA”. J. Gen. Appl. Microbiol. 15 (2): 209-216. doi:10.2323/jgam.15.209.
(204)^ Zuckerkandl, E., Pauling, L. (March 1965). “Molecules as documents of evolutionary history”. Journal of Theoretical Biology 8 (2): 357–66. doi:10.1016/0022-5193(65)90083-4. PMID 5876245.
(205)^ 長野2003、83-84頁。
(206)^ ab長野2003、85-86頁。
(207)^ 長野2003、91-92頁。
(208)^ Makula, R.A., Singer, M. E. (1978). “Ether-containing lipids of methanogenic bacteria”. Biochem. Biophys. Res. Commun. 82 (2): 716–22. PMID 666868.
(209)^ Tornabene, T.G., et al. (1978). “Phytanyl-glycerol ethers and squalenes in the archaebacterium Methanobacterium thermoautotrophicum”. J. Mol. Evol. 11 (3): 259–66. PMID 691077.
(210)^ Woese, C. R., et al. (1978). “Archaebacteria”. J Mol Evol 11 (3): 245–51. PMID 691075.
(211)^ Mayr, E. (1998). “Two empires or three?”. Proc. Natl. Acad. Sci. U S A 95 (17): 9720-3. PMID 9707542.
(212)^ Sapp, J. (2007). “The structure of microbial evolutionary theory.”. Stud. Hist. Philos. Biol. Biomed. Sci. 38 (4): 780-95. doi:10.1016/j.shpsc.2007.09.011. PMID 18053933.
(213)^ Stetter, K. O. (1982). “Ultrathin mycelia-forming organisms from submarine volcanic areas having an optimum growth temperature of 105 °C”. Nature 300: 258 - 260. doi:10.1038/300258a0.
(214)^ abBult, C.J., et al. (1996). “Complete genome sequence of the methanogenic archaeon, Methanococcus jannaschii”. Science 273 (5278): 1058–1073. doi:10.1126/science.273.5278.1058. PMID 868808.
(215)^ No authors (2002). “Validation of publication of new names and new combinations previously effectively published outside the IJSEM. International Journal of Systematic and Evolutionary Microbiology”. Int. J. Syst. Evol. Microbiol. 52: 685-90. doi:10.1099/00207713-52-3-685. PMID 12054225.
(216)^ 石野, 跡見2017、5頁。
(217)^ Kaneko, T., et al. (1996). “Sequence analysis of the genome of the unicellular cyanobacterium Synechocystis sp. strain PCC6803. II. Sequence determination of the entire genome and assignment of potential protein-coding regions”. DNA Res. 3 (3): 109-36. PMID 8905231.
(218)^ 斉藤2007、44頁。
参考文献[編集]
●古賀洋介, 亀倉正博︵編︶﹃古細菌の生物学﹄東京大学出版会、1998年。ISBN 4130602039。 ●石野良純粋, 跡見晴幸︵編︶﹃アーキア生物学﹄共立出版、2017年。ISBN 9784320057852。 ●David W. Wolf (著), 長野敬 (訳), 赤松真紀 (訳)﹃地中生命の驚異︵第3版︶﹄青土社、2003年。ISBN 978-4791760411。 ●Andrew H. Knoll (著), 斉藤隆央(訳)﹃生命 最初の30億年︵第6版︶﹄紀伊國屋書店、2007年。ISBN 978-4314009881。 ●Nick Lane(著), 斉藤隆央(訳)﹃生命、エネルギー、進化﹄みすず書房、2016年。ISBN 978-4-622-08534-8。 ●Boone, D. R., Castenholz, R. W. & Garrity, G. M. (2001). Bergey's Manual of Systematic Bacteriology, 2nd Edition, Volume One,The Archaea and the Deeply Branching and Phototrophic Bacteria. Springer-Verlag. ISBN 0387987711 ●井上勲﹃藻類30億年の自然史︵第2版︶ - 藻類から見る生物進化・地球・環境﹄東海大学出版会、2007年。ISBN 978-4-486-01777-6。 ●黒岩常祥﹃ミトコンドリアはどこからきたか 生命40億年を遡る﹄NHKブックス、2000年。ISBN 9784140018873。 ●川上紳一﹃生命と地球の共進化﹄NHKブックス、2000年。ISBN 978-4-14-001888-0。 ●Nick Lane(著), 斉藤隆央(訳)﹃ミトコンドリアが進化を決めた﹄みすず書房、2007年。ISBN 9784622073406。 ●Richard A. Fortey(著), 渡辺政隆(訳)﹃生命40億年全史﹄草思社、2003年。ISBN 9784794211897。 ●中沢弘基﹃生命の起源・地球が書いたシナリオ﹄新日本出版社、2006年。ISBN 9784406032551。参考・関連ウェブサイト[編集]
●Euzéby, J. P.. “List of Prokaryotic names with Standing in Nomenclature” (英語). 2018年10月11日閲覧。 ●Kanehisa Laboratories. “KEGG” (英語・日本語). 2018年10月11日閲覧。 ●“International Journal of Systematic and Evolutionary Microbiology” (英語). Microbiology Society. 2018年10月11日閲覧。 ●“NCBI taxonomy page on Archaea” (英語). 米国立生物工学情報センター. 2018年11月5日閲覧。 ●“GTDB - taxonomy tree” (英語). ゲノム分類データベース. 2018年11月5日閲覧。関連項目[編集]
外部リンク[編集]

















